[English] 日本語
 Yorodumi
Yorodumi- PDB-5lyy: Fragment-based inhibitors of Lipoprotein associated Phospholipase A2 -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5lyy | ||||||
|---|---|---|---|---|---|---|---|
| Title | Fragment-based inhibitors of Lipoprotein associated Phospholipase A2 | ||||||
 Components Components | Platelet-activating factor acetylhydrolase | ||||||
 Keywords Keywords | HYDROLASE / Lp-PLA2 phospholipase / lipid metabolism / inhibitors / Lp-PLA2#4 | ||||||
| Function / homology |  Function and homology information Function and homology informationplasma lipoprotein particle oxidation / platelet activating factor catabolic process / 1-alkyl-2-acetylglycerophosphocholine esterase / : / 1-alkyl-2-acetylglycerophosphocholine esterase activity / platelet activating factor metabolic process / lipid oxidation / low-density lipoprotein particle / high-density lipoprotein particle / low-density lipoprotein particle remodeling ...plasma lipoprotein particle oxidation / platelet activating factor catabolic process / 1-alkyl-2-acetylglycerophosphocholine esterase / : / 1-alkyl-2-acetylglycerophosphocholine esterase activity / platelet activating factor metabolic process / lipid oxidation / low-density lipoprotein particle / high-density lipoprotein particle / low-density lipoprotein particle remodeling / positive regulation of monocyte chemotaxis / phosphatidylcholine catabolic process / peptide hormone processing / hydrolase activity, acting on ester bonds / Synthesis, secretion, and deacylation of Ghrelin / phospholipid binding / positive regulation of inflammatory response / extracellular region Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  FOURIER SYNTHESIS / Resolution: 2.17 Å FOURIER SYNTHESIS / Resolution: 2.17 Å | ||||||
 Authors Authors | Woolford, A. / Day, P. | ||||||
 Citation Citation |  Journal: J. Med. Chem. / Year: 2016 Journal: J. Med. Chem. / Year: 2016Title: Fragment-Based Approach to the Development of an Orally Bioavailable Lactam Inhibitor of Lipoprotein-Associated Phospholipase A2 (Lp-PLA2). Authors: Woolford, A.J. / Day, P.J. / Beneton, V. / Berdini, V. / Coyle, J.E. / Dudit, Y. / Grondin, P. / Huet, P. / Lee, L.Y. / Manas, E.S. / McMenamin, R.L. / Murray, C.W. / Page, L.W. / Patel, V.K. ...Authors: Woolford, A.J. / Day, P.J. / Beneton, V. / Berdini, V. / Coyle, J.E. / Dudit, Y. / Grondin, P. / Huet, P. / Lee, L.Y. / Manas, E.S. / McMenamin, R.L. / Murray, C.W. / Page, L.W. / Patel, V.K. / Potvain, F. / Rich, S.J. / Sang, Y. / Somers, D.O. / Trottet, L. / Wan, Z. / Zhang, X. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5lyy.cif.gz 5lyy.cif.gz | 170.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5lyy.ent.gz pdb5lyy.ent.gz | 133.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5lyy.json.gz 5lyy.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  5lyy_validation.pdf.gz 5lyy_validation.pdf.gz | 436.2 KB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  5lyy_full_validation.pdf.gz 5lyy_full_validation.pdf.gz | 437.6 KB | Display | |
| Data in XML |  5lyy_validation.xml.gz 5lyy_validation.xml.gz | 17.1 KB | Display | |
| Data in CIF |  5lyy_validation.cif.gz 5lyy_validation.cif.gz | 24.9 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ly/5lyy https://data.pdbj.org/pub/pdb/validation_reports/ly/5lyy ftp://data.pdbj.org/pub/pdb/validation_reports/ly/5lyy ftp://data.pdbj.org/pub/pdb/validation_reports/ly/5lyy | HTTPS FTP |
-Related structure data
| Related structure data |  5lz2C  5lz4C  5lz5C  5lz7C  5lz8C  5lz9C C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 44203.129 Da / Num. of mol.: 1 Mutation: Deletion 1-45, Deletion 430- 442, I429H, insertion HHHH at C-terminal Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: PLA2G7, PAFAH / Production host: Homo sapiens (human) / Gene: PLA2G7, PAFAH / Production host:  References: UniProt: Q13093, 1-alkyl-2-acetylglycerophosphocholine esterase |
|---|---|
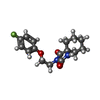
| #2: Chemical | ChemComp-7BJ / |
| #3: Water | ChemComp-HOH / |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.45 Å3/Da / Density % sol: 49.88 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, sitting drop / pH: 7.4 Details: 28.0%w/v PEG 3350, 0.1M HEPES/NaOHpH=7.4, 1.3M NaCl |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  ROTATING ANODE / Type: RIGAKU FR-E SUPERBRIGHT / Wavelength: 1.5418 Å ROTATING ANODE / Type: RIGAKU FR-E SUPERBRIGHT / Wavelength: 1.5418 Å |
| Detector | Type: RIGAKU RAXIS II / Detector: IMAGE PLATE / Date: Sep 17, 2010 / Details: mirrors |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.5418 Å / Relative weight: 1 |
| Reflection | Resolution: 2.17→34.38 Å / Num. obs: 22495 / % possible obs: 98.2 % / Redundancy: 2.3 % / Rmerge(I) obs: 0.062 / Net I/σ(I): 10.3 |
| Reflection shell | Resolution: 2.17→2.31 Å / Rmerge(I) obs: 0.459 / Mean I/σ(I) obs: 1.9 / % possible all: 99.7 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  FOURIER SYNTHESIS / Resolution: 2.17→34.38 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.935 / SU B: 12.634 / SU ML: 0.164 / Cross valid method: THROUGHOUT / ESU R: 0.252 / ESU R Free: 0.197 / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS FOURIER SYNTHESIS / Resolution: 2.17→34.38 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.935 / SU B: 12.634 / SU ML: 0.164 / Cross valid method: THROUGHOUT / ESU R: 0.252 / ESU R Free: 0.197 / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 40.016 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: 1 / Resolution: 2.17→34.38 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj