| 登録情報 | データベース: PDB / ID: 5isc
|
|---|
| タイトル | Crystal structure of mouse CARM1 in complex with inhibitor SA0491 |
|---|
 要素 要素 | Histone-arginine methyltransferase CARM1 |
|---|
 キーワード キーワード | TRANSFERASE / PROTEIN ARGININE METHYLTRANSFERASE / CATALYTIC DOMAIN / CHROMATIN REGULATOR / MRNA PROCESSING / MRNA SPLICING / NUCLEUS / S-ADENOSYL-L-METHIONINE / TRANSCRIPTION / TRANSCRIPTION REGULATION |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of growth plate cartilage chondrocyte proliferation / histone H3R26 methyltransferase activity / histone H3R17 methyltransferase activity / endochondral bone morphogenesis / histone H3R2 methyltransferase activity / RMTs methylate histone arginines / Regulation of lipid metabolism by PPARalpha / Cytoprotection by HMOX1 / protein-arginine omega-N asymmetric methyltransferase activity / type I protein arginine methyltransferase ...regulation of growth plate cartilage chondrocyte proliferation / histone H3R26 methyltransferase activity / histone H3R17 methyltransferase activity / endochondral bone morphogenesis / histone H3R2 methyltransferase activity / RMTs methylate histone arginines / Regulation of lipid metabolism by PPARalpha / Cytoprotection by HMOX1 / protein-arginine omega-N asymmetric methyltransferase activity / type I protein arginine methyltransferase / Estrogen-dependent gene expression / protein methyltransferase activity / regulation of intracellular estrogen receptor signaling pathway / replication fork reversal / protein-arginine N-methyltransferase activity / histone methyltransferase activity / nuclear replication fork / nuclear receptor-mediated steroid hormone signaling pathway / positive regulation of fat cell differentiation / estrogen receptor signaling pathway / protein localization to chromatin / RNA polymerase II transcription regulator complex / methylation / transcription coactivator activity / cell population proliferation / transcription cis-regulatory region binding / positive regulation of cell population proliferation / regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / protein-containing complex / nucleus / cytosol類似検索 - 分子機能 Histone-arginine methyltransferase CARM1, N-terminal / Coactivator-associated arginine methyltransferase 1 N terminal / Ribosomal protein L11 methyltransferase (PrmA) / Hnrnp arginine n-methyltransferase1 / Hnrnp arginine n-methyltransferase1 / : / Arginine methyltransferase oligomerization subdomain / Protein arginine N-methyltransferase / SAM-dependent methyltransferase PRMT-type domain profile. / Vaccinia Virus protein VP39 ...Histone-arginine methyltransferase CARM1, N-terminal / Coactivator-associated arginine methyltransferase 1 N terminal / Ribosomal protein L11 methyltransferase (PrmA) / Hnrnp arginine n-methyltransferase1 / Hnrnp arginine n-methyltransferase1 / : / Arginine methyltransferase oligomerization subdomain / Protein arginine N-methyltransferase / SAM-dependent methyltransferase PRMT-type domain profile. / Vaccinia Virus protein VP39 / Distorted Sandwich / PH-like domain superfamily / S-adenosyl-L-methionine-dependent methyltransferase superfamily / Rossmann fold / 3-Layer(aba) Sandwich / Mainly Beta / Alpha Beta類似検索 - ドメイン・相同性 Chem-6D0 / 1,2-DIMETHOXYETHANE / 1-METHOXY-2-(2-METHOXYETHOXY)ETHANE / Chem-SAO / Histone-arginine methyltransferase CARM1類似検索 - 構成要素 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.6 Å 分子置換 / 解像度: 2.6 Å |
|---|
 データ登録者 データ登録者 | Cura, V. / Marechal, N. / Mailliot, J. / Troffer-Charlier, N. / Hassenboehler, P. / Wurtz, J.M. / Bonnefond, L. / Cavarelli, J. |
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Crystal structure of mouse CARM1 in complex with inhibitor SA0491
著者: Cura, V. / Marechal, N. / Mailliot, J. / Troffer-Charlier, N. / Wurtz, J.M. / Bonnefond, L. / Cavarelli, J. |
|---|
| 履歴 | | 登録 | 2016年3月15日 | 登録サイト: RCSB / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年3月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年1月10日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 2.0 | 2025年12月10日 | Group: Atomic model / Data collection / Structure summary / カテゴリ: atom_site / chem_comp / pdbx_entry_details
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_atom_id / _atom_site.label_atom_id / _chem_comp.type |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.6 Å
分子置換 / 解像度: 2.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5isc.cif.gz
5isc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5isc.ent.gz
pdb5isc.ent.gz PDB形式
PDB形式 5isc.json.gz
5isc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5isc_validation.pdf.gz
5isc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5isc_full_validation.pdf.gz
5isc_full_validation.pdf.gz 5isc_validation.xml.gz
5isc_validation.xml.gz 5isc_validation.cif.gz
5isc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/is/5isc
https://data.pdbj.org/pub/pdb/validation_reports/is/5isc ftp://data.pdbj.org/pub/pdb/validation_reports/is/5isc
ftp://data.pdbj.org/pub/pdb/validation_reports/is/5isc
 リンク
リンク 集合体
集合体
 要素
要素

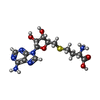

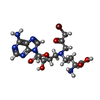

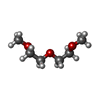






 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SOLEIL
SOLEIL  / ビームライン: PROXIMA 1 / 波長: 0.979 Å
/ ビームライン: PROXIMA 1 / 波長: 0.979 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







