+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5673 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of enterovirus 71 complexed with a Fab fragment | |||||||||
 マップデータ マップデータ | Asymmetric reconstruction of ev71-Fab complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | EV71 / enterovirus / HFMD / neutralizing antibody / Fab / cryoEM / strain-specific eptitope / VP1-145 | |||||||||
| 生物種 |    Human enterovirus 71 (エンテロウイルス) Human enterovirus 71 (エンテロウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 23.4 Å | |||||||||
 データ登録者 データ登録者 | Lee H / Cifuente JO / Ashley RE / Conway JF / Makhov AM / Tano Y / Shimizu H / Nishimura Y / Hafenstein S | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2013 ジャーナル: J Virol / 年: 2013タイトル: A strain-specific epitope of enterovirus 71 identified by cryo-electron microscopy of the complex with fab from neutralizing antibody. 著者: Hyunwook Lee / Javier O Cifuente / Robert E Ashley / James F Conway / Alexander M Makhov / Yoshio Tano / Hiroyuki Shimizu / Yorihiro Nishimura / Susan Hafenstein /  要旨: Enterovirus 71 (EV71) is a picornavirus that causes outbreaks of hand, foot, and mouth disease (HFMD), primarily in the Asia-Pacific area. Unlike coxsackievirus A16, which also causes HFMD, EV71 ...Enterovirus 71 (EV71) is a picornavirus that causes outbreaks of hand, foot, and mouth disease (HFMD), primarily in the Asia-Pacific area. Unlike coxsackievirus A16, which also causes HFMD, EV71 induces severe neuropathology leading to high fatalities, especially among children under the age of 6 years. Currently, no established vaccines or treatments are available against EV71 infection. The monoclonal antibody MA28-7 neutralizes only specific strains of EV71 that have a conserved glycine at amino acid VP1-145, a surface-exposed residue that maps to the 5-fold vertex and that has been implicated in receptor binding. The cryo-electron microscopy structure of a complex between EV71 and the Fab fragment of MA28-7 shows that only one Fab fragment occupies each 5-fold vertex. A positively charged patch, which has also been implicated in receptor binding, lies within the Fab footprint. We identify the strain-specific epitope of EV71 and discuss the possible neutralization mechanisms of the antibody. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5673.map.gz emd_5673.map.gz | 78.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5673-v30.xml emd-5673-v30.xml emd-5673.xml emd-5673.xml | 12.2 KB 12.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5673_1.jpg emd_5673_1.jpg | 96.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5673 http://ftp.pdbj.org/pub/emdb/structures/EMD-5673 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5673 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5673 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5673.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5673.map.gz / 形式: CCP4 / 大きさ: 412 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetric reconstruction of ev71-Fab complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.23 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
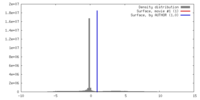
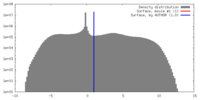
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Enterovirus 71 complexed with Fab Fragment of MA28-7 neutralizing...
| 全体 | 名称: Enterovirus 71 complexed with Fab Fragment of MA28-7 neutralizing antibody |
|---|---|
| 要素 |
|
-超分子 #1000: Enterovirus 71 complexed with Fab Fragment of MA28-7 neutralizing...
| 超分子 | 名称: Enterovirus 71 complexed with Fab Fragment of MA28-7 neutralizing antibody タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 8.05 MDa |
-超分子 #1: Human enterovirus 71
| 超分子 | 名称: Human enterovirus 71 / タイプ: virus / ID: 1 / Name.synonym: EV71 / 詳細: Fab fragment is bound on the virus surface / NCBI-ID: 39054 / 生物種: Human enterovirus 71 / Sci species strain: 1095/Shiga / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: EV71 |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| 分子量 | 理論値: 8 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: VP1-4 / 直径: 300 Å / T番号(三角分割数): 1 |
-分子 #1: MA28-7 neutralizing antibody Fab fragment
| 分子 | 名称: MA28-7 neutralizing antibody Fab fragment / タイプ: protein_or_peptide / ID: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10 mM Tris, 200 mM NaCl, 50 mM MgCl2 |
| グリッド | 詳細: glow-discharged holey carbon Quantifoil electron microscopy grids |
| 凍結 | 凍結剤: ETHANE-PROPANE MIXTURE / チャンバー内湿度: 95 % / チャンバー内温度: 76 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 平均: 95 K |
| 日付 | 2011年2月2日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 実像数: 45 / 平均電子線量: 15 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 48425 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 6.12 µm / 最小 デフォーカス(公称値): 1.9 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Selected particles were low-pass filtered at 10 Angstrom. Asymmetric reconstruction was performed with breaksym option in EMAN2. |
|---|---|
| CTF補正 | 詳細: Each image |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 23.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN2 詳細: The breaksym option in EMAN2 was used to search for the icosahedrally-related orientations. 使用した粒子像数: 27026 |
| 最終 2次元分類 | クラス数: 2300 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: H / Chain - #1 - Chain ID: L |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The Fab structure was fitted manually, then refined in Chimera. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
| 得られたモデル |  PDB-3j3z: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)