| 登録情報 | データベース: PDB / ID: 4x1i
|
|---|
| タイトル | Discovery of cytotoxic Dolastatin 10 analogs with N-terminal modifications |
|---|
 要素 要素 | - Stathmin-4
- Tubulin alpha chain
- Tubulin beta chain
|
|---|
 キーワード キーワード | STRUCTURAL PROTEIN/INHIBITOR / Binding Sites / Competitive / Cattle / Tumor / Colchicine / Humans / Microtubules / Protein Binding / Protein Conformation / Protein Multimerization / Tubulin / Tubulin Modulators / STRUCTURAL PROTEIN-INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
axonemal microtubule / organelle transport along microtubule / glial cell differentiation / forebrain morphogenesis / neuron projection arborization / cerebellar cortex morphogenesis / dentate gyrus development / pyramidal neuron differentiation / centrosome cycle / motor behavior ...axonemal microtubule / organelle transport along microtubule / glial cell differentiation / forebrain morphogenesis / neuron projection arborization / cerebellar cortex morphogenesis / dentate gyrus development / pyramidal neuron differentiation / centrosome cycle / motor behavior / microtubule depolymerization / response to L-glutamate / smoothened signaling pathway / regulation of synapse organization / startle response / locomotory exploration behavior / microtubule polymerization / regulation of microtubule polymerization or depolymerization / response to tumor necrosis factor / response to mechanical stimulus / condensed chromosome / homeostasis of number of cells within a tissue / cellular response to calcium ion / tubulin binding / adult locomotory behavior / intracellular protein transport / synapse organization / neuron migration / visual learning / neuromuscular junction / recycling endosome / structural constituent of cytoskeleton / memory / cerebral cortex development / microtubule cytoskeleton organization / cytoplasmic ribonucleoprotein granule / neuron projection development / mitotic cell cycle / gene expression / growth cone / neuron apoptotic process / microtubule / hydrolase activity / neuron projection / protein heterodimerization activity / GTPase activity / protein-containing complex binding / GTP binding / Golgi apparatus / identical protein binding / metal ion binding / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 Methane Monooxygenase Hydroxylase; Chain G, domain 1 - #30 / Methane Monooxygenase Hydroxylase; Chain G, domain 1 / Helix hairpin bin / Tubulin/FtsZ, C-terminal domain / Tubulin/FtsZ, GTPase domain / Stathmin family / Stathmin, conserved site / Stathmin superfamily / Stathmin family / Stathmin family signature 1. ...Methane Monooxygenase Hydroxylase; Chain G, domain 1 - #30 / Methane Monooxygenase Hydroxylase; Chain G, domain 1 / Helix hairpin bin / Tubulin/FtsZ, C-terminal domain / Tubulin/FtsZ, GTPase domain / Stathmin family / Stathmin, conserved site / Stathmin superfamily / Stathmin family / Stathmin family signature 1. / Stathmin family signature 2. / Stathmin-like (SLD) domain profile. / 60s Ribosomal Protein L30; Chain: A; / Alpha tubulin / Tubulin-beta mRNA autoregulation signal. / Beta tubulin, autoregulation binding site / Beta tubulin / Tubulin / Tubulin, C-terminal / Tubulin C-terminal domain / Tubulin, conserved site / Tubulin subunits alpha, beta, and gamma signature. / Tubulin/FtsZ family, C-terminal domain / Tubulin/FtsZ-like, C-terminal domain / Tubulin/FtsZ, C-terminal / Tubulin/FtsZ, 2-layer sandwich domain / Tubulin/FtsZ family, GTPase domain / Tubulin/FtsZ family, GTPase domain / Tubulin/FtsZ, GTPase domain / Tubulin/FtsZ, GTPase domain superfamily / Helix non-globular / Special / Helix Hairpins / Rossmann fold / 2-Layer Sandwich / Orthogonal Bundle / 3-Layer(aba) Sandwich / Mainly Alpha / Alpha Beta類似検索 - ドメイン・相同性 2-methyl-L-alanyl-N-[(3R,4S,5S)-3-methoxy-1-{(2S)-2-[(1R,2R)-1-methoxy-2-methyl-3-oxo-3-{[(1S)-2-phenyl-1-(1,3-thiazol-2-yl)ethyl]amino}propyl]pyrrolidin-1-yl}-5-methyl-1-oxoheptan-4-yl]-N-methyl-L-valinamide / Chem-3WD / GUANOSINE-5'-DIPHOSPHATE / GUANOSINE-5'-TRIPHOSPHATE / Chem-LOC / Tubulin beta chain / Tubulin alpha chain / Stathmin-4類似検索 - 構成要素 |
|---|
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ)
  Ovis aries (ヒツジ) Ovis aries (ヒツジ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.11 Å 分子置換 / 解像度: 3.11 Å |
|---|
 データ登録者 データ登録者 | Parris, K.D. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2014 ジャーナル: J.Med.Chem. / 年: 2014
タイトル: Discovery of cytotoxic dolastatin 10 analogues with N-terminal modifications.
著者: Maderna, A. / Doroski, M. / Subramanyam, C. / Porte, A. / Leverett, C.A. / Vetelino, B.C. / Chen, Z. / Risley, H. / Parris, K. / Pandit, J. / Varghese, A.H. / Shanker, S. / Song, C. / Sukuru, ...著者: Maderna, A. / Doroski, M. / Subramanyam, C. / Porte, A. / Leverett, C.A. / Vetelino, B.C. / Chen, Z. / Risley, H. / Parris, K. / Pandit, J. / Varghese, A.H. / Shanker, S. / Song, C. / Sukuru, S.C. / Farley, K.A. / Wagenaar, M.M. / Shapiro, M.J. / Musto, S. / Lam, M.H. / Loganzo, F. / O'Donnell, C.J. |
|---|
| 履歴 | | 登録 | 2014年11月24日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2015年3月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年4月8日 | Group: Non-polymer description |
|---|
| 改定 1.2 | 2017年11月22日 | Group: Derived calculations / Refinement description / Source and taxonomy
カテゴリ: entity_src_gen / entity_src_nat ...entity_src_gen / entity_src_nat / pdbx_struct_oper_list / software
Item: _entity_src_gen.pdbx_alt_source_flag / _entity_src_nat.pdbx_alt_source_flag / _pdbx_struct_oper_list.symmetry_operation |
|---|
| 改定 1.3 | 2023年9月27日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3.11 Å
分子置換 / 解像度: 3.11 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2014
ジャーナル: J.Med.Chem. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4x1i.cif.gz
4x1i.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4x1i.ent.gz
pdb4x1i.ent.gz PDB形式
PDB形式 4x1i.json.gz
4x1i.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/x1/4x1i
https://data.pdbj.org/pub/pdb/validation_reports/x1/4x1i ftp://data.pdbj.org/pub/pdb/validation_reports/x1/4x1i
ftp://data.pdbj.org/pub/pdb/validation_reports/x1/4x1i リンク
リンク 集合体
集合体
 要素
要素






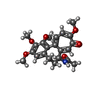
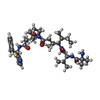





 タイプ: peptide-like, Peptide-like / クラス: 阻害剤 / 分子量: 743.011 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C39H62N6O6S
タイプ: peptide-like, Peptide-like / クラス: 阻害剤 / 分子量: 743.011 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C39H62N6O6S X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 17-ID / 波長: 1 Å
/ ビームライン: 17-ID / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



























 PDBj
PDBj






