+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4phv | ||||||
|---|---|---|---|---|---|---|---|
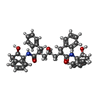
| タイトル | X-RAY CRYSTAL STRUCTURE OF THE HIV PROTEASE COMPLEX WITH L-700,417, AN INHIBITOR WITH PSEUDO C2 SYMMETRY | ||||||
 要素 要素 | HIV-1 PROTEASE | ||||||
 キーワード キーワード | HYDROLASE(ASPARTIC PROTEINASE) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報HIV-1 retropepsin / symbiont-mediated activation of host apoptosis / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / host multivesicular body / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency ...HIV-1 retropepsin / symbiont-mediated activation of host apoptosis / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / host multivesicular body / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / viral penetration into host nucleus / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / host cell / viral nucleocapsid / DNA recombination / DNA-directed DNA polymerase / aspartic-type endopeptidase activity / 加水分解酵素; エステル加水分解酵素 / DNA-directed DNA polymerase activity / symbiont-mediated suppression of host gene expression / viral translational frameshifting / lipid binding / symbiont entry into host cell / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / proteolysis / DNA binding / zinc ion binding / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||
| 手法 |  X線回折 / 解像度: 2.1 Å X線回折 / 解像度: 2.1 Å | ||||||
 データ登録者 データ登録者 | Bone, R. | ||||||
 引用 引用 | ジャーナル: J.Am.Chem.Soc. / 年: 1991 タイトル: X-Ray Crystal Structure of the HIV Protease Complex with L-700,417, an Inhibitor with Pseudo C2 Symmetry 著者: Bone, R. / Vacca, J.P. / Anderson, P.S. / Holloway, M.K. #1:  ジャーナル: Science / 年: 1990 ジャーナル: Science / 年: 1990タイトル: Design, Activity, and 2.8 Angstroms Crystal Structure of a C2 Symmetric Inhibitor Complexed to HIV-1 Protease 著者: Erickson, J. / Neidhart, D.J. / Van Drie, J. / Kempf, D.J. / Wang, X.C. / Norbeck, D.W. / Plattner, J.J. / Rittenhouse, J.W. / Turon, M. / Wideburg, N. / Kohlbrenner, W.E. / Simmer, R. / ...著者: Erickson, J. / Neidhart, D.J. / Van Drie, J. / Kempf, D.J. / Wang, X.C. / Norbeck, D.W. / Plattner, J.J. / Rittenhouse, J.W. / Turon, M. / Wideburg, N. / Kohlbrenner, W.E. / Simmer, R. / Helfrich, R. / Paul, D.A. / Knigge, M. #2:  ジャーナル: J.Biol.Chem. / 年: 1990 ジャーナル: J.Biol.Chem. / 年: 1990タイトル: Crystallographic Analysis of a Complex between Human Immunodeficiency Virus Type 1 Protease and Acetyl-Pepstatin at 2.0 Angstroms Resolution 著者: Fitzgerald, P.M.D. / Mckeever, B.M. / Van Middlesworth, J.F. / Springer, J.P. / Heimbach, J.C. / Leu, C.-T. / Herber, W.K. / Dixon, R.A.F. / Darke, P.L. #3:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1990 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1990タイトル: X-Ray Crystallographic Structure of a Complex between a Synthetic Protease of Human Immunodeficiency Virus 1 and a Substrate-Based Hydroxyethylamine Inhibitor 著者: Swain, A.L. / Miller, M.M. / Green, J. / Rich, D.H. / Schneider, J. / Kent, S.B.H. / Wlodawer, A. #4:  ジャーナル: Nature / 年: 1989 ジャーナル: Nature / 年: 1989タイトル: Three-Dimensional Structure of Aspartyl Protease from Human Immunodeficiency Virus HIV-1 著者: Navia, M.A. / Fitzgerald, P.M.D. / Mckeever, B.M. / Leu, C.-T. / Heimbach, J.C. / Herber, W.K. / Sigal, I.S. / Darke, P.L. / Springer, J.P. #5:  ジャーナル: Science / 年: 1989 ジャーナル: Science / 年: 1989タイトル: Conserved Folding in Retroviral Proteases: Crystal Structure of a Synthetic HIV-1 Protease 著者: Wlodawer, A. / Miller, M. / Jaskolski, M. / Sathyanarayana, B.K. / Baldwin, E. / Weber, I.T. / Selk, L. / Clawson, L. / Schneider, J. / Kent, S.B.H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4phv.cif.gz 4phv.cif.gz | 55.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4phv.ent.gz pdb4phv.ent.gz | 40.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4phv.json.gz 4phv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4phv_validation.pdf.gz 4phv_validation.pdf.gz | 535.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4phv_full_validation.pdf.gz 4phv_full_validation.pdf.gz | 544.2 KB | 表示 | |
| XML形式データ |  4phv_validation.xml.gz 4phv_validation.xml.gz | 7.4 KB | 表示 | |
| CIF形式データ |  4phv_validation.cif.gz 4phv_validation.cif.gz | 10.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ph/4phv https://data.pdbj.org/pub/pdb/validation_reports/ph/4phv ftp://data.pdbj.org/pub/pdb/validation_reports/ph/4phv ftp://data.pdbj.org/pub/pdb/validation_reports/ph/4phv | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 10830.781 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)属: Lentivirus / 発現宿主:  #2: 化合物 | #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.76 Å3/Da / 溶媒含有率: 55.41 % | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS pH: 5.4 / 手法: 蒸気拡散法 / 詳細: macro-seeding | |||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 反射 | *PLUS 最高解像度: 2.1 Å / % possible obs: 97.2 % / Observed criterion σ(I): 2 / Rmerge(I) obs: 0.07 |
|---|
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.1→8 Å / σ(F): 0 /
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→8 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: PROLSQ / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.1 Å / 最低解像度: 8 Å / Num. reflection all: 12981 / σ(F): 0 / Rfactor all: 0.18 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj