[English] 日本語
 Yorodumi
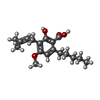
Yorodumi- PDB-4a4v: Ligand binding domain of human PPAR gamma in complex with amorfrutin 2 -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4a4v | ||||||
|---|---|---|---|---|---|---|---|
| Title | Ligand binding domain of human PPAR gamma in complex with amorfrutin 2 | ||||||
 Components Components | PEROXISOME PROLIFERATOR-ACTIVATED RECEPTOR GAMMA | ||||||
 Keywords Keywords | RECEPTOR / AGONIST / DIABETES / INSULIN RESISTANCE | ||||||
| Function / homology |  Function and homology information Function and homology informationprostaglandin receptor activity / beige fat cell differentiation / negative regulation of receptor signaling pathway via STAT / MECP2 regulates transcription factors / negative regulation of vascular endothelial cell proliferation / negative regulation of extracellular matrix assembly / negative regulation of connective tissue replacement involved in inflammatory response wound healing / positive regulation of cholesterol transport / negative regulation of cellular response to transforming growth factor beta stimulus / arachidonate binding ...prostaglandin receptor activity / beige fat cell differentiation / negative regulation of receptor signaling pathway via STAT / MECP2 regulates transcription factors / negative regulation of vascular endothelial cell proliferation / negative regulation of extracellular matrix assembly / negative regulation of connective tissue replacement involved in inflammatory response wound healing / positive regulation of cholesterol transport / negative regulation of cellular response to transforming growth factor beta stimulus / arachidonate binding / positive regulation of adiponectin secretion / DNA binding domain binding / positive regulation of vascular associated smooth muscle cell apoptotic process / negative regulation of cardiac muscle hypertrophy in response to stress / positive regulation of fatty acid metabolic process / STAT family protein binding / WW domain binding / positive regulation of lipid metabolic process / response to lipid / negative regulation of type II interferon-mediated signaling pathway / LBD domain binding / negative regulation of cholesterol storage / positive regulation of lipoprotein transport / negative regulation of SMAD protein signal transduction / lipid homeostasis / E-box binding / alpha-actinin binding / R-SMAD binding / negative regulation of vascular associated smooth muscle cell proliferation / negative regulation of blood vessel endothelial cell migration / white fat cell differentiation / negative regulation of macrophage derived foam cell differentiation / negative regulation of lipid storage / positive regulation of cholesterol efflux / negative regulation of BMP signaling pathway / monocyte differentiation / cell fate commitment / cellular response to low-density lipoprotein particle stimulus / negative regulation of mitochondrial fission / negative regulation of osteoblast differentiation / long-chain fatty acid transport / BMP signaling pathway / positive regulation of fat cell differentiation / nuclear retinoid X receptor binding / fat cell differentiation / Transcriptional regulation of brown and beige adipocyte differentiation by EBF2 / retinoic acid receptor signaling pathway / negative regulation of MAPK cascade / intracellular receptor signaling pathway / positive regulation of adipose tissue development / cell maturation / hormone-mediated signaling pathway / peroxisome proliferator activated receptor signaling pathway / epithelial cell differentiation / regulation of cellular response to insulin stimulus / peptide binding / response to nutrient / brown fat cell differentiation / negative regulation of miRNA transcription / placenta development / negative regulation of angiogenesis / Regulation of PTEN gene transcription / transcription coregulator binding / positive regulation of apoptotic signaling pathway / SUMOylation of intracellular receptors / negative regulation of smooth muscle cell proliferation / negative regulation of transforming growth factor beta receptor signaling pathway / PPARA activates gene expression / fatty acid metabolic process / regulation of circadian rhythm / Nuclear Receptor transcription pathway / Transcriptional regulation of white adipocyte differentiation / mRNA transcription by RNA polymerase II / positive regulation of miRNA transcription / negative regulation of inflammatory response / DNA-binding transcription repressor activity, RNA polymerase II-specific / regulation of blood pressure / RNA polymerase II transcription regulator complex / nuclear receptor activity / cellular response to insulin stimulus / rhythmic process / glucose homeostasis / MLL4 and MLL3 complexes regulate expression of PPARG target genes in adipogenesis and hepatic steatosis / DNA-binding transcription activator activity, RNA polymerase II-specific / double-stranded DNA binding / cellular response to hypoxia / sequence-specific DNA binding / DNA-binding transcription factor binding / nucleic acid binding / DNA-binding transcription factor activity, RNA polymerase II-specific / cell differentiation / receptor complex / transcription cis-regulatory region binding / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / negative regulation of gene expression / innate immune response / negative regulation of DNA-templated transcription / chromatin binding / positive regulation of gene expression Similarity search - Function | ||||||
| Biological species |  HOMO SAPIENS (human) HOMO SAPIENS (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  MOLECULAR REPLACEMENT / Resolution: 2 Å MOLECULAR REPLACEMENT / Resolution: 2 Å | ||||||
 Authors Authors | de Groot, J.C. / Weidner, C. / Krausze, J. / Kawamoto, K. / Schroeder, F.C. / Sauer, S. / Buessow, K. | ||||||
 Citation Citation |  Journal: J.Med.Chem. / Year: 2013 Journal: J.Med.Chem. / Year: 2013Title: Structural Characterization of Amorfrutins Bound to the Peroxisome Proliferator-Activated Receptor Gamma. Authors: De Groot, J.C. / Weidner, C. / Krausze, J. / Kawamoto, K. / Schroeder, F.C. / Sauer, S. / Bussow, K. | ||||||
| History |
| ||||||
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR PROVIDED. | ||||||
| Remark 700 | SHEET DETERMINATION METHOD: AUTHOR PROVIDED. |
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4a4v.cif.gz 4a4v.cif.gz | 239.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4a4v.ent.gz pdb4a4v.ent.gz | 194.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4a4v.json.gz 4a4v.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/a4/4a4v https://data.pdbj.org/pub/pdb/validation_reports/a4/4a4v ftp://data.pdbj.org/pub/pdb/validation_reports/a4/4a4v ftp://data.pdbj.org/pub/pdb/validation_reports/a4/4a4v | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4a4wC  1prgS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
| ||||||||
| Noncrystallographic symmetry (NCS) | NCS oper: (Code: given Matrix: (0.09994, 0.97651, 0.19092), Vector: |
- Components
Components
| #1: Protein | Mass: 32693.824 Da / Num. of mol.: 2 / Fragment: LIGAND BINDING DOMAIN, RESIDUES 195-477 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  HOMO SAPIENS (human) / Production host: HOMO SAPIENS (human) / Production host:  #2: Chemical | #3: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.48 Å3/Da / Density % sol: 50.39 % / Description: NONE |
|---|---|
| Crystal grow | pH: 8 / Details: 0.8 M TRI-SODIUM CITRATE, 0.1 M IMIDAZOLE, PH 8.0 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  ROTATING ANODE / Type: RIGAKU MICROMAX-007 HF / Wavelength: 1.5419 ROTATING ANODE / Type: RIGAKU MICROMAX-007 HF / Wavelength: 1.5419 |
| Detector | Type: RIGAKU-MSC SATURN 944 PLUS / Detector: CCD / Date: Sep 25, 2010 |
| Radiation | Monochromator: RIGAKU VARIMAX / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.5419 Å / Relative weight: 1 |
| Reflection | Resolution: 2→27 Å / Num. obs: 43005 / % possible obs: 99 % / Observed criterion σ(I): 3 / Redundancy: 3.5 % / Rmerge(I) obs: 0.11 / Net I/σ(I): 13.7 |
| Reflection shell | Resolution: 2→2.1 Å / Redundancy: 3.2 % / Rmerge(I) obs: 0.56 / Mean I/σ(I) obs: 2.8 / % possible all: 99.5 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 1PRG Resolution: 2→27.45 Å / Cor.coef. Fo:Fc: 0.954 / Cor.coef. Fo:Fc free: 0.926 / SU B: 10.761 / SU ML: 0.137 / Cross valid method: THROUGHOUT / ESU R: 0.195 / ESU R Free: 0.178 / Stereochemistry target values: MAXIMUM LIKELIHOOD / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.4 Å / Solvent model: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 42.458 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2→27.45 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller




































 PDBj
PDBj