+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4521 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EM structure of a EBOV-GP bound to 4M0368 neutralizing antibody | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell endoplasmic reticulum / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / membrane raft / fusion of virus membrane with host endosome membrane / viral envelope ...host cell endoplasmic reticulum / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / suppression by virus of host tetherin activity / host cell cytoplasm / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / membrane raft / fusion of virus membrane with host endosome membrane / viral envelope / lipid binding / host cell plasma membrane / virion membrane / extracellular region / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Diskin R / Cohen-Dvashi H | |||||||||
 引用 引用 |  ジャーナル: Nat Med / 年: 2019 ジャーナル: Nat Med / 年: 2019タイトル: Polyclonal and convergent antibody response to Ebola virus vaccine rVSV-ZEBOV. 著者: Stefanie A Ehrhardt / Matthias Zehner / Verena Krähling / Hadas Cohen-Dvashi / Christoph Kreer / Nadav Elad / Henning Gruell / Meryem S Ercanoglu / Philipp Schommers / Lutz Gieselmann / Ralf ...著者: Stefanie A Ehrhardt / Matthias Zehner / Verena Krähling / Hadas Cohen-Dvashi / Christoph Kreer / Nadav Elad / Henning Gruell / Meryem S Ercanoglu / Philipp Schommers / Lutz Gieselmann / Ralf Eggeling / Christine Dahlke / Timo Wolf / Nico Pfeifer / Marylyn M Addo / Ron Diskin / Stephan Becker / Florian Klein /   要旨: Recombinant vesicular stomatitis virus-Zaire Ebola virus (rVSV-ZEBOV) is the most advanced Ebola virus vaccine candidate and is currently being used to combat the outbreak of Ebola virus disease (EVD) ...Recombinant vesicular stomatitis virus-Zaire Ebola virus (rVSV-ZEBOV) is the most advanced Ebola virus vaccine candidate and is currently being used to combat the outbreak of Ebola virus disease (EVD) in the Democratic Republic of the Congo (DRC). Here we examine the humoral immune response in a subset of human volunteers enrolled in a phase 1 rVSV-ZEBOV vaccination trial by performing comprehensive single B cell and electron microscopy structure analyses. Four studied vaccinees show polyclonal, yet reproducible and convergent B cell responses with shared sequence characteristics. EBOV-targeting antibodies cross-react with other Ebolavirus species, and detailed epitope mapping revealed overlapping target epitopes with antibodies isolated from EVD survivors. Moreover, in all vaccinees, we detected highly potent EBOV-neutralizing antibodies with activities comparable or superior to the monoclonal antibodies currently used in clinical trials. These include antibodies combining the IGHV3-15/IGLV1-40 immunoglobulin gene segments that were identified in all investigated individuals. Our findings will help to evaluate and direct current and future vaccination strategies and offer opportunities for novel EVD therapies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4521.map.gz emd_4521.map.gz | 6.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4521-v30.xml emd-4521-v30.xml emd-4521.xml emd-4521.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4521_fsc.xml emd_4521_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4521.png emd_4521.png | 125.3 KB | ||
| その他 |  emd_4521_half_map_1.map.gz emd_4521_half_map_1.map.gz emd_4521_half_map_2.map.gz emd_4521_half_map_2.map.gz | 139.3 MB 139.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4521 http://ftp.pdbj.org/pub/emdb/structures/EMD-4521 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4521 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4521 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4521_validation.pdf.gz emd_4521_validation.pdf.gz | 476.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4521_full_validation.pdf.gz emd_4521_full_validation.pdf.gz | 475.8 KB | 表示 | |
| XML形式データ |  emd_4521_validation.xml.gz emd_4521_validation.xml.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4521 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4521 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4521 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4521 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4521.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4521.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_4521_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
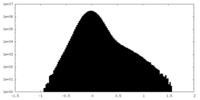
| 投影像・断面図 |
| ||||||||||||
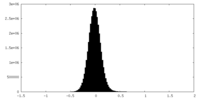
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_4521_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
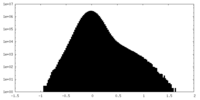
| 投影像・断面図 |
| ||||||||||||
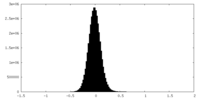
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EBOV-GP in complex with neutralizing 3T0331 antibody
| 全体 | 名称: EBOV-GP in complex with neutralizing 3T0331 antibody |
|---|---|
| 要素 |
|
-超分子 #1: EBOV-GP in complex with neutralizing 3T0331 antibody
| 超分子 | 名称: EBOV-GP in complex with neutralizing 3T0331 antibody タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|
-超分子 #2: EBOV-GP
| 超分子 | 名称: EBOV-GP / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-超分子 #3: neutralizing 3T0331 antibody
| 超分子 | 名称: neutralizing 3T0331 antibody / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1, #4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-分子 #1: Light chain
| 分子 | 名称: Light chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.4119 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQLTQSPSS LSASVGDRVT ITCRASQSIS TYLNWYQQKP GKAPKFLIFS ASNLQSGVPS RFSGGGSGTD FTLTISSLQP EDFATYYCQ QTYNTPRTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQLTQSPSS LSASVGDRVT ITCRASQSIS TYLNWYQQKP GKAPKFLIFS ASNLQSGVPS RFSGGGSGTD FTLTISSLQP EDFATYYCQ QTYNTPRTFG QGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #2: Envelope glycoprotein,Virion spike glycoprotein,EBOV-GP1
| 分子 | 名称: Envelope glycoprotein,Virion spike glycoprotein,EBOV-GP1 タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.887578 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GSRSIPLGVI HNSALQVSDV DKLVCRDKLS STNQLRSVGL NLEGNGVATD VPSATKRWGF RSGVPPKVVN YEAGEWAENC YNLEIKKPD GSECLPAAPD GIRGFPRCRY VHKVSGTGPC AGDFAFHKEG AFFLYDRLAS TVIYRGTTFA EGVVAFLILP Q AKKDFFSS ...文字列: GSRSIPLGVI HNSALQVSDV DKLVCRDKLS STNQLRSVGL NLEGNGVATD VPSATKRWGF RSGVPPKVVN YEAGEWAENC YNLEIKKPD GSECLPAAPD GIRGFPRCRY VHKVSGTGPC AGDFAFHKEG AFFLYDRLAS TVIYRGTTFA EGVVAFLILP Q AKKDFFSS HPLREPVNAT EDPSSGYYST TIRYQATGFG TNETEYLFEV DNLTYVQLES RFTPQFLLQL NETIYTSGKR SN TTGKLIW KVNPEIDTTI GEWAFWETKK NLTRKIRSEE LSFTVV(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #3: Envelope glycoprotein
| 分子 | 名称: Envelope glycoprotein / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 18.989391 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EAIVNAQPKC NPNLHYWTTQ DEGAAIGLAW IPYFGPAAEG IYIEGLMHNQ DGLICGLRQL ANETTQALQL FLRATTELRT FSILNRKAI DFLLQRWGGT CHILGPDCCI EPHDWTKNIT DKIDQIIHDF VDGSGYIPEA PRDGQAYVRK DGEWVLLSTF L GTHHHHHH |
-分子 #4: Heavy chain
| 分子 | 名称: Heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.091916 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSGTLSL TCAVSGGSIS GTNWWTWVRQ PPGRGLEWIG EIYYRGSPNY NPSLKSRVTI SVDQSKSQFS LKLTSVTAA DTAVYYCLRL SHYGGAWGQG TLVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS G ALTSGVHT ...文字列: QVQLQESGPG LVKPSGTLSL TCAVSGGSIS GTNWWTWVRQ PPGRGLEWIG EIYYRGSPNY NPSLKSRVTI SVDQSKSQFS LKLTSVTAA DTAVYYCLRL SHYGGAWGQG TLVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS G ALTSGVHT FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KVDKRVEPKS C |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 27.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X