+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4461 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | High resolution electron cryo-microscopy structure of the bacteriophage PR772 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Phage / Tectiviridae / Membrane / double-barrel / helix turn helix / helix with a kink / beta-propeller / heteropentamer / penton / VIRUS | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Enterobacteria phage PR772 (ファージ) Enterobacteria phage PR772 (ファージ) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.75 Å | ||||||||||||
 データ登録者 データ登録者 | Narayana Reddy HK / Svenda M | ||||||||||||
| 資金援助 |  スウェーデン, 3件 スウェーデン, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Electron cryo-microscopy of bacteriophage PR772 reveals the elusive vertex complex and the capsid architecture. 著者: Hemanth Kn Reddy / Marta Carroni / Janos Hajdu / Martin Svenda /   要旨: Bacteriophage PR772, a member of the family, has a 70 nm diameter icosahedral protein capsid that encapsulates a lipid membrane, dsDNA, and various internal proteins. An icosahedrally averaged ...Bacteriophage PR772, a member of the family, has a 70 nm diameter icosahedral protein capsid that encapsulates a lipid membrane, dsDNA, and various internal proteins. An icosahedrally averaged CryoEM reconstruction of the wild-type virion and a localized reconstruction of the vertex region reveal the composition and the structure of the vertex complex along with new protein conformations that play a vital role in maintaining the capsid architecture of the virion. The overall resolution of the virion is 2.75 Å, while the resolution of the protein capsid is 2.3 Å. The conventional penta-symmetron formed by the capsomeres is replaced by a large vertex complex in the pseudo T = 25 capsid. All the vertices contain the host-recognition protein, P5; two of these vertices show the presence of the receptor-binding protein, P2. The 3D structure of the vertex complex shows interactions with the viral membrane, indicating a possible mechanism for viral infection. #1:  ジャーナル: Biorxiv / 年: 2019 ジャーナル: Biorxiv / 年: 2019タイトル: Electron cryo-microscopy of Bacteriophage PR772 reveals the composition and structure of the elusive vertex complex and the capsid architecture 著者: Narayana Reddy HK / Hajdu J / Carroni M / Svenda M | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4461.map.gz emd_4461.map.gz | 2 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4461-v30.xml emd-4461-v30.xml emd-4461.xml emd-4461.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
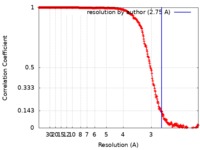
| FSC (解像度算出) |  emd_4461_fsc.xml emd_4461_fsc.xml | 30 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4461.png emd_4461.png | 335.9 KB | ||
| Filedesc metadata |  emd-4461.cif.gz emd-4461.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4461 http://ftp.pdbj.org/pub/emdb/structures/EMD-4461 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4461 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4461 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4461_validation.pdf.gz emd_4461_validation.pdf.gz | 316.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4461_full_validation.pdf.gz emd_4461_full_validation.pdf.gz | 315.8 KB | 表示 | |
| XML形式データ |  emd_4461_validation.xml.gz emd_4461_validation.xml.gz | 23.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4461 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4461 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4461 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4461 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4461.map.gz / 形式: CCP4 / 大きさ: 2.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4461.map.gz / 形式: CCP4 / 大きさ: 2.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Bacteriophage PR772
| 全体 | 名称: Bacteriophage PR772 |
|---|---|
| 要素 |
|
-超分子 #1: Bacteriophage PR772
| 超分子 | 名称: Bacteriophage PR772 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Wild type |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage PR772 (ファージ) Enterobacteria phage PR772 (ファージ) |
| 分子量 | 理論値: 86 MDa |
-分子 #1: Major Capsid Protein (P3)
| 分子 | 名称: Major Capsid Protein (P3) / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage PR772 (ファージ) Enterobacteria phage PR772 (ファージ) |
| 分子量 | 理論値: 43.505492 KDa |
| 配列 | 文字列: MAQVQQLTPA QQAALRNQQA MAANLQARQI VLQQSYPVIQ QVETQTFDPA NRSVFDVTPA NVGIVKGFLV KVTAAIKNNH ATEAVALTD FGPANLVQRV IYYDPDNQRH TETSGWHLHF VNTAKQGAPF LSSMVTDSPI KYGDVMNVID APATIAAGAT G ELTMYYWV ...文字列: MAQVQQLTPA QQAALRNQQA MAANLQARQI VLQQSYPVIQ QVETQTFDPA NRSVFDVTPA NVGIVKGFLV KVTAAIKNNH ATEAVALTD FGPANLVQRV IYYDPDNQRH TETSGWHLHF VNTAKQGAPF LSSMVTDSPI KYGDVMNVID APATIAAGAT G ELTMYYWV PLAYSETDLT GAVLANVPQS KQRLKLEFAN NNTAFAAVGA NPLEAIYQGA GAADCEFEEI SYTVYQSYLD QL PVGQNGY ILPLIDLSTL YNLENSAQAG LTPNVDFVVQ YANLYRYLST IAVFDNGGSF NAGTDINYLS QRTANFSDTR KLD PKTWAA QTRRRIATDF PKGVYYCDNR DKPIYTLQYG NVGFVVNPKT VNQNARLLMG YEYFTSRTEL VNAGTISTT UniProtKB: Major capsid protein |
-分子 #2: Minor Capsid Protein (P30)
| 分子 | 名称: Minor Capsid Protein (P30) / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage PR772 (ファージ) Enterobacteria phage PR772 (ファージ) |
| 分子量 | 理論値: 9.275559 KDa |
| 配列 | 文字列: MALINPQFPY AGPVPIPGPA PTETMPLLNY RVEGRIAGIQ QARQFMPFLQ GPHREVAEQT YYAIGTGIQM GQTFNQPLIN TQEG UniProtKB: Minor capsid protein |
-分子 #3: Infectivity Protein (P16)
| 分子 | 名称: Infectivity Protein (P16) / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage PR772 (ファージ) Enterobacteria phage PR772 (ファージ) |
| 分子量 | 理論値: 12.616262 KDa |
| 配列 | 文字列: MDKKKLLYWV GGGLVLIIIW LWFRNRPAAQ VASNWEGPPY MTYNQPQAGS VTLPVAGYTS PSLTLPNRNR SCGCNPAVSA AMAQGADLA SKLTESISSQ LNNYAESLND YLASQAGV UniProtKB: Infectivity protein |
-分子 #4: Spike protein
| 分子 | 名称: Spike protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage PR772 (ファージ) Enterobacteria phage PR772 (ファージ) |
| 分子量 | 理論値: 34.483898 KDa |
| 配列 | 文字列: MANQQIGGST VTYNGAIPMG GPVAINSVIE IAGTEVLVDL KLDYATGKIS GVQTLYIDLR DFLGDVTVTM PDTGQRITAR AGTQGYYPV LSTNLMKFIV SATIDGKFPM NFINFPIALG VWPSGIKGDK GDPGAPGPAG GTVVVEDSGA SFGESLLDTT S EPGKILVK ...文字列: MANQQIGGST VTYNGAIPMG GPVAINSVIE IAGTEVLVDL KLDYATGKIS GVQTLYIDLR DFLGDVTVTM PDTGQRITAR AGTQGYYPV LSTNLMKFIV SATIDGKFPM NFINFPIALG VWPSGIKGDK GDPGAPGPAG GTVVVEDSGA SFGESLLDTT S EPGKILVK RISGGSGITV TDYGDEVEIE ASGGGGGGGG VTDALSLMYS TSTGGPASIA ANALTDFDLS GALTVNTVGT GL TKSAAGI QLAAGKSGLY QITMTVKNNT VTTGNYLLRV KYGSSDFVVA CPASSLTAGG TISLLIYCDV LGVPSLDVLK FSL CNDGAA LSNYIINITA AKIN UniProtKB: Spike |
-分子 #5: Penton protein
| 分子 | 名称: Penton protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage PR772 (ファージ) Enterobacteria phage PR772 (ファージ) |
| 分子量 | 理論値: 13.757388 KDa |
| 配列 | 文字列: MNVNNPNQMT VTPVYNGCDS GEGPQSVRGY FDAVAGENVK YDLTYLADTQ GFTGVQCIYI DNAENDGAFE IDVEETGQRI KCPAGKQGY FPLLVPGRAK FVARHLGSGK KSVPLFFLNF TIAQGVW UniProtKB: Penton |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 7 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) デジタル化 - 画像ごとのフレーム数: 3-40 / 撮影したグリッド数: 1 / 実像数: 3200 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| ソフトウェア | 名称:  Coot (ver. 0.8.9.1) Coot (ver. 0.8.9.1) |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 104.93 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-6q5u: |
 ムービー
ムービー コントローラー
コントローラー



















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)