+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Pseudomonas phage DEV neck and tail (portal, head-to-tail and tail tube proteins) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | portal / tail hub / tail tube / complex / STRUCTURAL PROTEIN / gp75 / gp83 / gp80 / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Tail protein / N4 gp54-like protein / Portal protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas phage vB_PaeP_DEV (ファージ) Pseudomonas phage vB_PaeP_DEV (ファージ) | |||||||||
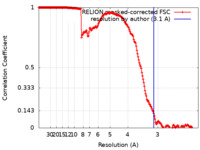
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Iglesias SM / Hou CFD / Li F / Cingolani G | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Integrative structural analysis of Pseudomonas phage DEV reveals a genome ejection motor. 著者: Ravi K Lokareddy / Chun-Feng David Hou / Francesca Forti / Stephano M Iglesias / Fenglin Li / Mikhail Pavlenok / David S Horner / Michael Niederweis / Federica Briani / Gino Cingolani /   要旨: DEV is an obligatory lytic Pseudomonas phage of the N4-like genus, recently reclassified as Schitoviridae. The DEV genome encodes 91 ORFs, including a 3398 amino acid virion-associated RNA polymerase ...DEV is an obligatory lytic Pseudomonas phage of the N4-like genus, recently reclassified as Schitoviridae. The DEV genome encodes 91 ORFs, including a 3398 amino acid virion-associated RNA polymerase (vRNAP). Here, we describe the complete architecture of DEV, determined using a combination of cryo-electron microscopy localized reconstruction, biochemical methods, and genetic knockouts. We built de novo structures of all capsid factors and tail components involved in host attachment. We demonstrate that DEV long tail fibers are essential for infection of Pseudomonas aeruginosa but dispensable for infecting mutants with a truncated lipopolysaccharide devoid of the O-antigen. We determine that DEV vRNAP is part of a three-gene operon conserved in 191 Schitoviridae genomes. We propose these three proteins are ejected into the host to form a genome ejection motor spanning the cell envelope. We posit that the design principles of the DEV ejection apparatus are conserved in all Schitoviridae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44517.map.gz emd_44517.map.gz | 400.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44517-v30.xml emd-44517-v30.xml emd-44517.xml emd-44517.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44517_fsc.xml emd_44517_fsc.xml | 18 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44517.png emd_44517.png | 33.4 KB | ||
| マスクデータ |  emd_44517_msk_1.map emd_44517_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-44517.cif.gz emd-44517.cif.gz | 6 KB | ||
| その他 |  emd_44517_half_map_1.map.gz emd_44517_half_map_1.map.gz emd_44517_half_map_2.map.gz emd_44517_half_map_2.map.gz | 405.7 MB 405.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44517 http://ftp.pdbj.org/pub/emdb/structures/EMD-44517 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44517 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44517 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44517_validation.pdf.gz emd_44517_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44517_full_validation.pdf.gz emd_44517_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_44517_validation.xml.gz emd_44517_validation.xml.gz | 26 KB | 表示 | |
| CIF形式データ |  emd_44517_validation.cif.gz emd_44517_validation.cif.gz | 34.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44517 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44517 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44517 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44517 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44517.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44517.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||
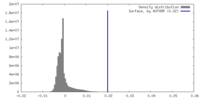
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_44517_msk_1.map emd_44517_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
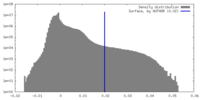
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_44517_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_44517_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
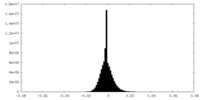
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Portal to tail complex of Pseudomonas phage DEV
| 全体 | 名称: Portal to tail complex of Pseudomonas phage DEV |
|---|---|
| 要素 |
|
-超分子 #1: Portal to tail complex of Pseudomonas phage DEV
| 超分子 | 名称: Portal to tail complex of Pseudomonas phage DEV / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage vB_PaeP_DEV (ファージ) Pseudomonas phage vB_PaeP_DEV (ファージ) |
-分子 #1: gp75 tail tube
| 分子 | 名称: gp75 tail tube / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage vB_PaeP_DEV (ファージ) Pseudomonas phage vB_PaeP_DEV (ファージ) |
| 分子量 | 理論値: 35.267215 KDa |
| 配列 | 文字列: MAVEPITIAD LTEVKLDGKG ALDQLLQVTR LHLAKEHDAG RLKGQEYAAV LTGGITAVLQ NAVMFLLQKD EAANKAALVE AQIKLTEKQ GELLDKQIAQ ADKDAELIAA KVKLTLEQAK LPDSQIRSAG FQDLLVQEQT KVQTAQTRRI DQEILSAGFQ D LLVKEQTA ...文字列: MAVEPITIAD LTEVKLDGKG ALDQLLQVTR LHLAKEHDAG RLKGQEYAAV LTGGITAVLQ NAVMFLLQKD EAANKAALVE AQIKLTEKQ GELLDKQIAQ ADKDAELIAA KVKLTLEQAK LPDSQIRSAG FQDLLVQEQT KVQTAQTRRI DQEILSAGFQ D LLVKEQTA KTKQDVLTAV QQTKVMEQQV LESTQKVLNM KQELLNLVAQ ECLLKAQFDL TKDQGLNTQE QTILVRQKVA SE RAQTIGA GVDADSVIGR QKELYKAQAD GFKRDAEQKA AKILIDTWNV RRTTDTGTQA NTTNRLDDAN VGRVVNMLMT GVG A UniProtKB: N4 gp54-like protein |
-分子 #2: gp83 head-to-tail
| 分子 | 名称: gp83 head-to-tail / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage vB_PaeP_DEV (ファージ) Pseudomonas phage vB_PaeP_DEV (ファージ) |
| 分子量 | 理論値: 27.868934 KDa |
| 配列 | 文字列: MTIQLKQVID LLAEGELSNI KYVNIDTGAL VLERVPSLIR AINLGVLDLH KRFLLKEGML KIQLEEGRRL YPLRPAYQVG QKPKPGVPQ FITEGNKLGR QSILKIEKII GDNGVEYYLN DTWQPLNITT PEFDVLEISD EFYCHSSSKT LEVRYRRAPT P MKICVDNL ...文字列: MTIQLKQVID LLAEGELSNI KYVNIDTGAL VLERVPSLIR AINLGVLDLH KRFLLKEGML KIQLEEGRRL YPLRPAYQVG QKPKPGVPQ FITEGNKLGR QSILKIEKII GDNGVEYYLN DTWQPLNITT PEFDVLEISD EFYCHSSSKT LEVRYRRAPT P MKICVDNL DSWGCIDIDL PYTHLQALLY FVASRCQTPI GFMENTAQEG FNFSQKYEAE CANLDAQNLR IDPVGNQDRF TR GGWV UniProtKB: Tail protein |
-分子 #3: gp80 portal protein
| 分子 | 名称: gp80 portal protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage vB_PaeP_DEV (ファージ) Pseudomonas phage vB_PaeP_DEV (ファージ) |
| 分子量 | 理論値: 81.739453 KDa |
| 配列 | 文字列: MADVDEDYLT LPNEDGDPSK RLQPEWSNAP SLAQLKQDYQ EAKQVTDEKI TQINRWLDYM HVRGEGKPKT EKGKSAVQPP TIRKQAEWR YSSLSEPFLS SPNIFEVNPV TWEDAESARQ NGLVLNQQFN TKLNKQRFID EYVRAGVDEG TIIVKVGWNY Q SRTVKEQV ...文字列: MADVDEDYLT LPNEDGDPSK RLQPEWSNAP SLAQLKQDYQ EAKQVTDEKI TQINRWLDYM HVRGEGKPKT EKGKSAVQPP TIRKQAEWR YSSLSEPFLS SPNIFEVNPV TWEDAESARQ NGLVLNQQFN TKLNKQRFID EYVRAGVDEG TIIVKVGWNY Q SRTVKEQV VTYEMMPDSS EELAQIYQTA AQIREESPSE YPEIPEDVRL GLEETEANGI QVRAVPVGSE EEEREETVEN HP TVQVCDY NNIVIDPSCG SDFSKAKFLI ETFESSYAEL KADGRYKNLD KIQVEGQNLL SEPDYTGPSE GVRNFDFQDK SRK RLVVHE YWGYYDIHGD GVLHPIVATW VGAVMIRMEE NPFPDKKIPY VVVSYIPRKR DLYGESDGAL LIDNQRIIGA VTRG MIDTM ARSANGQVGV MKGALDVTNR RRFDRGENYE FNPGADPRAA VHMHTFPEIP QSAQYMINLQ QAEAESMTGV KAFNA GISG AALGDTATAV RGALDAASKR ELGILRRLSA GIIEIGRKII AMNAEFLDDV EVVRITNEHF VDIRRDDLAG NFDLKL DIS TAEEDNAKVN DLTFMLQTMG PNMDPMMAQQ IMGQIMELKK MPDFAKRIRE FQPQPDPIAQ QKAQLELMLL QAQIEAE RA RAAHYMSGAG LQDSKVGTEQ AKARALASQA DMTDLNFLEQ ESGVQQARKR ELQQAQSEAQ GKLAMLNSQL KRLDEATS A RTSQK UniProtKB: Portal protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)