+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of synaptic vesicle protein 2A in complex with a nanobody | |||||||||
 マップデータ マップデータ | DeepEMhancer sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Synaptic vesicle / SLC22 / Inhibitor / Nanobody / AEDs / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of gamma-aminobutyric acid secretion / Toxicity of botulinum toxin type F (botF) / Toxicity of botulinum toxin type D (botD) / Toxicity of botulinum toxin type E (botE) / Toxicity of botulinum toxin type A (botA) / synaptic vesicle priming / presynaptic active zone / transmembrane transporter activity / GABA-ergic synapse / neuromuscular junction ...regulation of gamma-aminobutyric acid secretion / Toxicity of botulinum toxin type F (botF) / Toxicity of botulinum toxin type D (botD) / Toxicity of botulinum toxin type E (botE) / Toxicity of botulinum toxin type A (botA) / synaptic vesicle priming / presynaptic active zone / transmembrane transporter activity / GABA-ergic synapse / neuromuscular junction / synaptic vesicle membrane / intracellular calcium ion homeostasis / cell-cell junction / synaptic vesicle / neuron projection / neuronal cell body / glutamatergic synapse / dendrite / protein kinase binding / endoplasmic reticulum / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
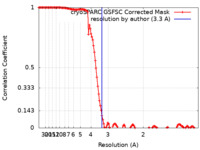
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Mittal A / Martin MF / Levin E / Adams C / Yang M / Ledecq M / Horanyi PS / Coleman JA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structures of synaptic vesicle protein 2A and 2B bound to anticonvulsants. 著者: Anshumali Mittal / Matthew F Martin / Elena J Levin / Christopher Adams / Meng Yang / Laurent Provins / Adrian Hall / Martin Procter / Marie Ledecq / Alexander Hillisch / Christian Wolff / ...著者: Anshumali Mittal / Matthew F Martin / Elena J Levin / Christopher Adams / Meng Yang / Laurent Provins / Adrian Hall / Martin Procter / Marie Ledecq / Alexander Hillisch / Christian Wolff / Michel Gillard / Peter S Horanyi / Jonathan A Coleman /     要旨: Epilepsy is a common neurological disorder characterized by abnormal activity of neuronal networks, leading to seizures. The racetam class of anti-seizure medications bind specifically to a membrane ...Epilepsy is a common neurological disorder characterized by abnormal activity of neuronal networks, leading to seizures. The racetam class of anti-seizure medications bind specifically to a membrane protein found in the synaptic vesicles of neurons called synaptic vesicle protein 2 (SV2) A (SV2A). SV2A belongs to an orphan subfamily of the solute carrier 22 organic ion transporter family that also includes SV2B and SV2C. The molecular basis for how anti-seizure medications act on SV2s remains unknown. Here we report cryo-electron microscopy structures of SV2A and SV2B captured in a luminal-occluded conformation complexed with anticonvulsant ligands. The conformation bound by anticonvulsants resembles an inhibited transporter with closed luminal and intracellular gates. Anticonvulsants bind to a highly conserved central site in SV2s. These structures provide blueprints for future drug design and will facilitate future investigations into the biological function of SV2s. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42431.map.gz emd_42431.map.gz | 180.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42431-v30.xml emd-42431-v30.xml emd-42431.xml emd-42431.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42431_fsc.xml emd_42431_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42431.png emd_42431.png | 45.7 KB | ||
| マスクデータ |  emd_42431_msk_1.map emd_42431_msk_1.map emd_42431_msk_2.map emd_42431_msk_2.map | 216 MB 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42431.cif.gz emd-42431.cif.gz | 7 KB | ||
| その他 |  emd_42431_additional_1.map.gz emd_42431_additional_1.map.gz emd_42431_half_map_1.map.gz emd_42431_half_map_1.map.gz emd_42431_half_map_2.map.gz emd_42431_half_map_2.map.gz | 106.9 MB 200.7 MB 200.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42431 http://ftp.pdbj.org/pub/emdb/structures/EMD-42431 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42431 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42431 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42431_validation.pdf.gz emd_42431_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42431_full_validation.pdf.gz emd_42431_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_42431_validation.xml.gz emd_42431_validation.xml.gz | 21.1 KB | 表示 | |
| CIF形式データ |  emd_42431_validation.cif.gz emd_42431_validation.cif.gz | 26.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42431 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42431 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42431 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42431 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42431.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42431.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.648 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42431_msk_1.map emd_42431_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_42431_msk_2.map emd_42431_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_42431_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_42431_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_42431_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Protein with a nanobody
| 全体 | 名称: Protein with a nanobody |
|---|---|
| 要素 |
|
-超分子 #1: Protein with a nanobody
| 超分子 | 名称: Protein with a nanobody / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 98.1 kDa/nm |
-分子 #1: Synaptic vesicle glycoprotein 2A
| 分子 | 名称: Synaptic vesicle glycoprotein 2A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 75.589188 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGYYRGEGTQ DEEEGGASSD ATEGHDEDDE IYEGEYQGIP RAESGGKGER MADGAPLAGV RGGLSDGEGP PGGRGEAQRR KEREELAQQ YEAILRECGH GRFQWTLYFV LGLALMADGV EVFVVGFVLP SAEKDMCLSD SNKGMLGLIV YLGMMVGAFL W GGLADRLG ...文字列: MGYYRGEGTQ DEEEGGASSD ATEGHDEDDE IYEGEYQGIP RAESGGKGER MADGAPLAGV RGGLSDGEGP PGGRGEAQRR KEREELAQQ YEAILRECGH GRFQWTLYFV LGLALMADGV EVFVVGFVLP SAEKDMCLSD SNKGMLGLIV YLGMMVGAFL W GGLADRLG RRQCLLISLS VNSVFAFFSS FVQGYGTFLF CRLLSGVGIG GSIPIVFSYF SEFLAQEKRG EHLSWLCMFW MI GGVYAAA MAWAIIPHYG WSFQMGSAYQ FHSWRVFVLV CAFPSVFAIG ALTTQPESPR FFLENGKHDE AWMVLKQVHD TNM RAKGHP ERVFSVTHIK TIHQEDELIE IQSDTGTWYQ RWGVRALSLG GQVWGNFLSC FGPEYRRITL MMMGVWFTMS FSYY GLTVW FPDMIRHLQA VDYASRTKVF PGERVEHVTF NFTLENQIHR GGQYFNDKFI GLRLKSVSFE DSLFEECYFE DVTSS NTFF RNCTFINTVF YNTDLFEYKF VNSRLINSTF LHNKEGCPLD VTGTGEGAYM VYFVSFLGTL AVLPGNIVSA LLMDKI GRL RMLAGSSVMS CVSCFFLSFG NSESAMIALL CLFGGVSIAS WNALDVLTVE LYPSDKRTTA FGFLNALCKL AAVLGIS IF TSFVGITKAA PILFASAALA LGSSLALKLP ETRGQVLQ UniProtKB: Synaptic vesicle glycoprotein 2A |
-分子 #2: Nanobody
| 分子 | 名称: Nanobody / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.469103 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVESGGG LVQPGGSLRL SCAASGSIFN MRVMGWYRQA PGEQRESVAS MASGDKTTYA DSVKGRFTIS RDNAKNTVAL QMNSLKPED TAVYYCHAVD LTRNGPRVYW GQGTQVTVSS AAAENLYFQG GSGHHHHHHH HHH |
-分子 #5: (4S)-1-{[(4S)-2-(methoxymethyl)-6-(trifluoromethyl)imidazo[2,1-b]...
| 分子 | 名称: (4S)-1-{[(4S)-2-(methoxymethyl)-6-(trifluoromethyl)imidazo[2,1-b][1,3,4]thiadiazol-5-yl]methyl}-4-(4,4,4-trifluorobutyl)pyrrolidin-2-one タイプ: ligand / ID: 5 / コピー数: 1 / 式: X49 |
|---|---|
| 分子量 | 理論値: 444.395 Da |
-分子 #6: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-分子 #7: 1,2-DIDECANOYL-SN-GLYCERO-3-[PHOSPHO-L-SERINE]
| 分子 | 名称: 1,2-DIDECANOYL-SN-GLYCERO-3-[PHOSPHO-L-SERINE] / タイプ: ligand / ID: 7 / コピー数: 1 / 式: PS1 |
|---|---|
| 分子量 | 理論値: 566.642 Da |
| Chemical component information |  ChemComp-PS1: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.38 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 77160 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)