+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4112 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Aichi virus 1: empty particle | |||||||||
 マップデータ マップデータ | Archie Virus 1: Empty particle | |||||||||
 試料 試料 | Aichi != Aichi virus 1 Aichi
| |||||||||
 キーワード キーワード | picornavirus / picornaviridae / Aichi virus 1 / empty particle / kobuvirus / 50 genome / release / human / pathogen / virus / RNA | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / symbiont-mediated suppression of host mRNA export from nucleus / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / protein complex oligomerization / monoatomic ion channel activity / symbiont-mediated suppression of host gene expression / RNA helicase activity / RNA helicase ...host cell Golgi membrane / symbiont-mediated suppression of host mRNA export from nucleus / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / protein complex oligomerization / monoatomic ion channel activity / symbiont-mediated suppression of host gene expression / RNA helicase activity / RNA helicase / symbiont entry into host cell / RNA-directed RNA polymerase / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / structural molecule activity / ATP hydrolysis activity / proteolysis / RNA binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Aichi virus (ウイルス) / Aichi virus (ウイルス) /  Aichi virus 1 (ウイルス) Aichi virus 1 (ウイルス) | |||||||||
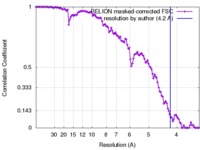
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Sabin C / Fuzik T | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2016 ジャーナル: J Virol / 年: 2016タイトル: Structure of Aichi Virus 1 and Its Empty Particle: Clues to Kobuvirus Genome Release Mechanism. 著者: Charles Sabin / Tibor Füzik / Karel Škubník / Lenka Pálková / A Michael Lindberg / Pavel Plevka /   要旨: (AiV-1) is a human pathogen from the genus of the family. Worldwide, 80 to 95% of adults have antibodies against the virus. AiV-1 infections are associated with nausea, gastroenteritis, and fever. ... (AiV-1) is a human pathogen from the genus of the family. Worldwide, 80 to 95% of adults have antibodies against the virus. AiV-1 infections are associated with nausea, gastroenteritis, and fever. Unlike most picornaviruses, kobuvirus capsids are composed of only three types of subunits: VP0, VP1, and VP3. We present here the structure of the AiV-1 virion determined to a resolution of 2.1 Å using X-ray crystallography. The surface loop puff of VP0 and knob of VP3 in AiV-1 are shorter than those in other picornaviruses. Instead, the 42-residue BC loop of VP0 forms the most prominent surface feature of the AiV-1 virion. We determined the structure of AiV-1 empty particle to a resolution of 4.2 Å using cryo-electron microscopy. The empty capsids are expanded relative to the native virus. The N-terminal arms of capsid proteins VP0, which mediate contacts between the pentamers of capsid protein protomers in the native AiV-1 virion, are disordered in the empty capsid. Nevertheless, the empty particles are stable, at least , and do not contain pores that might serve as channels for genome release. Therefore, extensive and probably reversible local reorganization of AiV-1 capsid is required for its genome release. Aichi virus 1 (AiV-1) is a human pathogen that can cause diarrhea, abdominal pain, nausea, vomiting, and fever. AiV-1 is identified in environmental screening studies with higher frequency and greater abundance than other human enteric viruses. Accordingly, 80 to 95% of adults worldwide have suffered from AiV-1 infections. We determined the structure of the AiV-1 virion. Based on the structure, we show that antiviral compounds that were developed against related enteroviruses are unlikely to be effective against AiV-1. The surface of the AiV-1 virion has a unique topology distinct from other related viruses from the family. We also determined that AiV-1 capsids form compact shells even after genome release. Therefore, AiV-1 genome release requires large localized and probably reversible reorganization of the capsid. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4112.map.gz emd_4112.map.gz | 8.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4112-v30.xml emd-4112-v30.xml emd-4112.xml emd-4112.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4112_fsc.xml emd_4112_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4112.png emd_4112.png | 170.7 KB | ||
| Filedesc metadata |  emd-4112.cif.gz emd-4112.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4112 http://ftp.pdbj.org/pub/emdb/structures/EMD-4112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4112 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4112 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4112_validation.pdf.gz emd_4112_validation.pdf.gz | 291.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4112_full_validation.pdf.gz emd_4112_full_validation.pdf.gz | 290.9 KB | 表示 | |
| XML形式データ |  emd_4112_validation.xml.gz emd_4112_validation.xml.gz | 12.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4112 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4112 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4112 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4112.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4112.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Archie Virus 1: Empty particle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.73 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Aichi
| 全体 | 名称: Aichi |
|---|---|
| 要素 |
|
-超分子 #1: Aichi virus 1
| 超分子 | 名称: Aichi virus 1 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 1313215 / 生物種: Aichi virus 1 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Aichi virus (ウイルス) Aichi virus (ウイルス) |
| 分子量 | 理論値: 27.194688 KDa |
| 組換発現 | 生物種:  Chlorocebus sabaeus (オナガザル) Chlorocebus sabaeus (オナガザル) |
| 配列 | 文字列: TLTEDLDAPQ DTGNIENGAA DNSPQPRTTF DYTGNPLPPD TKLENFFSFY RLLPMGGSGA PSLSFPADEG TIIPLNPINW LKGADVSGI AAMLSCFTYI AADLRITLRF SNPNDNPATM LVAFAPPGAT IPLKPTRQML SNFYMAEVPV SAATSTMVSF S IPYTSPLS ...文字列: TLTEDLDAPQ DTGNIENGAA DNSPQPRTTF DYTGNPLPPD TKLENFFSFY RLLPMGGSGA PSLSFPADEG TIIPLNPINW LKGADVSGI AAMLSCFTYI AADLRITLRF SNPNDNPATM LVAFAPPGAT IPLKPTRQML SNFYMAEVPV SAATSTMVSF S IPYTSPLS AIPTSYFGWE DWSGTNFGQL SSGSWGNLML IPSLSVDSAI PFDFQLSCWV AFGNFKAWVP RPPPPLPPLP TP AANAERT VAVIKQ UniProtKB: Genome polyprotein |
-分子 #2: VP0
| 分子 | 名称: VP0 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Aichi virus (ウイルス) Aichi virus (ウイルス) |
| 分子量 | 理論値: 38.950758 KDa |
| 組換発現 | 生物種:  Chlorocebus sabaeus (オナガザル) Chlorocebus sabaeus (オナガザル) |
| 配列 | 文字列: GNSVTNIYGN GNNVTTDVGA NGWAPTVSTG LGDGPVSASA DSLPGRSGGA SSEKTHTVSG SSNKVGSRFS KWWEPAAARA SESATDSAI EGIDAAGKAA SKAITRKLDR PAAPSSTANP QPSLIALNPS ATQSGNASIL TGSTAPSLLA YPTATPVPLP N PDEPSQPG ...文字列: GNSVTNIYGN GNNVTTDVGA NGWAPTVSTG LGDGPVSASA DSLPGRSGGA SSEKTHTVSG SSNKVGSRFS KWWEPAAARA SESATDSAI EGIDAAGKAA SKAITRKLDR PAAPSSTANP QPSLIALNPS ATQSGNASIL TGSTAPSLLA YPTATPVPLP N PDEPSQPG PSGDRTWLLD TVTWSQEFTR GWNIAGSNGM QWTGLESLIF PVSTDTNWTS TSSPTAYPLP FSFVRAYPDS SW AAMYNTH SMWNCGWRVQ VTVNGSQFHA GALILYMVPE ATTHAIQTAR DNAGFVFPYV ILNLYESNTA TIEVPYISPT PNT SSGLHA PWTFYLQVLS PLNPPPSLPT SLSCSIYVTP VDSSFHGLRY LAPQ UniProtKB: Genome polyprotein |
-分子 #3: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 3 / 詳細: VP3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Aichi virus (ウイルス) Aichi virus (ウイルス) |
| 分子量 | 理論値: 24.082244 KDa |
| 配列 | 文字列: HWKTRAVPGA GTFGSAVAGQ ELPLCGVRAY YPPNAYIPAQ VRDWLEFAHR PGLMATVPWT MADEPAERLG IFPVSPSAIA GTGAPISYV ISLFSQWRGE LAAHLLFTGS AQHYGRLVVC YTPAAPQPPS TMQEAMRGTY TVWDVNAAST LEFTIPFISN S YWKTVDVN ...文字列: HWKTRAVPGA GTFGSAVAGQ ELPLCGVRAY YPPNAYIPAQ VRDWLEFAHR PGLMATVPWT MADEPAERLG IFPVSPSAIA GTGAPISYV ISLFSQWRGE LAAHLLFTGS AQHYGRLVVC YTPAAPQPPS TMQEAMRGTY TVWDVNAAST LEFTIPFISN S YWKTVDVN NPDALLSTTG YVSIWVQNPL VGPHTAPASA LVQAFISAGE SFNVRLMQNP ALTSQ UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー