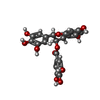
| 登録情報 | データベース: PDB / ID: 3oob
|
|---|
| タイトル | Structural and functional insights of directly targeting Pin1 by Epigallocatechin-3-gallate |
|---|
 要素 要素 | Peptidyl-prolyl cis-trans isomerase NIMA-interacting 1 |
|---|
 キーワード キーワード | ISOMERASE / WW domain-like peptidyl prolyl isomerase / phosphorylation-dependent cis/trans isomerase / proteins with pSer/pThr-Pro motif |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
cis-trans isomerase activity / phosphothreonine residue binding / negative regulation of cell motility / ubiquitin ligase activator activity / regulation of protein localization to nucleus / GTPase activating protein binding / mitogen-activated protein kinase kinase binding / protein peptidyl-prolyl isomerization / : / regulation of mitotic nuclear division ...cis-trans isomerase activity / phosphothreonine residue binding / negative regulation of cell motility / ubiquitin ligase activator activity / regulation of protein localization to nucleus / GTPase activating protein binding / mitogen-activated protein kinase kinase binding / protein peptidyl-prolyl isomerization / : / regulation of mitotic nuclear division / negative regulation of SMAD protein signal transduction / PI5P Regulates TP53 Acetylation / negative regulation of amyloid-beta formation / cytoskeletal motor activity / RHO GTPases Activate NADPH Oxidases / phosphoserine residue binding / postsynaptic cytosol / Rho protein signal transduction / negative regulation of protein binding / regulation of cytokinesis / peptidylprolyl isomerase / peptidyl-prolyl cis-trans isomerase activity / Negative regulators of DDX58/IFIH1 signaling / negative regulation of transforming growth factor beta receptor signaling pathway / phosphoprotein binding / synapse organization / beta-catenin binding / negative regulation of protein catabolic process / regulation of protein stability / negative regulation of ERK1 and ERK2 cascade / ISG15 antiviral mechanism / tau protein binding / neuron differentiation / positive regulation of protein phosphorylation / positive regulation of canonical Wnt signaling pathway / regulation of gene expression / midbody / cellular response to hypoxia / Regulation of TP53 Activity through Phosphorylation / response to hypoxia / protein stabilization / nuclear speck / ciliary basal body / glutamatergic synapse / positive regulation of transcription by RNA polymerase II / nucleoplasm / nucleus / cytosol / cytoplasm類似検索 - 分子機能 Ubiquitin Ligase Nedd4; Chain: W; - #10 / Ubiquitin Ligase Nedd4; Chain: W; / : / Peptidyl-prolyl cis-trans isomerase, PpiC-type, conserved site / PpiC-type peptidyl-prolyl cis-trans isomerase signature. / PPIC-type PPIASE domain / PpiC-type peptidyl-prolyl cis-trans isomerase family profile. / Peptidyl-prolyl cis-trans isomerase, PpiC-type / Chitinase A; domain 3 - #40 / Chitinase A; domain 3 ...Ubiquitin Ligase Nedd4; Chain: W; - #10 / Ubiquitin Ligase Nedd4; Chain: W; / : / Peptidyl-prolyl cis-trans isomerase, PpiC-type, conserved site / PpiC-type peptidyl-prolyl cis-trans isomerase signature. / PPIC-type PPIASE domain / PpiC-type peptidyl-prolyl cis-trans isomerase family profile. / Peptidyl-prolyl cis-trans isomerase, PpiC-type / Chitinase A; domain 3 - #40 / Chitinase A; domain 3 / WW domain / WW/rsp5/WWP domain signature. / WW domain superfamily / WW/rsp5/WWP domain profile. / Domain with 2 conserved Trp (W) residues / WW domain / Single Sheet / Peptidyl-prolyl cis-trans isomerase domain superfamily / Roll / Mainly Beta / Alpha Beta類似検索 - ドメイン・相同性 Chem-KDH / Peptidyl-prolyl cis-trans isomerase NIMA-interacting 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.89 Å 分子置換 / 解像度: 1.89 Å |
|---|
 データ登録者 データ登録者 | Urusova, D.V. / Shim, J.-H. / Kim, D.-J. / Jung, S.K. / Zykova, T.A. / Bode, A.M. / Dong, Z. |
|---|
 引用 引用 |  ジャーナル: Cancer Prev Res (Phila) / 年: 2011 ジャーナル: Cancer Prev Res (Phila) / 年: 2011
タイトル: Epigallocatechin-gallate suppresses tumorigenesis by directly targeting Pin1.
著者: Urusova, D.V. / Shim, J.H. / Kim, D.J. / Jung, S.K. / Zykova, T.A. / Carper, A. / Bode, A.M. / Dong, Z. |
|---|
| 履歴 | | 登録 | 2010年8月30日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2011年8月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年8月24日 | Group: Other |
|---|
| 改定 1.2 | 2011年11月30日 | Group: Database references |
|---|
| 改定 1.3 | 2017年11月8日 | Group: Refinement description / カテゴリ: software |
|---|
| 改定 1.4 | 2024年2月21日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / struct_ref_seq_dif / struct_site
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.89 Å
分子置換 / 解像度: 1.89 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cancer Prev Res (Phila) / 年: 2011
ジャーナル: Cancer Prev Res (Phila) / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3oob.cif.gz
3oob.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3oob.ent.gz
pdb3oob.ent.gz PDB形式
PDB形式 3oob.json.gz
3oob.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/oo/3oob
https://data.pdbj.org/pub/pdb/validation_reports/oo/3oob ftp://data.pdbj.org/pub/pdb/validation_reports/oo/3oob
ftp://data.pdbj.org/pub/pdb/validation_reports/oo/3oob リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: PIN1 / プラスミド: pET28a(+) / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PIN1 / プラスミド: pET28a(+) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 23-ID-D / 波長: 0.99188 Å
/ ビームライン: 23-ID-D / 波長: 0.99188 Å 解析
解析 分子置換 / 解像度: 1.89→28.1 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.949 / WRfactor Rfree: 0.2367 / WRfactor Rwork: 0.1943 / Occupancy max: 1 / Occupancy min: 0.1 / FOM work R set: 0.8449 / SU B: 3.041 / SU ML: 0.091 / SU R Cruickshank DPI: 0.1291 / SU Rfree: 0.1299 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.13 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
分子置換 / 解像度: 1.89→28.1 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.949 / WRfactor Rfree: 0.2367 / WRfactor Rwork: 0.1943 / Occupancy max: 1 / Occupancy min: 0.1 / FOM work R set: 0.8449 / SU B: 3.041 / SU ML: 0.091 / SU R Cruickshank DPI: 0.1291 / SU Rfree: 0.1299 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.13 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj