+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3399 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the core NuRD complex (MTA1:HDAC1:RBBP4) | |||||||||
 マップデータ マップデータ | Structure of the core NuRD complex: MTA1:HDAC1:RBBP4 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Transcription / HDAC1 / MTA1 / RBBP4 / Chromatin / Histone Deacetylase / Metastasis associated protein / Histone binding protein | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
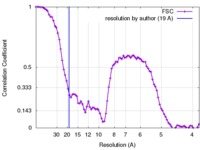
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 19.0 Å | |||||||||
 データ登録者 データ登録者 | Millard CJ / Saleh A / Morris K / Fairall L / Smith CJ / Schwabe JWR | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2016 ジャーナル: Elife / 年: 2016タイトル: The structure of the core NuRD repression complex provides insights into its interaction with chromatin. 著者: Christopher J Millard / Niranjan Varma / Almutasem Saleh / Kyle Morris / Peter J Watson / Andrew R Bottrill / Louise Fairall / Corinne J Smith / John W R Schwabe /  要旨: The NuRD complex is a multi-protein transcriptional corepressor that couples histone deacetylase and ATP-dependent chromatin remodelling activities. The complex regulates the higher-order structure ...The NuRD complex is a multi-protein transcriptional corepressor that couples histone deacetylase and ATP-dependent chromatin remodelling activities. The complex regulates the higher-order structure of chromatin, and has important roles in the regulation of gene expression, DNA damage repair and cell differentiation. HDACs 1 and 2 are recruited by the MTA1 corepressor to form the catalytic core of the complex. The histone chaperone protein RBBP4, has previously been shown to bind to the carboxy-terminal tail of MTA1. We show that MTA1 recruits a second copy of RBBP4. The crystal structure reveals an extensive interface between MTA1 and RBBP4. An EM structure, supported by SAXS and crosslinking, reveals the architecture of the dimeric HDAC1:MTA1:RBBP4 assembly which forms the core of the NuRD complex. We find evidence that in this complex RBBP4 mediates interaction with histone H3 tails, but not histone H4, suggesting a mechanism for recruitment of the NuRD complex to chromatin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3399.map.gz emd_3399.map.gz | 3.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3399-v30.xml emd-3399-v30.xml emd-3399.xml emd-3399.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3399_fsc.xml emd_3399_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3399.tif emd_3399.tif | 122.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3399 http://ftp.pdbj.org/pub/emdb/structures/EMD-3399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3399 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3399 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3399.map.gz / 形式: CCP4 / 大きさ: 41.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3399.map.gz / 形式: CCP4 / 大きさ: 41.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the core NuRD complex: MTA1:HDAC1:RBBP4 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.921 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Core NuRD complex (MTA1:HDAC1:RBBP4)
| 全体 | 名称: Core NuRD complex (MTA1:HDAC1:RBBP4) |
|---|---|
| 要素 |
|
-超分子 #1000: Core NuRD complex (MTA1:HDAC1:RBBP4)
| 超分子 | 名称: Core NuRD complex (MTA1:HDAC1:RBBP4) / タイプ: sample / ID: 1000 / 詳細: Dimer / 集合状態: 2 / Number unique components: 3 |
|---|---|
| 分子量 | 実験値: 300 KDa / 理論値: 300 KDa / 手法: SEC-MALS |
-分子 #1: MTA1
| 分子 | 名称: MTA1 / タイプ: ligand / ID: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 組換発現 | 生物種: HEK293F / 組換プラスミド: pcDNA3 |
-分子 #2: HDAC1
| 分子 | 名称: HDAC1 / タイプ: ligand / ID: 2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 組換発現 | 生物種: HEK293F / 組換プラスミド: pcDNA3 |
-分子 #3: RBBP4
| 分子 | 名称: RBBP4 / タイプ: ligand / ID: 3 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 組換発現 | 生物種: HEK293F / 組換プラスミド: pcDNA3 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20 mM Tris/HCl (pH 7.5), 40 mM NaCl |
| 染色 | タイプ: NEGATIVE / 詳細: 2% uranyl acetate for 1 min |
| グリッド | 詳細: Prepared by glow discharging carbon coated copper 300 mesh grids (agar scientific) at 10 mA for 30 seconds |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2010F |
|---|---|
| 日付 | 2015年7月22日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 308 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: JEOL |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Fit in map (Chimera) |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Fit in map (Chimera) |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)