+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30131 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the encapsulated DyP-type peroxidase from Mycobacterium smegmatis | |||||||||||||||
 マップデータ マップデータ | Post-processed Cryo-EM map generated by RELION | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  Cargo protein (貨物) / Cargo protein (貨物) /  Dodecamer / Heme-containing enzyme / Dodecamer / Heme-containing enzyme /  Oxidoreductase (酸化還元酵素) Oxidoreductase (酸化還元酵素) | |||||||||||||||
| 機能・相同性 |  Dyp-type peroxidase, N-terminal / Dyp-type peroxidase, N-terminal /  DyP-type peroxidase family. / DyP-type peroxidase family. /  Dyp-type peroxidase / Dyp-type peroxidase /  ペルオキシダーゼ / Dimeric alpha-beta barrel / ペルオキシダーゼ / Dimeric alpha-beta barrel /  peroxidase activity / peroxidase activity /  heme binding / heme binding /  Dyp-type peroxidase Dyp-type peroxidase 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) | |||||||||||||||
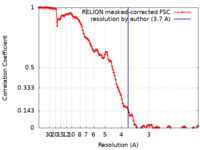
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Tang YT / Mu A / Gong HR / Wang Q / Rao ZH | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Cryo-EM structure of DyP-loaded encapsulin. 著者: Yanting Tang / An Mu / Yuying Zhang / Shan Zhou / Weiwei Wang / Yuezheng Lai / Xiaoting Zhou / Fengjiang Liu / Xiuna Yang / Hongri Gong / Quan Wang / Zihe Rao /  要旨: Encapsulins containing dye-decolorizing peroxidase (DyP)-type peroxidases are ubiquitous among prokaryotes, protecting cells against oxidative stress. However, little is known about how they interact ...Encapsulins containing dye-decolorizing peroxidase (DyP)-type peroxidases are ubiquitous among prokaryotes, protecting cells against oxidative stress. However, little is known about how they interact and function. Here, we have isolated a native cargo-packaging encapsulin from and determined its complete high-resolution structure by cryogenic electron microscopy (cryo-EM). This encapsulin comprises an icosahedral shell and a dodecameric DyP cargo. The dodecameric DyP consists of two hexamers with a twofold axis of symmetry and stretches across the interior of the encapsulin. Our results reveal that the encapsulin shell plays a role in stabilizing the dodecameric DyP. Furthermore, we have proposed a potential mechanism for removing the hydrogen peroxide based on the structural features. Our study also suggests that the DyP is the primary cargo protein of mycobacterial encapsulins and is a potential target for antituberculosis drug discovery. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30131.map.gz emd_30131.map.gz | 164.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30131-v30.xml emd-30131-v30.xml emd-30131.xml emd-30131.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30131_fsc.xml emd_30131_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30131.png emd_30131.png | 181.8 KB | ||
| マスクデータ |  emd_30131_msk_1.map emd_30131_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-30131.cif.gz emd-30131.cif.gz | 5.9 KB | ||
| その他 |  emd_30131_half_map_1.map.gz emd_30131_half_map_1.map.gz emd_30131_half_map_2.map.gz emd_30131_half_map_2.map.gz | 138.8 MB 139 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30131 http://ftp.pdbj.org/pub/emdb/structures/EMD-30131 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30131 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30131 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30131.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30131.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed Cryo-EM map generated by RELION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
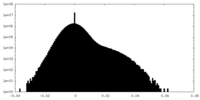
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_30131_msk_1.map emd_30131_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
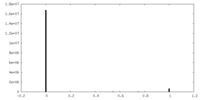
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Second one of the half map after 3D-refinement generated by RELION
| ファイル | emd_30131_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second one of the half map after 3D-refinement generated by RELION | ||||||||||||
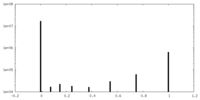
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: First one of the half map after 3D-refinement generated by RELION
| ファイル | emd_30131_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | First one of the half map after 3D-refinement generated by RELION | ||||||||||||
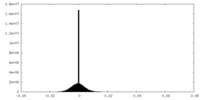
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Encapsulin from Mycobacterium smegmatis
| 全体 | 名称: Encapsulin from Mycobacterium smegmatis |
|---|---|
| 要素 |
|
-超分子 #1: Encapsulin from Mycobacterium smegmatis
| 超分子 | 名称: Encapsulin from Mycobacterium smegmatis / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
-分子 #1: Dyp-type peroxidase
| 分子 | 名称: Dyp-type peroxidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号:  ペルオキシダーゼ ペルオキシダーゼ |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 分子量 | 理論値: 37.25234 KDa |
| 配列 | 文字列: MPAPQPQPVL APLTPAAVFL VATIDEGQEA TVYDALPDIS GLVRAIGFRD PAKRLSAITS IGSDAWDRLF SGPRPAELHP FREIDGGRH HAPATPGDLL FHLRAESMDV CFELATKLVE AMSGAITIVD ETHGFRFFDN RDLMGFVDGT ENPDGNLAVV A TQIGDEDP ...文字列: MPAPQPQPVL APLTPAAVFL VATIDEGQEA TVYDALPDIS GLVRAIGFRD PAKRLSAITS IGSDAWDRLF SGPRPAELHP FREIDGGRH HAPATPGDLL FHLRAESMDV CFELATKLVE AMSGAITIVD ETHGFRFFDN RDLMGFVDGT ENPDGNLAVV A TQIGDEDP DFAGGCYVHV QKYLHDMASW NSLSVEEQER VIGRTKLDDI ELDDDVKPAN SHVALNVIED EDGNELKIIR HN MPFGEIG KGEFGTYYIG YSRTPSVTER MLDNMFIGDP PGNTDRILDF STAITGGLFF TPTVDFLDDP PPLPSEDDRA EPA SAPSAD PVHTDGSLGI GSLKGTR UniProtKB:  Dyp-type peroxidase Dyp-type peroxidase |
-分子 #2: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 平均露光時間: 6.4 sec. / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X