+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2cbr | ||||||
|---|---|---|---|---|---|---|---|
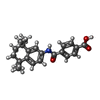
| タイトル | CELLULAR RETINOIC ACID BINDING PROTEIN I IN COMPLEX WITH A RETINOBENZOIC ACID (AM80) | ||||||
 要素 要素 | PROTEIN (CRABP-I) | ||||||
 キーワード キーワード | TRANSPORT PROTEIN / RETINOIC-ACID TRANSPORT | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RA biosynthesis pathway / retinal binding / retinoic acid binding / retinol binding / fatty acid transport / fatty acid binding / protein-containing complex / identical protein binding / nucleus / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Chaudhuri, B. / Kleywegt, G.J. / Bergfors, T. / Jones, T.A. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1999 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1999タイトル: Structures of cellular retinoic acid binding proteins I and II in complex with synthetic retinoids. 著者: Chaudhuri, B.N. / Kleywegt, G.J. / Broutin-L'Hermite, I. / Bergfors, T. / Senn, H. / Le Motte, P. / Partouche, O. / Jones, T.A. #1:  ジャーナル: Structure / 年: 1994 ジャーナル: Structure / 年: 1994タイトル: Crystal structures of cellular retinoic acid binding proteins I and II in complex with all-trans-retinoic acid and a synthetic retinoid. 著者: Kleywegt, G.J. / Bergfors, T. / Senn, H. / Le Motte, P. / Gsell, B. / Shudo, K. / Jones, T.A. #2: ジャーナル: Adv.Protein Chem. / 年: 1994 タイトル: Lipid-binding proteins: a family of fatty acid and retinoid transport proteins. 著者: Banaszak, L. / Winter, N. / Xu, Z. / Bernlohr, D.A. / Cowan, S. / Jones, T.A. #3: ジャーナル: Acta Crystallogr.,Sect.D / 年: 1994 タイトル: Crystallization and preliminary X-ray analysis of recombinant bovine cellular retinoic acid-binding protein. 著者: Bergfors, T. / Kleywegt, G.J. / Jones, T.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2cbr.cif.gz 2cbr.cif.gz | 39.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2cbr.ent.gz pdb2cbr.ent.gz | 27 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2cbr.json.gz 2cbr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2cbr_validation.pdf.gz 2cbr_validation.pdf.gz | 667.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2cbr_full_validation.pdf.gz 2cbr_full_validation.pdf.gz | 670 KB | 表示 | |
| XML形式データ |  2cbr_validation.xml.gz 2cbr_validation.xml.gz | 8.1 KB | 表示 | |
| CIF形式データ |  2cbr_validation.cif.gz 2cbr_validation.cif.gz | 9.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cb/2cbr https://data.pdbj.org/pub/pdb/validation_reports/cb/2cbr ftp://data.pdbj.org/pub/pdb/validation_reports/cb/2cbr ftp://data.pdbj.org/pub/pdb/validation_reports/cb/2cbr | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: generate Matrix: (0.231781, -0.961217, 0.149465), ベクター: |
- 要素
要素
| #1: タンパク質 | 分子量: 15480.392 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  解説: MUS MUSCULUS (IDENTICAL WITH BOVINE PROTEIN) RECOMBINANT GENE 遺伝子: MOUSE / プラスミド: PT7-1-3 / 生物種 (発現宿主): Escherichia coli / 細胞内の位置 (発現宿主): CYTOPLASM / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-A80 / |
| #3: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶化 | pH: 8 詳細: IN 30% PEG4000, 0.2M LI2SO4 AND 0.1 M TRIS-HCL, PH 8.0 | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS 温度: 277 K / 手法: 蒸気拡散法, ハンギングドロップ法詳細: drop contained equal volume of protein and reservoir solution | ||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 277 K |
|---|---|
| 放射光源 | 波長: 1.5418 |
| 検出器 | タイプ: RIGAKU / 検出器: IMAGE PLATE |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→38.3 Å / Num. obs: 10128 / % possible obs: 97.6 % / 冗長度: 3.7 % / Biso Wilson estimate: 24 Å2 / Rmerge(I) obs: 0.086 / Net I/σ(I): 20.2 |
| 反射 シェル | 解像度: 2.8→2.95 Å / 冗長度: 3.8 % / Rmerge(I) obs: 0.26 / Mean I/σ(I) obs: 5.2 / % possible all: 95.7 |
| 反射 | *PLUS 最低解像度: 38 Å |
| 反射 シェル | *PLUS % possible obs: 95.7 % |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1CBR 解像度: 2.8→30 Å / Rfactor Rfree error: 0.009 / Data cutoff high rms absF: 1543984.01 / Isotropic thermal model: GROUP / 交差検証法: THROUGHOUT / σ(F): 0 詳細: BULK SOLVENT MODEL USED THE STRUCTURE WAS REFINED WITH NCS CONSTRAINTS IN CNS (I.E., THE TWO MOLECULES ARE IDENTICAL).
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 37.7 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→30 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: CONSTR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.98 Å / Rfactor Rfree error: 0.032 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / バージョン: 0.3 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Num. reflection obs: 9305 / Rfactor Rfree: 0.266 / Rfactor Rwork: 0.228 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj