+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23931 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human HUWE1 (d169-189 variant) | ||||||||||||||||||
 マップデータ マップデータ | main map from Relion PostProcess | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of mitochondrial fusion / positive regulation of mitophagy in response to mitochondrial depolarization / histone ubiquitin ligase activity / HECT-type E3 ubiquitin transferase / positive regulation of protein targeting to mitochondrion / Golgi organization / protein monoubiquitination / positive regulation of protein ubiquitination / circadian regulation of gene expression /  塩基除去修復 ...negative regulation of mitochondrial fusion / positive regulation of mitophagy in response to mitochondrial depolarization / histone ubiquitin ligase activity / HECT-type E3 ubiquitin transferase / positive regulation of protein targeting to mitochondrion / Golgi organization / protein monoubiquitination / positive regulation of protein ubiquitination / circadian regulation of gene expression / 塩基除去修復 ...negative regulation of mitochondrial fusion / positive regulation of mitophagy in response to mitochondrial depolarization / histone ubiquitin ligase activity / HECT-type E3 ubiquitin transferase / positive regulation of protein targeting to mitochondrion / Golgi organization / protein monoubiquitination / positive regulation of protein ubiquitination / circadian regulation of gene expression /  塩基除去修復 / protein polyubiquitination / ubiquitin-protein transferase activity / 塩基除去修復 / protein polyubiquitination / ubiquitin-protein transferase activity /  ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / ubiquitin-dependent protein catabolic process / secretory granule lumen / ficolin-1-rich granule lumen / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / ubiquitin-dependent protein catabolic process / secretory granule lumen / ficolin-1-rich granule lumen /  membrane fusion / membrane fusion /  細胞分化 / 細胞分化 /  ゴルジ体 / Neutrophil degranulation / ゴルジ体 / Neutrophil degranulation /  ミトコンドリア / ミトコンドリア /  DNA binding / DNA binding /  RNA binding / extracellular exosome / extracellular region / RNA binding / extracellular exosome / extracellular region /  核質 / 核質 /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
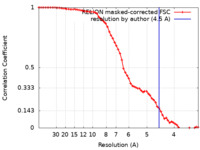
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.5 Å クライオ電子顕微鏡法 / 解像度: 4.5 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Hunkeler M / Fischer ES | ||||||||||||||||||
| 資金援助 |  米国, 米国,  スイス, 5件 スイス, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Solenoid architecture of HUWE1 contributes to ligase activity and substrate recognition. 著者: Moritz Hunkeler / Cyrus Y Jin / Michelle W Ma / Julie K Monda / Daan Overwijn / Eric J Bennett / Eric S Fischer /  要旨: HECT ubiquitin ligases play essential roles in metazoan development and physiology. The HECT ligase HUWE1 is central to the cellular stress response by mediating degradation of key death or survival ...HECT ubiquitin ligases play essential roles in metazoan development and physiology. The HECT ligase HUWE1 is central to the cellular stress response by mediating degradation of key death or survival factors, including Mcl1, p53, DDIT4, and Myc. Although mutations in HUWE1 and related HECT ligases are widely implicated in human disease, our molecular understanding remains limited. Here we present a comprehensive investigation of full-length HUWE1, deepening our understanding of this class of enzymes. The N-terminal ∼3,900 amino acids of HUWE1 are indispensable for proper ligase function, and our cryo-EM structures of HUWE1 offer a complete molecular picture of this large HECT ubiquitin ligase. HUWE1 forms an alpha solenoid-shaped assembly with a central pore decorated with protein interaction modules. Structures of HUWE1 variants linked to neurodevelopmental disorders as well as of HUWE1 bound to a model substrate link the functions of this essential enzyme to its three-dimensional organization. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23931.map.gz emd_23931.map.gz | 2.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23931-v30.xml emd-23931-v30.xml emd-23931.xml emd-23931.xml | 29.5 KB 29.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23931_fsc.xml emd_23931_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23931.png emd_23931.png | 42.6 KB | ||
| マスクデータ |  emd_23931_msk_1.map emd_23931_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23931_additional_1.map.gz emd_23931_additional_1.map.gz emd_23931_additional_2.map.gz emd_23931_additional_2.map.gz emd_23931_half_map_1.map.gz emd_23931_half_map_1.map.gz emd_23931_half_map_2.map.gz emd_23931_half_map_2.map.gz | 13.9 MB 110 MB 17 MB 17 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23931 http://ftp.pdbj.org/pub/emdb/structures/EMD-23931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23931 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23931.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23931.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map from Relion PostProcess | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23931_msk_1.map emd_23931_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: filtered map from Relion locres
| ファイル | emd_23931_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | filtered map from Relion locres | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: main map postprocessed with deepEMhancer (wideTarget)
| ファイル | emd_23931_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map postprocessed with deepEMhancer (wideTarget) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2 from Relion refine
| ファイル | emd_23931_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 from Relion refine | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2 from Relion refine
| ファイル | emd_23931_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 from Relion refine | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E3 ubiquitin-protein ligase HUWE1 (d169-189 variant)
| 全体 | 名称: E3 ubiquitin-protein ligase HUWE1 (d169-189 variant) |
|---|---|
| 要素 |
|
-超分子 #1: E3 ubiquitin-protein ligase HUWE1 (d169-189 variant)
| 超分子 | 名称: E3 ubiquitin-protein ligase HUWE1 (d169-189 variant) タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: patient mutation variant d169-189 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 480 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換株: expi293 / 組換プラスミド: pDEST Homo sapiens (ヒト) / 組換株: expi293 / 組換プラスミド: pDEST |
-分子 #1: E3 ubiquitin-protein ligase HUWE1
| 分子 | 名称: E3 ubiquitin-protein ligase HUWE1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: HECT-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDDDDKL AAANSSIDLI STSLYKKAGF KGTNSVDMKV DRTKLKKTPT EAPADCRALI DKLKVCNDEQ LLLELQQIKT WNIGKCELYH WVDLLDRFDG ILADAGQTVE NMSWMLVCDR PEREQLKMLL LAVLNFTALL IEYSFSRHLY SSIEHLTTLL ASSDMQVVLA ...文字列: MDYKDDDDKL AAANSSIDLI STSLYKKAGF KGTNSVDMKV DRTKLKKTPT EAPADCRALI DKLKVCNDEQ LLLELQQIKT WNIGKCELYH WVDLLDRFDG ILADAGQTVE NMSWMLVCDR PEREQLKMLL LAVLNFTALL IEYSFSRHLY SSIEHLTTLL ASSDMQVVLA VLNLLYVFSK RSNYITRLGS DKRTPLLTRL QHLAEKYPPS ATTLHFEFYA DPGAEVKIEK RTTSNTLHYI HIEQLDKISE SPSEIMESLT KMYSIPKDKQ MLLFTHIRLA HGFSNHRKRL QAVQARLHAI SILVYSNALQ ESANSILYNG LIEELVDVLQ ITDKQLMEIK AASLRTLTSI VHLERTPKLS SIIDCTGTAS YHGFLPVLVR NCIQAMIDPS MDPYPHQFAT ALFSFLYHLA SYDAGGEALV SCGMMEALLK VIKFLGDEQD QITFVTRAVR VVDLITNLDM AAFQSHSGLS IFIYRLEHEV DLCRKECPFV IKPKIQRPNT TQEGEEMETD MDGVQCIPQR AALLKSMLNF LKKAIQDPAF SDGIRHVMDG SLPTSLKHII SNAEYYGPSL FLLATEVVTV FVFQEPSLLS SLQDNGLTDV MLHALLIKDV PATREVLGSL PNVFSALCLN ARGLQSFVQC QPFERLFKVL LSPDYLPAMR RRRSSDPLGD TASNLGSAVD ELMRHQPTLK TDATTAIIKL LEEICNLGRD PKYICQKPSI QKADGTATAP PPRSNHAAEE ASSEDEEEEE VQAMQSFNST QQNETEPNQQ VVGTEERIPI PLMDYILNVM KFVESILSNN TTDDHCQEFV NQKGLLPLVT ILGLPNLPID FPTSAACQAV AGVCKSILTL SHEPKVLQEG LLQLDSILSS LEPLHRPIES PGGSVLLREL ACAGNVADAT LSAQATPLLH ALTAAHAYIM MFVHTCRVGQ SEIRSISVNQ WGSQLGLSVL SKLSQLYCSL VWESTVLLSL CTPNSLPSGC EFGQADMQKL VPKDEKAGTT QGGKRSDGEQ DGAAGSMDAS TQGLLEGIGL DGDTLAPMET DEPTASDSKG KSKITPAMAA RIKQIKPLLS ASSRLGRALA ELFGLLVKLC VGSPVRQRRS HHAASTTTAP TPAARSTASA LTKLLTKGLS WQPPPYTPTP RFRLTFFICS VGFTSPMLFD ERKYPYHLML QKFLCSGGHN ALFETFNWAL SMGGKVPVSE GLEHSDLPDG TGEFLDAWLM LVEKMVNPTT VLESPHSLPA KLPGGVQNFP QFSALRFLVV TQKAAFTCIK NLWNRKPLKV YGGRMAESML AILCHILRGE PVIRERLSKE KEGSRGEEDT GQEEGGSRRE PQVNQQQLQQ LMDMGFTREH AMEALLNTST MEQATEYLLT HPPPIMGGVV RDLSMSEEDQ MMRAIAMSLG QDIPMDQRAE SPEEVACRKE EEERKAREKQ EEEEAKCLEK FQDADPLEQD ELHTFTDTML PGCFHLLDEL PDTVYRVCDL IMTAIKRNGA DYRDMILKQV VNQVWEAADV LIKAALPLTT SDTKTVSEWI SQMATLPQAS NLATRILLLT LLFEELKLPC AWVVESSGIL NVLIKLLEVV QPCLQAAKEQ KEVQTPKWIT PVLLLIDFYE KTAISSKRRA QMTKYLQSNS NNWRWFDDRS GRWCSYSASN NSTIDSAWKS GETSVRFTAG RRRYTVQFTT MVQVNEETGN RRPVMLTLLR VPRLNKNSKN SNGQELEKTL EESKEMDIKR KENKGNDTPL ALESTNTEKE TSLEETKIGE ILIQGLTEDM VTVLIRACVS MLGVPVDPDT LHATLRLCLR LTRDHKYAMM FAELKSTRMI LNLTQSSGFN GFTPLVTLLL RHIIEDPCTL RHTMEKVVRS AATSGAGSTT SGVVSGSLGS REINYILRVL GPAACRNPDI FTEVANCCIR IALPAPRGSG TASDDEFENL RIKGPNAVQL VKTTPLKPSP LPVIPDTIKE VIYDMLNALA AYHAPEEADK SDPKPGVMTQ EVGQLLQDMG DDVYQQYRSL TRQSSDFDTQ SGFSINSQVF AADGASTETS ASGTSQGEAS TPEESRDGKK DKEGDRASEE GKQKGKGSKP LMPTSTILRL LAELVRSYVG IATLIANYSY TVGQSELIKE DCSVLAFVLD HLLPHTQNAE DKDTPALARL FLASLAAAGS GTDAQVALVN EVKAALGRAL AMAESTEKHA RLQAVMCIIS TIMESCPSTS SFYSSATAKT QHNGMNNIIR LFLKKGLVND LARVPHSLDL SSPNMANTVN AALKPLETLS RIVNQPSSLF GSKSASSKNK SEQDAQGASQ DSSSNQQDPG EPGEAEVQEE DHDVTQTEVA DGDIMDGEAE TDSVVIAGQP EVLSSQEMQV ENELEDLIDE LLERDGGSGN STIIVSRSGE DESQEDVLMD EAPSNLSQAS TLQANREDSM NILDPEDEEE HTQEEDSSGS NEDEDDSQDE EEEEEEDEED DQEDDEGEEG DEDDDDDGSE MELDEDYPDM NASPLVRFER FDREDDLIIE FDNMFSSATD IPPSPGNIPT THPLMVRHAD HSSLTLGSGS STTRLTQGIG RSQRTLRQLT ANTGHTIHVH YPGNRQPNPP LILQRLLGPS AAADILQLSS SLPLQSRGRA RLLVGNDDVH IIARSDDELL DDFFHDQSTA TSQAGTLSSI PTALTRWTEE CKVLDAESMH DCVSVVKVSI VNHLEFLRDE ELEERREKRR KQLAEEETKI TDKGKEDKEN RDQSAQCTAS KSNDSTEQNL SDGTPMPDSY PTTPSSTDAA TSESKETLGT LQSSQQQPTL PTPPALGEVP QELQSPAGEG GSSTQLLMPV EPEELGPTRP SGEAETTQME LSPAPTITSL SPERAEDSDA LTAVSSQLEG SPMDTSSLAS CTLEEAVGDT SAAGSSEQPR AGSSTPGDAP PAVAEVQGRS DGSGESAQPP EDSSPPASSE SSSTRDSAVA ISGADSRGIL EEPLPSTSSE EEDPLAGISL PEGVDPSFLA ALPDDIRREV LQNQLGIRPP TRTAPSTNSS APAVVGNPGV TEVSPEFLAA LPPAIQEEVL AQQRAEQQRR ELAQNASSDT PMDPVTFIQT LPSDLRRSVL EDMEDSVLAV MPPDIAAEAQ ALRREQEARQ RQLMHERLFG HSSTSALSAI LRSPAFTSRL SGNRGVQYTR LAVQRGGTFQ MGGSSSHNRP SGSNVDTLLR LRGRLLLDHE ALSCLLVLLF VDEPKLNTSR LHRVLRNLCY HAQTRHWVIR SLLSILQRSS ESELCIETPK LTTSEEKGKK SSKSCGSSSH ENRPLDLLHK MESKSSNQLS WLSVSMDAAL GCRTNIFQIQ RSGGRKHTEK HASGGSTVHI HPQAAPVVCR HVLDTLIQLA KVFPSHFTQQ RTKETNCESD RERGNKACSP CSSQSSSSGI CTDFWDLLVK LDNMNVSRKG KNSVKSVPVS AGGEGETSPY SLEASPLGQL MNMLSHPVIR RSSLLTEKLL RLLSLISIAL PENKVSEAQA NSGSGASSTT TATSTTSTTT TTAASTTPTP PTAPTPVTSA PALVAATAIS TIVVAASTTV TTPTTATTTV SISPTTKGSK SPAKVSDGGS SSTDFKMVSS GLTENQLQLS VEVLTSHSCS EEGLEDAANV LLQLSRGDSG TRDTVLKLLL NGARHLGYTL CKQIGTLLAE LREYNLEQQR RAQCETLSPD GLPEEQPQTT KLKGKMQSRF DMAENVVIVA SQKRPLGGRE LQLPSMSMLT SKTSTQKFFL RVLQVIIQLR DDTRRANKKA KQTGRLGSSG LGSASSIQAA VRQLEAEADA IIQMVREGQR ARRQQQAATS ESSQSEASVR REESPMDVDQ PSPSAQDTQS IASDGTPQGE KEKEERPPEL PLLSEQLSLD ELWDMLGECL KELEESHDQH AVLVLQPAVE AFFLVHATER ESKPPVRDTR ESQLAHIKDE PPPLSPAPLT PATPSSLDPF FSREPSSMHI SSSLPPDTQK FLRFAETHRT VLNQILRQST THLADGPFAV LVDYIRVLDF DVKRKYFRQE LERLDEGLRK EDMAVHVRRD HVFEDSYREL HRKSPEEMKN RLYIVFEGEE GQDAGGLLRE WYMIISREMF NPMYALFRTS PGDRVTYTIN PSSHCNPNHL SYFKFVGRIV AKAVYDNRLL ECYFTRSFYK HILGKSVRYT DMESEDYHFY QGLVYLLEND VSTLGYDLTF STEVQEFGVC EVRDLKPNGA NILVTEENKK EYVHLVCQMR MTGAIRKQLA AFLEGFYEII PKRLISIFTE QELELLISGL PTIDIDDLKS NTEYHKYQSN SIQIQWFWRA LRSFDQADRA KFLQFVTGTS KVPLQGFAAL EGMNGIQKFQ IHRDDRSTDR LPSAHTCFNQ LDLPAYESFE KLRHMLLLAI QECSEGFGLA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 12.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 91 % / チャンバー内温度: 283 K / 装置: LEICA EM GP / 詳細: CHAPSO detergent added to final conc. of 1 mM.. | |||||||||
| 詳細 | Monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.2 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 36000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.2 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 詳細 | Data collection in counting mode |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 3862 / 平均露光時間: 6.0 sec. / 平均電子線量: 54.86 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X