+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23805 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli MsbA in complex with G247 | |||||||||
 マップデータ マップデータ | E. coli MsbA in nanodisc in complex with G247 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Membrane protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ATPase-coupled lipid transmembrane transporter activity / ABC-type transporter activity / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Thelot F / Liao M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: Distinct allosteric mechanisms of first-generation MsbA inhibitors. 著者: François A Thélot / Wenyi Zhang / KangKang Song / Chen Xu / Jing Huang / Maofu Liao /   要旨: ATP-binding cassette (ABC) transporters couple adenosine 5′-triphosphate (ATP) hydrolysis to substrate transport across biological membranes. Although many are promising drug targets, their ...ATP-binding cassette (ABC) transporters couple adenosine 5′-triphosphate (ATP) hydrolysis to substrate transport across biological membranes. Although many are promising drug targets, their mechanisms of modulation by small-molecule inhibitors remain largely unknown. Two first-generation inhibitors of the MsbA transporter, tetrahydrobenzothiophene 1 (TBT1) and G247, induce opposite effects on ATP hydrolysis. Using single-particle cryo–electron microscopy and functional assays, we show that TBT1 and G247 bind adjacent yet separate pockets in the MsbA transmembrane domains. Two TBT1 molecules asymmetrically occupy the substrate-binding site, which leads to a collapsed inward-facing conformation with decreased distance between the nucleotide-binding domains (NBDs). By contrast, two G247 molecules symmetrically increase NBD distance in a wide inward-open state of MsbA. The divergent mechanisms of action of these MsbA inhibitors provide important insights into ABC transporter pharmacology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23805.map.gz emd_23805.map.gz | 4.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23805-v30.xml emd-23805-v30.xml emd-23805.xml emd-23805.xml | 12 KB 12 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23805.png emd_23805.png | 83.4 KB | ||
| Filedesc metadata |  emd-23805.cif.gz emd-23805.cif.gz | 5.5 KB | ||
| その他 |  emd_23805_additional_1.map.gz emd_23805_additional_1.map.gz | 19.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23805 http://ftp.pdbj.org/pub/emdb/structures/EMD-23805 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23805 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23805 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23805_validation.pdf.gz emd_23805_validation.pdf.gz | 445.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23805_full_validation.pdf.gz emd_23805_full_validation.pdf.gz | 445.4 KB | 表示 | |
| XML形式データ |  emd_23805_validation.xml.gz emd_23805_validation.xml.gz | 4.5 KB | 表示 | |
| CIF形式データ |  emd_23805_validation.cif.gz emd_23805_validation.cif.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23805 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23805 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23805 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23805 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7mewMC  7metC  7ritC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10799 (タイトル: E. coli MsbA in nanodiscs in complex with G247 EMPIAR-10799 (タイトル: E. coli MsbA in nanodiscs in complex with G247Data size: 442.5 Data #1: Motion-corrected micrographs of E. coli MsbA in nanodiscs complex with G247 - Krios Data [micrographs - single frame] Data #2: Motion-corrected micrographs of E. coli MsbA in nanodiscs complex with G247 - Polara Data [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23805.map.gz / 形式: CCP4 / 大きさ: 4.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23805.map.gz / 形式: CCP4 / 大きさ: 4.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli MsbA in nanodisc in complex with G247 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.23 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unfiltered, unsharpened map
| ファイル | emd_23805_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered, unsharpened map | ||||||||||||
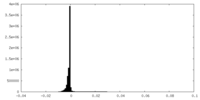
| 投影像・断面図 |
| ||||||||||||
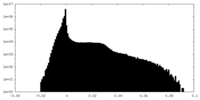
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E. coli MsbA in complex with G247
| 全体 | 名称: E. coli MsbA in complex with G247 |
|---|---|
| 要素 |
|
-超分子 #1: E. coli MsbA in complex with G247
| 超分子 | 名称: E. coli MsbA in complex with G247 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 130 KDa |
-分子 #1: ATP-dependent lipid A-core flippase
| 分子 | 名称: ATP-dependent lipid A-core flippase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: ABC-type lipid A-core oligosaccharide transporter |
|---|---|
| 由来(天然) | 生物種:  株: B / BL21-DE3 |
| 分子量 | 理論値: 67.310445 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHHH HHSSGHIDDD DKHMHNDKDL STWQTFRRLW PTIAPFKAGL IVAGVALILN AASDTFMLSL LKPLLDDGFG KTDRSVLVW MPLVVIGLMI LRGITSYVSS YCISWVSGKV VMTMRRRLFG HMMGMPVSFF DKQSTGTLLS RITYDSEQVA S SSSGALIT ...文字列: MGHHHHHHHH HHSSGHIDDD DKHMHNDKDL STWQTFRRLW PTIAPFKAGL IVAGVALILN AASDTFMLSL LKPLLDDGFG KTDRSVLVW MPLVVIGLMI LRGITSYVSS YCISWVSGKV VMTMRRRLFG HMMGMPVSFF DKQSTGTLLS RITYDSEQVA S SSSGALIT VVREGASIIG LFIMMFYYSW QLSIILIVLA PIVSIAIRVV SKRFRNISKN MQNTMGQVTT SAEQMLKGHK EV LIFGGQE VETKRFDKVS NRMRLQGMKM VSASSISDPI IQLIASLALA FVLYAASFPS VMDSLTAGTI TVVFSSMIAL MRP LKSLTN VNAQFQRGMA ACQTLFTILD SEQEKDEGKR VIERATGDVE FRNVTFTYPG RDVPALRNIN LKIPAGKTVA LVGR SGSGK STIASLITRF YDIDEGEILM DGHDLREYTL ASLRNQVALV SQNVHLFNDT VANNIAYART EQYSREQIEE AARMA YAMD FINKMDNGLD TVIGENGVLL SGGQRQRIAI ARALLRDSPI LILDEATSAL DTESERAIQA ALDELQKNRT SLVIAH RLS TIEKADEIVV VEDGVIVERG THNDLLEHRG VYAQLHKMQF GQ UniProtKB: Lipid A ABC exporter, fused ATPase and inner membrane subunits MsbA |
-分子 #2: (2E)-3-{1-cyclopropyl-7-[(1S)-1-(3,6-dichloro-2-fluorophenyl)etho...
| 分子 | 名称: (2E)-3-{1-cyclopropyl-7-[(1S)-1-(3,6-dichloro-2-fluorophenyl)ethoxy]naphthalen-2-yl}prop-2-enoic acid タイプ: ligand / ID: 2 / コピー数: 2 / 式: Z5Y |
|---|---|
| 分子量 | 理論値: 445.31 Da |
| Chemical component information | 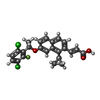 ChemComp-Z5Y: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 63.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 349674 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X