+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23492 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of squirrel TRPV1 in complex with RTX | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Transient Receptor Potential V Family Member 1 / TRP channel / TRPV1 full length / TRPV1 wild type / RTX / resiniferatoxin / MEMBRANE PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報temperature-gated ion channel activity / response to capsazepine / sensory perception of mechanical stimulus / peptide secretion / detection of chemical stimulus involved in sensory perception of pain / smooth muscle contraction involved in micturition / cellular response to acidic pH / fever generation / detection of temperature stimulus involved in thermoception / dendritic spine membrane ...temperature-gated ion channel activity / response to capsazepine / sensory perception of mechanical stimulus / peptide secretion / detection of chemical stimulus involved in sensory perception of pain / smooth muscle contraction involved in micturition / cellular response to acidic pH / fever generation / detection of temperature stimulus involved in thermoception / dendritic spine membrane / calcium ion import across plasma membrane / diet induced thermogenesis / detection of temperature stimulus involved in sensory perception of pain / behavioral response to pain / extracellular ligand-gated monoatomic ion channel activity / phosphoprotein binding / calcium channel activity / lipid metabolic process / cellular response to heat / postsynaptic membrane / calmodulin binding / negative regulation of transcription by RNA polymerase II / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Ictidomys tridecemlineatus (哺乳類) Ictidomys tridecemlineatus (哺乳類) | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.41 Å | |||||||||||||||
 データ登録者 データ登録者 | Neuberger A / Nadezhdin KD | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Extracellular cap domain is an essential component of the TRPV1 gating mechanism. 著者: Kirill D Nadezhdin / Arthur Neuberger / Yury A Nikolaev / Lyle A Murphy / Elena O Gracheva / Sviatoslav N Bagriantsev / Alexander I Sobolevsky /  要旨: Transient receptor potential (TRP) channels are polymodal molecular sensors involved in numerous physiological processes and implicated in a variety of human diseases. Several structures of the ...Transient receptor potential (TRP) channels are polymodal molecular sensors involved in numerous physiological processes and implicated in a variety of human diseases. Several structures of the founding member of the TRP channel family, TRPV1, are available, all of which were determined for the protein missing the N- and C-termini and the extracellular S5-P-loop. Here, we present structures of the full-length thirteen-lined ground squirrel TRPV1 solved by cryo-EM. Our structures resolve the extracellular cap domain formed by the S5-P-loops and the C-terminus that wraps around the three-stranded β-sheet connecting elements of the TRPV1 intracellular skirt. The cap domain forms a dome above the pore's extracellular entrance, with four portals leading to the ion conductance pathway. Deletion of the cap increases the TRPV1 average conductance, reduces the open probability and affects ion selectivity. Our data show that both the termini and the cap domain are critical determinants of TRPV1 function. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23492.map.gz emd_23492.map.gz | 97.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23492-v30.xml emd-23492-v30.xml emd-23492.xml emd-23492.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23492.png emd_23492.png | 200.1 KB | ||
| Filedesc metadata |  emd-23492.cif.gz emd-23492.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23492 http://ftp.pdbj.org/pub/emdb/structures/EMD-23492 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23492 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23492 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23492_validation.pdf.gz emd_23492_validation.pdf.gz | 496 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23492_full_validation.pdf.gz emd_23492_full_validation.pdf.gz | 495.6 KB | 表示 | |
| XML形式データ |  emd_23492_validation.xml.gz emd_23492_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_23492_validation.cif.gz emd_23492_validation.cif.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23492 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23492 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23492 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23492 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23492.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23492.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
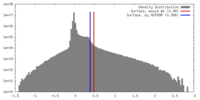
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : sample 1
| 全体 | 名称: sample 1 |
|---|---|
| 要素 |
|
-超分子 #1: sample 1
| 超分子 | 名称: sample 1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Osm-9-like TRP channel 1
| 分子 | 名称: Osm-9-like TRP channel 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Ictidomys tridecemlineatus (哺乳類) Ictidomys tridecemlineatus (哺乳類) |
| 分子量 | 理論値: 95.902781 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKKWANLDSG ESEDPPHEDS CPDPPDGDPN SKQSPAKPHI FSTTKSRTRL FGKGDSEEAS PMDCSYEEGE LASCPTITVS SVVIIQRPG DALTCARQLS QDSVSAGAEK PLKLYDRRSI FDAVAQNNCQ ELESLLPFLQ KSRKRLTDSE FKDPETGKTC L LKAMLNLH ...文字列: MKKWANLDSG ESEDPPHEDS CPDPPDGDPN SKQSPAKPHI FSTTKSRTRL FGKGDSEEAS PMDCSYEEGE LASCPTITVS SVVIIQRPG DALTCARQLS QDSVSAGAEK PLKLYDRRSI FDAVAQNNCQ ELESLLPFLQ KSRKRLTDSE FKDPETGKTC L LKAMLNLH NGQNDTISLL LDIARQTDSL KEFVNASYTD SYYKGQTALH IAIERRNMAL VTLLVENGAD VQAAANGDFF KK TKGRPGF YFGELPLSLA ACTNQLAIVK FLLQNSWQPA DISARDSVGN TVLHALVEVA DNTADNTKFV TSMYNEILIL GAK LHPTLK LEELINKKGL TPLALAASSG KIGVLAYILQ REIQEPECRH LSRKFTEWAY GPVHSSLYDL SCIDTCEKNS VLEV IAYSS SETPNRHDML LVEPLNRLLQ DKWDRFVKRI FYFNFFVYCL YMIVFTTAAY YRPVDGLPPY KLKNTVGDYF RVTGE ILSV SGGVYFFLRG IQYFLQRRPS MKTLFVDSYS EMLFFVQSLF MLGSVVLYFS HRKEYVASMV FSLAMGWTNM LYYTRG FQQ MGIYAVMIEK MILRDLCRFM FVYLVFLFGF STAVVTLIED GKNYSEPAEY TSHRWRAPGC RPPDSYNSLY STCLELF KF TIGMGDLEFT ENYDFKAVFI ILLLAYVILT YILLLNMLIA LMGETVNKIA QESKNIWKLQ RAITILDTEK SFLKCMRK A FRSGKLLQVG YTPDGKDDYR WCFRVDEVNW TTWNTNVGII NEDPGNCEGV KRTLSFSLRS GRVSGRNWRN FALVPLLRD ASTRDRHHAQ PEEVHLKHFA GSLKPEDAEV FKDSMAPGEK LPVR UniProtKB: Transient receptor potential cation channel subfamily V member 1 |
-分子 #2: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(tri...
| 分子 | 名称: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(trimethylammonio)ethyl phosphate タイプ: ligand / ID: 2 / コピー数: 32 / 式: POV |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-POV: |
-分子 #3: resiniferatoxin
| 分子 | 名称: resiniferatoxin / タイプ: ligand / ID: 3 / コピー数: 4 / 式: 6EU |
|---|---|
| 分子量 | 理論値: 628.708 Da |
| Chemical component information |  ChemComp-6EU: |
-分子 #4: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 25 sec. | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 30 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 3253 / 平均露光時間: 2.5 sec. / 平均電子線量: 70.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): -3.5 µm / 最小 デフォーカス(公称値): -1.5 µm |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-7lqz: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)