+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23271 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helitron transposase bound to LTS | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helitron / transposase / evolution / gene editing / gene capture / rolling circle / replication / recombination / nuclease / helicase / relaxase | |||||||||
| 生物種 | synthetic construct (人工物) /  Myotis lucifugus (トビイロホオヒゲコウモリ) Myotis lucifugus (トビイロホオヒゲコウモリ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.66 Å | |||||||||
 データ登録者 データ登録者 | Kosek D / Dyda F | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: The large bat Helitron DNA transposase forms a compact monomeric assembly that buries and protects its covalently bound 5'-transposon end. 著者: Dalibor Kosek / Ivana Grabundzija / Haotian Lei / Ilija Bilic / Huaibin Wang / Yukun Jin / Graham F Peaslee / Alison B Hickman / Fred Dyda /   要旨: Helitrons are widespread eukaryotic DNA transposons that have significantly contributed to genome variability and evolution, in part because of their distinctive, replicative rolling-circle ...Helitrons are widespread eukaryotic DNA transposons that have significantly contributed to genome variability and evolution, in part because of their distinctive, replicative rolling-circle mechanism, which often mobilizes adjacent genes. Although most eukaryotic transposases form oligomers and use RNase H-like domains to break and rejoin double-stranded DNA (dsDNA), Helitron transposases contain a single-stranded DNA (ssDNA)-specific HUH endonuclease domain. Here, we report the cryo-electron microscopy structure of a Helitron transposase bound to the 5'-transposon end, providing insight into its multidomain architecture and function. The monomeric transposase forms a tightly packed assembly that buries the covalently attached cleaved end, protecting it until the second end becomes available. The structure reveals unexpected architectural similarity to TraI, a bacterial relaxase that also catalyzes ssDNA movement. The HUH active site suggests how two juxtaposed tyrosines, a feature of many replication initiators that use HUH nucleases, couple the conformational shift of an α-helix to control strand cleavage and ligation reactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23271.map.gz emd_23271.map.gz | 28.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23271-v30.xml emd-23271-v30.xml emd-23271.xml emd-23271.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23271.png emd_23271.png | 103.5 KB | ||
| Filedesc metadata |  emd-23271.cif.gz emd-23271.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23271 http://ftp.pdbj.org/pub/emdb/structures/EMD-23271 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23271 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23271 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23271.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23271.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
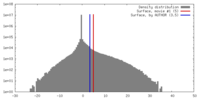
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Helraiser complex with LTS
| 全体 | 名称: Helraiser complex with LTS |
|---|---|
| 要素 |
|
-超分子 #1: Helraiser complex with LTS
| 超分子 | 名称: Helraiser complex with LTS / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 170 KDa |
-分子 #1: Helraiser K1068Q
| 分子 | 名称: Helraiser K1068Q / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 171.253484 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKEQLLIQR SSAAERCRRY RQKMSAEQRA SDLERRRRLQ QNVSEEQLLE KRRSEAEKQR RHRQKMSKDQ RAFEVERRRW RRQNMSREQ SSTSTTNTGR NCLLSKNGVH EDAILEHSCG GMTVRCEFCL SLNFSDEKPS DGKFTRCCSK GKVCPNDIHF P DYPAYLKR ...文字列: MSKEQLLIQR SSAAERCRRY RQKMSAEQRA SDLERRRRLQ QNVSEEQLLE KRRSEAEKQR RHRQKMSKDQ RAFEVERRRW RRQNMSREQ SSTSTTNTGR NCLLSKNGVH EDAILEHSCG GMTVRCEFCL SLNFSDEKPS DGKFTRCCSK GKVCPNDIHF P DYPAYLKR LMTNEDSDSK NFMENIRSIN SSFAFASMGA NIASPSGYGP YCFRIHGQVY HRTGTLHPSD GVSRKFAQLY IL DTAEATS KRLAMPENQG CSERLMININ NLMHEINELT KSYKMLHEVE KEAQSEAAAK GIAPTEVTMA IKYDRNSDPG RYN SPRVTE VAVIFRNEDG EPPFERDLLI HCKPDPNNPN ATKMKQISIL FPTLDAMTYP ILFPHGEKGW GTDIALRLRD NSVI DNNTR QNVRTRVTQM QYYGFHLSVR DTFNPILNAG KLTQQFIVDS YSKMEANRIN FIKANQSKLR VEKYSGLMDY LKSRS ENDN VPIGKMIILP SSFEGSPRNM QQRYQDAMAI VTKYGKPDLF ITMTCNPKWA DITNNLQRWQ KVENRPDLVA RVFNIK LNA LLNDICKFHL FGKVIAKIHV IEFQKRGLPH AHILLILDSE SKLRSEDDID RIVKAEIPDE DQCPRLFQIV KSNMVHG PC GIQNPNSPCM ENGKCSKGYP KEFQNATIGN IDGYPKYKRR SGSTMSIGNK VVDNTWIVPY NPYLCLKYNC HINVEVCA S IKSVKYLFKY IYKGHDCANI QISEKNIINH DEVQDFIDSR YVSAPEAVWR LFAMRMHDQS HAITRLAIHL PNDQNLYFH TDDFAEVLDR AKRHNSTLMA WFLLNREDSD ARNYYYWEIP QHYVFNNSLW TKRRKGGNKV LGRLFTVSFR EPERYYLRLL LLHVKGAIS FEDLRTVGGV TYDTFHEAAK HRGLLLDDTI WKDTIDDAII LNMPKQLRQL FAYICVFGCP SAADKLWDEN K SHFIEDFC WKLHRREGAC VNCEMHALNE IQEVFTLHGM KCSHFKLPDY PLLMNANTCD QLYEQQQAEV LINSLNDEQL AA FQTITSA IEDQTVHPKC FFLDGPGGSG QTYLYKVLTH YIRGRGGTVL PTASTGIAAN LLLGGRTFHS QYKLPIPLNE TSI SRLDIK SEVAKTIKKA QLLIIDECTM ASSHAINAID RLLREIMNLN VAFGGKVLLL GGDFRQCLSI VPHAMRSAIV QTSL KYCNV WGCFRKLSLK TNMRSEDSAY SEWLVKLGDG KLDSSFHLGM DIIEIPHEMI CNGSIIEATF GNSISIDNIK NISKR AILC PKNEHVQKLN EEILDILDGD FHTYLSDDSI DSTDDAEKEN FPIEFLNSIT PSGMPCHKLK LKVGAIIMLL RNLNSK WGL CNGTRFIIKR LRPNIIEAEV LTGSAEGEVV LIPRIDLSPS DTGLPFKLIR RQFPVMPAFA MTINKSQGQT LDRVGIF LP EPVFAHGQLY VAFSRVRRAC DVKVKVVNTS SQGKLVKHSE SVFTLNVVYR EILE |
-分子 #2: LTS
| 分子 | 名称: LTS / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Myotis lucifugus (トビイロホオヒゲコウモリ) Myotis lucifugus (トビイロホオヒゲコウモリ) |
| 分子量 | 理論値: 6.158058 KDa |
| 配列 | 文字列: (DT)(DC)(DC)(DT)(DA)(DT)(DA)(DT)(DA)(DA) (DT)(DA)(DA)(DA)(DA)(DG)(DA)(DG)(DA)(DA) |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 平均電子線量: 73.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | The initial model has been traced manualy, then refined in Rosetta and finished in Coot 9.0 |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-7lcc: |
 ムービー
ムービー コントローラー
コントローラー














 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)