+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22967 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain electron microscopy reconstruction of 2P SARS-CoV-2 spike ectodomain in the 3-RBD-down state after cold-denaturation at 4 degrees C for one week followed by re-naturation at 37 degrees C for 3 hours. | |||||||||
 マップデータ マップデータ | Final sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 16.8 Å | |||||||||
 データ登録者 データ登録者 | Edwards RJ / Mansouri K | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2020 ジャーナル: bioRxiv / 年: 2020タイトル: Cold sensitivity of the SARS-CoV-2 spike ectodomain. 著者: Robert J Edwards / Katayoun Mansouri / Victoria Stalls / Kartik Manne / Brian Watts / Rob Parks / Katarzyna Janowska / Sophie M C Gobeil / Megan Kopp / Dapeng Li / Xiaozhi Lu / Zekun Mu / ...著者: Robert J Edwards / Katayoun Mansouri / Victoria Stalls / Kartik Manne / Brian Watts / Rob Parks / Katarzyna Janowska / Sophie M C Gobeil / Megan Kopp / Dapeng Li / Xiaozhi Lu / Zekun Mu / Margaret Deyton / Thomas H Oguin / Jordan Sprenz / Wilton Williams / Kevin Saunders / David Montefiori / Gregory D Sempowski / Rory Henderson / Munir Alam / Barton F Haynes / Priyamvada Acharya 要旨: The SARS-CoV-2 spike (S) protein, a primary target for COVID-19 vaccine development, presents its Receptor Binding Domain in two conformations: receptor-accessible "up" or receptor-inaccessible ...The SARS-CoV-2 spike (S) protein, a primary target for COVID-19 vaccine development, presents its Receptor Binding Domain in two conformations: receptor-accessible "up" or receptor-inaccessible "down" conformations. Here, we report that the commonly used stabilized S ectodomain construct "2P" is sensitive to cold temperature, and that this cold sensitivity is resolved in a "down" state stabilized spike. Our results will impact structural, functional and vaccine studies that use the SARS-CoV-2 S ectodomain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22967.map.gz emd_22967.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22967-v30.xml emd-22967-v30.xml emd-22967.xml emd-22967.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22967_fsc.xml emd_22967_fsc.xml | 3.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22967.png emd_22967.png | 57 KB | ||
| マスクデータ |  emd_22967_msk_1.map emd_22967_msk_1.map | 3.4 MB |  マスクマップ マスクマップ | |
| その他 |  emd_22967_additional_1.map.gz emd_22967_additional_1.map.gz emd_22967_half_map_1.map.gz emd_22967_half_map_1.map.gz emd_22967_half_map_2.map.gz emd_22967_half_map_2.map.gz | 2.4 MB 2.4 MB 2.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22967 http://ftp.pdbj.org/pub/emdb/structures/EMD-22967 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22967 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22967 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22967_validation.pdf.gz emd_22967_validation.pdf.gz | 78.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22967_full_validation.pdf.gz emd_22967_full_validation.pdf.gz | 77.3 KB | 表示 | |
| XML形式データ |  emd_22967_validation.xml.gz emd_22967_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22967 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22967 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22967 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22967 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22967.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22967.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.02 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
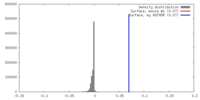
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22967_msk_1.map emd_22967_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
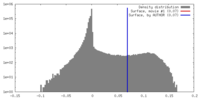
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_22967_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
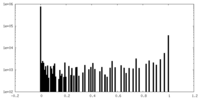
| 密度ヒストグラム |
-ハーフマップ: Half-map 1
| ファイル | emd_22967_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
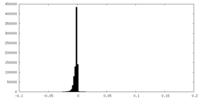
| 密度ヒストグラム |
-ハーフマップ: Half-map2
| ファイル | emd_22967_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 2P SARS-CoV-2 spike ectodomain trimer
| 全体 | 名称: 2P SARS-CoV-2 spike ectodomain trimer |
|---|---|
| 要素 |
|
-超分子 #1: 2P SARS-CoV-2 spike ectodomain trimer
| 超分子 | 名称: 2P SARS-CoV-2 spike ectodomain trimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: 293F / 組換プラスミド: p-alpha-H Homo sapiens (ヒト) / 組換細胞: 293F / 組換プラスミド: p-alpha-H |
| 分子量 | 理論値: 481.8 KDa |
-分子 #1: 2P SARS-CoV-2 spike ectodomain
| 分子 | 名称: 2P SARS-CoV-2 spike ectodomain / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY ...文字列: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDGV YFASTEKSNI IRGWIFGTTL DSKTQSLLIV NNATNVVIKV CEFQFCNDPF LGVYYHKNNK SWMESEFRVY SSANNCTFEY VSQPFLMDLE GKQGNFKNLR EFVFKNIDGY FKIYSKHTPI NLVRDLPQGF SALEPLVDLP IGINITRFQT LLALHRSYLT PGDSSSGWTA GAAAYYVGYL QPRTFLLKYN ENGTITDAVD CALDPLSETK CTLKSFTVEK GIYQTSNFRV QPTESIVRFP NITNLCPFGE VFNATRFASV YAWNRKRISN CVADYSVLYN SASFSTFKCY GVSPTKLNDL CFTNVYADSF VIRGDEVRQI APGQTGKIAD YNYKLPDDFT GCVIAWNSNN LDSKVGGNYN YLYRLFRKSN LKPFERDIST EIYQAGSTPC NGVEGFNCYF PLQSYGFQPT NGVGYQPYRV VVLSFELLHA PATVCGPKKS TNLVKNKCVN FNFNGLTGTG VLTESNKKFL PFQQFGRDIA DTTDAVRDPQ TLEILDITPC SFGGVSVITP GTNTSNQVAV LYQDVNCTEV PVAIHADQLT PTWRVYSTGS NVFQTRAGCL IGAEHVNNSY ECDIPIGAGI CASYQTQTNS PGSASSVASQ SIIAYTMSLG AENSVAYSNN SIAIPTNFTI SVTTEILPVS MTKTSVDCTM YICGDSTECS NLLLQYGSFC TQLNRALTGI AVEQDKNTQE VFAQVKQIYK TPPIKDFGGF NFSQILPDPS KPSKRSFIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFGA GAALQIPFAM QMAYRFNGIG VTQNVLYENQ KLIANQFNSA IGKIQDSLSS TASALGKLQD VVNQNAQALN TLVKQLSSNF GAISSVLNDI LSRLDPPEAE VQIDRLITGR LQSLQTYVTQ QLIRAAEIRA SANLAATKMS ECVLGQSKRV DFCGKGYHLM SFPQSAPHGV VFLHVTYVPA QEKNFTTAPA ICHDGKAHFP REGVFVSNGT HWFVTQRNFY EPQIITTDNT FVSGNCDVVI GIVNNTVYDP LQPELDSFKE ELDKYFKNHT SPDVDLGDIS GINASVVNIQ KEIDRLNEVA KNLNESLIDL QELGKYEQGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGRS LEVLFQGPGH HHHHHHHSAW SHPQFEKGGG SGGGGSGGSA WSHPQFEK |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Samples were stored in buffer at 4 degrees C for 1 week, then moved to a 37 degrees C heat block for 3 hours prior to dilution and preparation of negatively stained grids. | ||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate 詳細: Samples were diluted to 0.1 mg/mL in 20 mM HEPES buffer, pH 7.4, with 5% glycerol, 150 mM NaCl, and 7.5 mM glutaraldehyde. After 5 minute incubation at room temperature, sufficient 1 M Tris ...詳細: Samples were diluted to 0.1 mg/mL in 20 mM HEPES buffer, pH 7.4, with 5% glycerol, 150 mM NaCl, and 7.5 mM glutaraldehyde. After 5 minute incubation at room temperature, sufficient 1 M Tris stock, pH 7.4, was added to a final concentration of 75 mM Tris to quench unreacted glutaraldehyde, and was then incubated 5 minutes. Sample was then applied to a carbon film over 400 mesh copper EM grids that had been glow-discharged, incubated 1 minute, and then stained with 2% uranyl formate. | ||||||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 0.05 nm / 前処理 - タイプ: GLOW DISCHARGE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS EM420 |
|---|---|
| 撮影 | フィルム・検出器のモデル: OTHER / デジタル化 - サイズ - 横: 2048 pixel / デジタル化 - サイズ - 縦: 2048 pixel / デジタル化 - サンプリング間隔: 33.0 µm / 撮影したグリッド数: 1 / 実像数: 124 / 平均露光時間: 0.5 sec. / 平均電子線量: 32.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 82000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)