+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2236 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative Staining Structure of Human Polycomb Repressive Complex 2 bound to the co-factor AEBP2 | |||||||||
 マップデータ マップデータ | Negative Staining Reconstruction of the Polycomb Repressive Complex 2 bound to the co-factor AEBP2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Polycomb / PRC2 / epigenetic / gene silencing / nucleosome / labeling / chemical cross-linking / chromatin | |||||||||
| 機能・相同性 | ESC/E(Z) complex / SET domain 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 21.0 Å | |||||||||
 データ登録者 データ登録者 | Ciferri C / Lander GC / Maiolica A / Herzog F / Aebersold R / Nogales E | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2012 ジャーナル: Elife / 年: 2012タイトル: Molecular architecture of human polycomb repressive complex 2. 著者: Claudio Ciferri / Gabriel C Lander / Alessio Maiolica / Franz Herzog / Ruedi Aebersold / Eva Nogales /  要旨: Polycomb Repressive Complex 2 (PRC2) is essential for gene silencing, establishing transcriptional repression of specific genes by tri-methylating Lysine 27 of histone H3, a process mediated by ...Polycomb Repressive Complex 2 (PRC2) is essential for gene silencing, establishing transcriptional repression of specific genes by tri-methylating Lysine 27 of histone H3, a process mediated by cofactors such as AEBP2. In spite of its biological importance, little is known about PRC2 architecture and subunit organization. Here, we present the first three-dimensional electron microscopy structure of the human PRC2 complex bound to its cofactor AEBP2. Using a novel internal protein tagging-method, in combination with isotopic chemical cross-linking and mass spectrometry, we have localized all the PRC2 subunits and their functional domains and generated a detailed map of interactions. The position and stabilization effect of AEBP2 suggests an allosteric role of this cofactor in regulating gene silencing. Regions in PRC2 that interact with modified histone tails are localized near the methyltransferase site, suggesting a molecular mechanism for the chromatin-based regulation of PRC2 activity.DOI:http://dx.doi.org/10.7554/eLife.00005.001. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2236.map.gz emd_2236.map.gz | 3.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2236-v30.xml emd-2236-v30.xml emd-2236.xml emd-2236.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2236.jpg emd_2236.jpg | 482.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2236 http://ftp.pdbj.org/pub/emdb/structures/EMD-2236 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2236 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2236 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2236_validation.pdf.gz emd_2236_validation.pdf.gz | 186.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2236_full_validation.pdf.gz emd_2236_full_validation.pdf.gz | 185.3 KB | 表示 | |
| XML形式データ |  emd_2236_validation.xml.gz emd_2236_validation.xml.gz | 5.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2236 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2236 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2236 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2236 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2236.map.gz / 形式: CCP4 / 大きさ: 5.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2236.map.gz / 形式: CCP4 / 大きさ: 5.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative Staining Reconstruction of the Polycomb Repressive Complex 2 bound to the co-factor AEBP2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.14 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
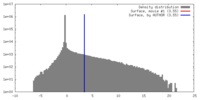
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Negative Staining Reconstruction of Polycomb repressive Complex 2...
| 全体 | 名称: Negative Staining Reconstruction of Polycomb repressive Complex 2 bound to the co-factor AEBP2 |
|---|---|
| 要素 |
|
-超分子 #1000: Negative Staining Reconstruction of Polycomb repressive Complex 2...
| 超分子 | 名称: Negative Staining Reconstruction of Polycomb repressive Complex 2 bound to the co-factor AEBP2 タイプ: sample / ID: 1000 / 集合状態: Pentameric Complex / Number unique components: 5 |
|---|---|
| 分子量 | 理論値: 265 MDa |
-分子 #1: RbAp48
| 分子 | 名称: RbAp48 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus |
| 組換発現 | 生物種:  組換プラスミド: pFast Bac |
| 配列 | GO: ESC/E(Z) complex / InterPro: SET domain |
-分子 #2: Ezh2
| 分子 | 名称: Ezh2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus |
| 組換発現 | 生物種:  組換プラスミド: pFast Bac |
| 配列 | GO: ESC/E(Z) complex / InterPro: SET domain |
-分子 #3: Suz12
| 分子 | 名称: Suz12 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus |
| 組換発現 | 生物種:  組換プラスミド: pFast Bac |
| 配列 | GO: ESC/E(Z) complex / InterPro: SET domain |
-分子 #4: AEBP2
| 分子 | 名称: AEBP2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus |
| 組換発現 | 生物種:  組換プラスミド: pFast Bac |
| 配列 | GO: ESC/E(Z) complex / InterPro: SET domain |
-分子 #5: EED
| 分子 | 名称: EED / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Nucleus |
| 組換発現 | 生物種:  組換プラスミド: pFast Bac |
| 配列 | GO: ESC/E(Z) complex / InterPro: SET domain |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 詳細: 25 mM Hepes pH 7.5, 138 mM NaCl, 10% Glycerol, 0.05% NP40, 1 mM Tris(2-carboxyethyl) phosphine hydrochloride [TCEP] |
| 染色 | タイプ: NEGATIVE / 詳細: 2% Uranyl Formate |
| グリッド | 詳細: 400 mesh copper carbon support |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 温度 | 平均: 298 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 150,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 日付 | 2011年4月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) 実像数: 500 / 平均電子線量: 20 e/Å2 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 80000 / 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD / Cs: 2.2 mm / 最大 デフォーカス(公称値): 1.3 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| 詳細 | Material and Methods section of Paper |
|---|---|
| CTF補正 | 詳細: whole micrograph |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 21.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF ソフトウェア - 名称: Appion, Leginon, Spider, Imagic, EMAN2, SPARX 使用した粒子像数: 39527 |
| 最終 2次元分類 | クラス数: 1000 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: local rigid-body fitting algorithm |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: local rigid-body fitting algorithm |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)