+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21308 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
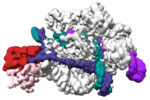
| タイトル | Structure of the SpCas9 DNA adenine base editor - ABE8e | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Base editor / ABE / Cas9 / CRISPR / DNA BINDING PROTEIN-DNA-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tRNA(adenine34) deaminase / tRNA wobble adenosine to inosine editing / tRNA-specific adenosine-34 deaminase activity / cytidine metabolic process / pyrimidine-containing compound salvage / cytosine deaminase activity / adenosine to inosine editing / cytosine metabolic process / maintenance of CRISPR repeat elements / 3'-5' exonuclease activity ...tRNA(adenine34) deaminase / tRNA wobble adenosine to inosine editing / tRNA-specific adenosine-34 deaminase activity / cytidine metabolic process / pyrimidine-containing compound salvage / cytosine deaminase activity / adenosine to inosine editing / cytosine metabolic process / maintenance of CRISPR repeat elements / 3'-5' exonuclease activity / DNA endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / protein homodimerization activity / DNA binding / RNA binding / zinc ion binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus pyogenes (化膿レンサ球菌) / Streptococcus pyogenes (化膿レンサ球菌) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Knott GJ / Lapinaite A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: DNA capture by a CRISPR-Cas9-guided adenine base editor. 著者: Audrone Lapinaite / Gavin J Knott / Cody M Palumbo / Enrique Lin-Shiao / Michelle F Richter / Kevin T Zhao / Peter A Beal / David R Liu / Jennifer A Doudna /   要旨: CRISPR-Cas-guided base editors convert A•T to G•C, or C•G to T•A, in cellular DNA for precision genome editing. To understand the molecular basis for DNA adenosine deamination by adenine base ...CRISPR-Cas-guided base editors convert A•T to G•C, or C•G to T•A, in cellular DNA for precision genome editing. To understand the molecular basis for DNA adenosine deamination by adenine base editors (ABEs), we determined a 3.2-angstrom resolution cryo-electron microscopy structure of ABE8e in a substrate-bound state in which the deaminase domain engages DNA exposed within the CRISPR-Cas9 R-loop complex. Kinetic and structural data suggest that ABE8e catalyzes DNA deamination up to ~1100-fold faster than earlier ABEs because of mutations that stabilize DNA substrates in a constrained, transfer RNA-like conformation. Furthermore, ABE8e's accelerated DNA deamination suggests a previously unobserved transient DNA melting that may occur during double-stranded DNA surveillance by CRISPR-Cas9. These results explain ABE8e-mediated base-editing outcomes and inform the future design of base editors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21308.map.gz emd_21308.map.gz | 41 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21308-v30.xml emd-21308-v30.xml emd-21308.xml emd-21308.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21308.png emd_21308.png | 223.9 KB | ||
| Filedesc metadata |  emd-21308.cif.gz emd-21308.cif.gz | 7.6 KB | ||
| その他 |  emd_21308_half_map_1.map.gz emd_21308_half_map_1.map.gz emd_21308_half_map_2.map.gz emd_21308_half_map_2.map.gz | 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21308 http://ftp.pdbj.org/pub/emdb/structures/EMD-21308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21308 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21308_validation.pdf.gz emd_21308_validation.pdf.gz | 768.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21308_full_validation.pdf.gz emd_21308_full_validation.pdf.gz | 768.3 KB | 表示 | |
| XML形式データ |  emd_21308_validation.xml.gz emd_21308_validation.xml.gz | 12.2 KB | 表示 | |
| CIF形式データ |  emd_21308_validation.cif.gz emd_21308_validation.cif.gz | 14.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21308 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21308 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21308 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21308 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21308.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21308.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.94 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_21308_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
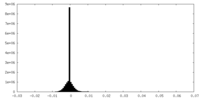
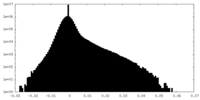
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_21308_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
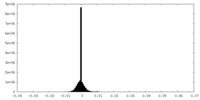
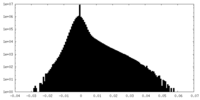
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CRISPR-Cas9 ABE8e in a substrate-bound state
| 全体 | 名称: CRISPR-Cas9 ABE8e in a substrate-bound state |
|---|---|
| 要素 |
|
-超分子 #1: CRISPR-Cas9 ABE8e in a substrate-bound state
| 超分子 | 名称: CRISPR-Cas9 ABE8e in a substrate-bound state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 詳細: ABE8e in a substrate-bound state where the deaminase (TadA-8e) engages DNA exposed within the CRISPR-Cas9 R-loop complex. |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
-分子 #1: Cas9 (SpCas9) single-guide RNA (sgRNA)
| 分子 | 名称: Cas9 (SpCas9) single-guide RNA (sgRNA) / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 26.357629 KDa |
| 配列 | 文字列: GGUUCCACUU UCUUAGACUG AGUUUUAGAG CUAGAAAUAG CAAGUUAAAA UAAGGCUAGU CCGUUAUCAA CUUGAAAAAG UG |
-分子 #2: CRISPR-associated endonuclease Cas9
| 分子 | 名称: CRISPR-associated endonuclease Cas9 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pyogenes (化膿レンサ球菌) Streptococcus pyogenes (化膿レンサ球菌) |
| 分子量 | 理論値: 157.870984 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KYSIGLAIGT NSVGWAVITD EYKVPSKKFK VLGNTDRHSI KKNLIGALLF DSGETAEATR LKRTARRRYT RRKNRICYLQ EIFSNEMAK VDDSFFHRLE ESFLVEEDKK HERHPIFGNI VDEVAYHEKY PTIYHLRKKL VDSTDKADLR LIYLALAHMI K FRGHFLIE ...文字列: KYSIGLAIGT NSVGWAVITD EYKVPSKKFK VLGNTDRHSI KKNLIGALLF DSGETAEATR LKRTARRRYT RRKNRICYLQ EIFSNEMAK VDDSFFHRLE ESFLVEEDKK HERHPIFGNI VDEVAYHEKY PTIYHLRKKL VDSTDKADLR LIYLALAHMI K FRGHFLIE GDLNPDNSDV DKLFIQLVQT YNQLFEENPI NASGVDAKAI LSARLSKSRR LENLIAQLPG EKKNGLFGNL IA LSLGLTP NFKSNFDLAE DAKLQLSKDT YDDDLDNLLA QIGDQYADLF LAAKNLSDAI LLSDILRVNT EITKAPLSAS MIK RYDEHH QDLTLLKALV RQQLPEKYKE IFFDQSKNGY AGYIDGGASQ EEFYKFIKPI LEKMDGTEEL LVKLNREDLL RKQR TFDNG SIPHQIHLGE LHAILRRQED FYPFLKDNRE KIEKILTFRI PYYVGPLARG NSRFAWMTRK SEETITPWNF EEVVD KGAS AQSFIERMTN FDKNLPNEKV LPKHSLLYEY FTVYNELTKV KYVTEGMRKP AFLSGEQKKA IVDLLFKTNR KVTVKQ LKE DYFKKIECFD SVEISGVEDR FNASLGTYHD LLKIIKDKDF LDNEENEDIL EDIVLTLTLF EDREMIEERL KTYAHLF DD KVMKQLKRRR YTGWGRLSRK LINGIRDKQS GKTILDFLKS DGFANRNFMQ LIHDDSLTFK EDIQKAQVSG QGDSLHEH I ANLAGSPAIK KGILQTVKVV DELVKVMGRH KPENIVIEMA RENQTTQKGQ KNSRERMKRI EEGIKELGSQ ILKEHPVEN TQLQNEKLYL YYLQNGRDMY VDQELDINRL SDYDVDAIVP QSFLKDDSID NKVLTRSDKN RGKSDNVPSE EVVKKMKNYW RQLLNAKLI TQRKFDNLTK AERGGLSELD KAGFIKRQLV ETRQITKHVA QILDSRMNTK YDENDKLIRE VKVITLKSKL V SDFRKDFQ FYKVREINNY HHAHDAYLNA VVGTALIKKY PKLESEFVYG DYKVYDVRKM IAKSEQEIGK ATAKYFFYSN IM NFFKTEI TLANGEIRKR PLIETNGETG EIVWDKGRDF ATVRKVLSMP QVNIVKKTEV QTGGFSKESI LPKRNSDKLI ARK KDWDPK KYGGFDSPTV AYSVLVVAKV EKGKSKKLKS VKELLGITIM ERSSFEKNPI DFLEAKGYKE VKKDLIIKLP KYSL FELEN GRKRMLASAG ELQKGNELAL PSKYVNFLYL ASHYEKLKGS PEDNEQKQLF VEQHKHYLDE IIEQISEFSK RVILA DANL DKVLSAYNKH RDKPIREQAE NIIHLFTLTN LGAPAAFKYF DTTIDRKRYT STKEVLDATL IHQSITGLYE TRIDLS Q UniProtKB: CRISPR-associated endonuclease Cas9 |
-分子 #5: t-RNA adenine deaminase A v8e (TadA-8e)
| 分子 | 名称: t-RNA adenine deaminase A v8e (TadA-8e) / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 24.657887 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH MKRTADGSEF ESPKKKRKVS EVEFSHEYWM RHALTLAKRA RDEREVPVGA VLVLNNRVIG EGWNRAIGLH DPTAHAEIM ALRQGGLVMQ NYRLIDATLY VTFEPCVMCA GAMIHSRIGR VVFGVRNSKR GAAGSLMNVL NYPGMNHRVE I TEGILADE ...文字列: MGSSHHHHHH MKRTADGSEF ESPKKKRKVS EVEFSHEYWM RHALTLAKRA RDEREVPVGA VLVLNNRVIG EGWNRAIGLH DPTAHAEIM ALRQGGLVMQ NYRLIDATLY VTFEPCVMCA GAMIHSRIGR VVFGVRNSKR GAAGSLMNVL NYPGMNHRVE I TEGILADE CAALLCDFYR MPRQVFNAQK KAQSSINSGG SSGGSSGSET PGTSESATPE SSGGSSGGS |
-分子 #3: DNA target strand (TS)
| 分子 | 名称: DNA target strand (TS) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.390882 KDa |
| 配列 | 文字列: (DC)(DT)(DA)(DA)(DT)(DC)(DG)(DC)(DC)(DC) (DT)(DC)(DA)(DG)(DT)(DC)(DT)(DA)(DA)(DG) (DA)(DA)(DA)(DG)(DT)(DG)(DG)(DA)(DA) (DC)(DA)(DC)(DG)(DG)(DT)(DC)(DG)(DG)(DA) (DG) (DC)(DC)(DA)(DC)(DC)(DG)(DA)(DT) (DC)(DG) |
-分子 #4: DNA non-target strand (NTS)
| 分子 | 名称: DNA non-target strand (NTS) / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.436831 KDa |
| 配列 | 文字列: (DC)(DG)(DA)(DT)(DC)(DG)(DG)(DT)(DG)(DG) (DC)(DT)(DC)(DC)(DG)(DA)(DC)(DC)(DG)(DT) (DG)(DT)(DT)(DC)(DC)(8AZ)(DC)(DT)(DT) (DT)(DC)(DT)(DT)(DA)(DG)(DA)(DC)(DT)(DG) (DA)(DG)(DG)(DG)(DC)(DG)(DA)(DT)(DT) (DA)(DG) |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 7 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-HCL (pH 7.5) 200 mM KCl 1 mM DTT 0.25% (v/v) Glycerol |
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281.15 K / 装置: FEI VITROBOT MARK IV 詳細: The grids were rapidly plunged into liquid ethane using a FEI Vitrobot MarkIV maintained at 8C and 100% humidity, after being blotted for 4.5s with blot force 8.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 5022 / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 得られたモデル |  PDB-6vpc: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X