+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21225 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of PilA-N/C from Geobacter sulfurreducens | |||||||||||||||
 マップデータ マップデータ | Cryo-EM structure of PilA-N-C from Geobacter sulfurreducens | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Protein transport / Pili / PROTEIN FIBRIL | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報pilus assembly / protein secretion by the type II secretion system / type II protein secretion system complex / plasma membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) | |||||||||||||||
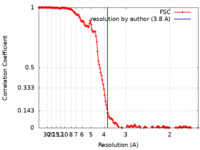
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||||||||
 データ登録者 データ登録者 | Gu Y / Srikanth V | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structure of Geobacter pili reveals secretory rather than nanowire behaviour. 著者: Yangqi Gu / Vishok Srikanth / Aldo I Salazar-Morales / Ruchi Jain / J Patrick O'Brien / Sophia M Yi / Rajesh Kumar Soni / Fadel A Samatey / Sibel Ebru Yalcin / Nikhil S Malvankar /  要旨: Extracellular electron transfer by Geobacter species through surface appendages known as microbial nanowires is important in a range of globally important environmental phenomena, as well as for ...Extracellular electron transfer by Geobacter species through surface appendages known as microbial nanowires is important in a range of globally important environmental phenomena, as well as for applications in bio-remediation, bioenergy, biofuels and bioelectronics. Since 2005, these nanowires have been thought to be type 4 pili composed solely of the PilA-N protein. However, previous structural analyses have demonstrated that, during extracellular electron transfer, cells do not produce pili but rather nanowires made up of the cytochromes OmcS and OmcZ. Here we show that Geobacter sulfurreducens binds PilA-N to PilA-C to assemble heterodimeric pili, which remain periplasmic under nanowire-producing conditions that require extracellular electron transfer. Cryo-electron microscopy revealed that C-terminal residues of PilA-N stabilize its copolymerization with PilA-C (to form PilA-N-C) through electrostatic and hydrophobic interactions that position PilA-C along the outer surface of the filament. PilA-N-C filaments lack π-stacking of aromatic side chains and show a conductivity that is 20,000-fold lower than that of OmcZ nanowires. In contrast with surface-displayed type 4 pili, PilA-N-C filaments show structure, function and localization akin to those of type 2 secretion pseudopili. The secretion of OmcS and OmcZ nanowires is lost when pilA-N is deleted and restored when PilA-N-C filaments are reconstituted. The substitution of pilA-N with the type 4 pili of other microorganisms also causes a loss of secretion of OmcZ nanowires. As all major phyla of prokaryotes use systems similar to type 4 pili, this nanowire translocation machinery may have a widespread effect in identifying the evolution and prevalence of diverse electron-transferring microorganisms and in determining nanowire assembly architecture for designing synthetic protein nanowires. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21225.map.gz emd_21225.map.gz | 17.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21225-v30.xml emd-21225-v30.xml emd-21225.xml emd-21225.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21225_fsc.xml emd_21225_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21225.png emd_21225.png | 175 KB | ||
| Filedesc metadata |  emd-21225.cif.gz emd-21225.cif.gz | 5.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21225 http://ftp.pdbj.org/pub/emdb/structures/EMD-21225 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21225 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21225 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21225_validation.pdf.gz emd_21225_validation.pdf.gz | 415.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21225_full_validation.pdf.gz emd_21225_full_validation.pdf.gz | 414.9 KB | 表示 | |
| XML形式データ |  emd_21225_validation.xml.gz emd_21225_validation.xml.gz | 13.8 KB | 表示 | |
| CIF形式データ |  emd_21225_validation.cif.gz emd_21225_validation.cif.gz | 18.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21225 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21225 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21225 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21225 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6vk9MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10797 (タイトル: Cryo-EM structure of PilA-N/C from Geobacter sulfurreducens EMPIAR-10797 (タイトル: Cryo-EM structure of PilA-N/C from Geobacter sulfurreducensData size: 1.5 TB Data #1: Geobacter sulfurreducens PilA-N-C pili - raw unaligned/uncorrected super resolution images [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21225.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21225.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of PilA-N-C from Geobacter sulfurreducens | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.822 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Polymerized hetero-dimers of PilA-N and PilA-C
| 全体 | 名称: Polymerized hetero-dimers of PilA-N and PilA-C |
|---|---|
| 要素 |
|
-超分子 #1: Polymerized hetero-dimers of PilA-N and PilA-C
| 超分子 | 名称: Polymerized hetero-dimers of PilA-N and PilA-C / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
-分子 #1: Geopilin domain 1 protein
| 分子 | 名称: Geopilin domain 1 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 6.576502 KDa |
| 組換発現 | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 配列 | 文字列: FTLIELLIVV AIIGILAAIA IPQFSAYRVK AYNSAASSDL RNLKTALESA FADDQTYPPE S UniProtKB: Geopilin domain 1 protein |
-分子 #2: Geopilin domain 2 protein
| 分子 | 名称: Geopilin domain 2 protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 分子量 | 理論値: 10.973763 KDa |
| 組換発現 | 生物種:  Geobacter sulfurreducens (バクテリア) Geobacter sulfurreducens (バクテリア) |
| 配列 | 文字列: AGKIPTTTMG GKDFTFKPST NVSVSYFTTN GATSTAGTVN TDYAVNTKNS SGNRVFTSTN NTSNIWYIEN DAWKGKAVSD SDVTALGTG DVGKSDFSGT EWKSQ UniProtKB: Geopilin domain 2 protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.032 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 2-10 / 撮影したグリッド数: 1 / 実像数: 7664 / 平均露光時間: 0.2 sec. / 平均電子線量: 2.308 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6vk9: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)