+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21132 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of PCAT1 bound to its CtA peptide substrate | |||||||||
 マップデータ マップデータ | full map from cryosparc | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATP-Binding Cassette / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ABC-type bacteriocin transporter activity / ABC-type oligopeptide transporter activity / cysteine-type peptidase activity / ATP hydrolysis activity / proteolysis / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Hungateiclostridium thermocellum (バクテリア) / Hungateiclostridium thermocellum (バクテリア) /  Hungateiclostridium thermocellum (strain ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372) (バクテリア) Hungateiclostridium thermocellum (strain ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372) (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||
 データ登録者 データ登録者 | Kieuvongngam V / Oldham ML | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Structural basis of substrate recognition by a polypeptide processing and secretion transporter. 著者: Virapat Kieuvongngam / Paul Dominic B Olinares / Anthony Palillo / Michael L Oldham / Brian T Chait / Jue Chen /  要旨: The peptidase-containing ATP-binding cassette transporters (PCATs) are unique members of the ABC transporter family that proteolytically process and export peptides and proteins. Each PCAT contains ...The peptidase-containing ATP-binding cassette transporters (PCATs) are unique members of the ABC transporter family that proteolytically process and export peptides and proteins. Each PCAT contains two peptidase domains that cleave off the secretion signal, two transmembrane domains forming a translocation pathway, and two nucleotide-binding domains that hydrolyze ATP. Previously the crystal structures of a PCAT from (PCAT1) were determined in the absence and presence of ATP, revealing how ATP binding regulates the protease activity and access to the translocation pathway. However, how the substrate CtA, a 90-residue polypeptide, is recognized by PCAT1 remained elusive. To address this question, we determined the structure of the PCAT1-CtA complex by electron cryo-microscopy (cryo-EM) to 3.4 Å resolution. The structure shows that two CtAs are bound via their N-terminal leader peptides, but only one is positioned for cleavage and translocation. Based on these results, we propose a model of how substrate cleavage, ATP hydrolysis, and substrate translocation are coordinated in a transport cycle. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21132.map.gz emd_21132.map.gz | 52 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21132-v30.xml emd-21132-v30.xml emd-21132.xml emd-21132.xml | 22.5 KB 22.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21132.png emd_21132.png | 113.1 KB | ||
| Filedesc metadata |  emd-21132.cif.gz emd-21132.cif.gz | 6.7 KB | ||
| その他 |  emd_21132_additional.map.gz emd_21132_additional.map.gz emd_21132_half_map_1.map.gz emd_21132_half_map_1.map.gz emd_21132_half_map_2.map.gz emd_21132_half_map_2.map.gz | 96.9 MB 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21132 http://ftp.pdbj.org/pub/emdb/structures/EMD-21132 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21132 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21132 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6v9zMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10818 (タイトル: Cryo-electron microscopy reconstruction of PCAT1 bound to its CtA peptide substrate EMPIAR-10818 (タイトル: Cryo-electron microscopy reconstruction of PCAT1 bound to its CtA peptide substrateData size: 1.8 TB Data #1: Unaligned and uncorrected multiframe movies of PCAT1 bound to its CtA peptide substrate [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21132.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21132.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full map from cryosparc | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
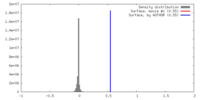
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: sharpened map (bfactor -75 Angstrom^2) from cryosparc
| ファイル | emd_21132_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map (bfactor -75 Angstrom^2) from cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
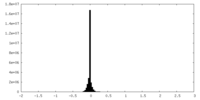
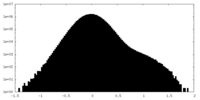
| 密度ヒストグラム |
-ハーフマップ: half map2 from cryosparc
| ファイル | emd_21132_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map2 from cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
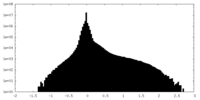
| 密度ヒストグラム |
-ハーフマップ: half map1 from cryosparc
| ファイル | emd_21132_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map1 from cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
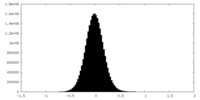
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of a homodimeric PCAT1 ABC transporter with two c...
| 全体 | 名称: Ternary complex of a homodimeric PCAT1 ABC transporter with two copies of bound peptide substrate |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of a homodimeric PCAT1 ABC transporter with two c...
| 超分子 | 名称: Ternary complex of a homodimeric PCAT1 ABC transporter with two copies of bound peptide substrate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Hungateiclostridium thermocellum (バクテリア) Hungateiclostridium thermocellum (バクテリア) |
| 分子量 | 理論値: 182.558 KDa |
-分子 #1: ABC-type bacteriocin transporter
| 分子 | 名称: ABC-type bacteriocin transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hungateiclostridium thermocellum (strain ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372) (バクテリア) Hungateiclostridium thermocellum (strain ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372) (バクテリア)株: ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372 |
| 分子量 | 理論値: 81.148742 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMLRRLFK KKYVCVRQYD LTDAGAACLS SIAQYYGLKM SLAKIREMTG TDTQGTNAYG LIHAAKQLGF SAKGVKASKE DLLKDFRLP AIANVIVDNR LAHFVVIYSI KNRIITVADP GKGIVRYSMD DFCSIWTGGL VLLEPGEAFQ KGDYTQNMMV K FAGFLKPL ...文字列: SNAMLRRLFK KKYVCVRQYD LTDAGAACLS SIAQYYGLKM SLAKIREMTG TDTQGTNAYG LIHAAKQLGF SAKGVKASKE DLLKDFRLP AIANVIVDNR LAHFVVIYSI KNRIITVADP GKGIVRYSMD DFCSIWTGGL VLLEPGEAFQ KGDYTQNMMV K FAGFLKPL KKTVLCIFLA SLLYTALGIA GSFYIKFLFD DLIKFEKLND LHIISAGFAV IFLLQIFLNY YRSILVTKLG MS IDKSIMM EYYSHVLKLP MNFFNSRKVG EIISRFMDAS KIRQAISGAT LTIMIDTIMA VIGGILLYIQ NSSLFFISFI IIL LYGIIV TVFNKPIQNA NRQIMEDNAK LTSALVESVK GIETIKSFGA EEQTEKSTRD KIETVMKSSF KEGMLYINLS SLTG IVAGL GGIVILWAGA YNVIKGNMSG GQLLAFNALL AYFLTPVKNL IDLQPLIQTA VVASNRLGEI LELATEKELR EDSDD FVIS LKGDIEFRNV DFRYGLRKPV LKNINLTIPK GKTVAIVGES GSGKTTLAKL LMNFYSPEKG DILINGHSIK NISLEL IRK KIAFVSQDVF IFSGTVKENL CLGNENVDMD EIIKAAKMAN AHDFIEKLPL KYDTFLNESG ANLSEGQKQR LAIARAL LK KPDILILDEA TSNLDSITEN HIKDAIYGLE DDVTVIIIAH RLSTIVNCDK IYLLKDGEIV ESGSHTELIA LKGCYFKM W KQTENTLAS UniProtKB: ABC-type bacteriocin transporter |
-分子 #2: CtA
| 分子 | 名称: CtA / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hungateiclostridium thermocellum (strain ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372) (バクテリア) Hungateiclostridium thermocellum (strain ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372) (バクテリア)株: ATCC 27405 / DSM 1237 / JCM 9322 / NBRC 103400 / NCIMB 10682 / NRRL B-4536 / VPI 7372 |
| 分子量 | 理論値: 10.217867 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMSEAKKL NIGRELTDEE LMEMTGGSTF SIQCQKDYTY KPSLPVVKYG VVIDEPEVVI KYGVGPIVGI KYGVEPIGPI QPMYGIKPV ETLK UniProtKB: Bacteriocin-type signal sequence-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| ||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR | ||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 100.0 K |
| 特殊光学系 | 球面収差補正装置: Microscope was modified with a Cs corrector エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-60 / 撮影したグリッド数: 2 / 実像数: 3478 / 平均露光時間: 0.2 sec. / 平均電子線量: 1.33 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.0 mm / 最大 デフォーカス(公称値): 2.2 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)