+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20614 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Vesicular Stomatitis Virus L Protein in Complex with Its Phosphoprotein Cofactor (3.0 A resolution) | |||||||||
 マップデータ マップデータ | filtered, sharpened | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Vesicular stomatitis virus / RNA-dependent RNA polymerase / L protein / P protein / phosphoprotein / single particle analysis / transcription / replication / virus / viral / RdRp / PRNTase / nonsegmented negative-sense RNA viruses / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NNS virus cap methyltransferase / RNA folding chaperone / GDP polyribonucleotidyltransferase / negative stranded viral RNA replication / viral transcription / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / phosphorylation / viral genome replication / virion component / mRNA 5'-cap (guanine-N7-)-methyltransferase activity ...NNS virus cap methyltransferase / RNA folding chaperone / GDP polyribonucleotidyltransferase / negative stranded viral RNA replication / viral transcription / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / phosphorylation / viral genome replication / virion component / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / host cell cytoplasm / RNA-directed RNA polymerase / RNA-dependent RNA polymerase activity / GTPase activity / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Vesicular stomatitis virus (ウイルス) / Vesicular stomatitis virus (ウイルス) /  Vesicular stomatitis Indiana virus (strain San Juan) (ウイルス) Vesicular stomatitis Indiana virus (strain San Juan) (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Jenni S / Bloyet LM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2020 ジャーナル: Cell Rep / 年: 2020タイトル: Structure of the Vesicular Stomatitis Virus L Protein in Complex with Its Phosphoprotein Cofactor. 著者: Simon Jenni / Louis-Marie Bloyet / Ruben Diaz-Avalos / Bo Liang / Sean P J Whelan / Nikolaus Grigorieff / Stephen C Harrison /  要旨: The large (L) proteins of non-segmented, negative-strand RNA viruses are multifunctional enzymes that produce capped, methylated, and polyadenylated mRNA and replicate the viral genome. A ...The large (L) proteins of non-segmented, negative-strand RNA viruses are multifunctional enzymes that produce capped, methylated, and polyadenylated mRNA and replicate the viral genome. A phosphoprotein (P), required for efficient RNA-dependent RNA polymerization from the viral ribonucleoprotein (RNP) template, regulates the function and conformation of the L protein. We report the structure of vesicular stomatitis virus L in complex with its P cofactor determined by electron cryomicroscopy at 3.0 Å resolution, enabling us to visualize bound segments of P. The contacts of three P segments with multiple L domains show how P induces a closed, compact, initiation-competent conformation. Binding of P to L positions its N-terminal domain adjacent to a putative RNA exit channel for efficient encapsidation of newly synthesized genomes with the nucleoprotein and orients its C-terminal domain to interact with an RNP template. The model shows that a conserved tryptophan in the priming loop can support the initiating 5' nucleotide. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20614.map.gz emd_20614.map.gz | 226.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20614-v30.xml emd-20614-v30.xml emd-20614.xml emd-20614.xml | 27.5 KB 27.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
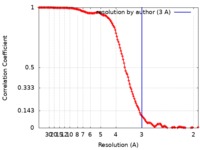
| FSC (解像度算出) |  emd_20614_fsc.xml emd_20614_fsc.xml | 16.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20614.png emd_20614.png | 169.6 KB | ||
| マスクデータ |  emd_20614_msk_1.map emd_20614_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-20614.cif.gz emd-20614.cif.gz | 7.9 KB | ||
| その他 |  emd_20614_additional_1.map.gz emd_20614_additional_1.map.gz emd_20614_additional_2.map.gz emd_20614_additional_2.map.gz emd_20614_additional_3.map.gz emd_20614_additional_3.map.gz emd_20614_half_map_1.map.gz emd_20614_half_map_1.map.gz emd_20614_half_map_2.map.gz emd_20614_half_map_2.map.gz | 226.7 MB 963.3 MB 33.1 MB 8.8 MB 8.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20614 http://ftp.pdbj.org/pub/emdb/structures/EMD-20614 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20614 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20614 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20614_validation.pdf.gz emd_20614_validation.pdf.gz | 872.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20614_full_validation.pdf.gz emd_20614_full_validation.pdf.gz | 872.1 KB | 表示 | |
| XML形式データ |  emd_20614_validation.xml.gz emd_20614_validation.xml.gz | 21.9 KB | 表示 | |
| CIF形式データ |  emd_20614_validation.cif.gz emd_20614_validation.cif.gz | 28.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20614 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20614 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20614 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20614 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20614.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20614.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | filtered, sharpened | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.97 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20614_msk_1.map emd_20614_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: filtered
| ファイル | emd_20614_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | filtered | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: filtered, sharpened, calculated on fine grid
| ファイル | emd_20614_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | filtered, sharpened, calculated on fine grid | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: filtered, sharpened, calculated on fine grid, boxed
| ファイル | emd_20614_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | filtered, sharpened, calculated on fine grid, boxed | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map1, unmodified
| ファイル | emd_20614_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map1, unmodified | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map2, unmodified
| ファイル | emd_20614_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map2, unmodified | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : VSV L:P
| 全体 | 名称: VSV L:P |
|---|---|
| 要素 |
|
-超分子 #1: VSV L:P
| 超分子 | 名称: VSV L:P / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: L protein (full-length) in complex with P protein (35-106) |
|---|---|
| 由来(天然) | 生物種:  Vesicular stomatitis virus (ウイルス) Vesicular stomatitis virus (ウイルス) |
| 分子量 | 理論値: 240 KDa |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Vesicular stomatitis Indiana virus (strain San Juan) (ウイルス) Vesicular stomatitis Indiana virus (strain San Juan) (ウイルス)株: San Juan |
| 分子量 | 理論値: 241.313859 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEVHDFETDE FNDFNEDDYA TREFLNPDER MTYLNHADYN LNSPLISDDI DNLIRKFNSL PIPSMWDSKN WDGVLEMLTS CQANPISTS QMHKWMGSWL MSDNHDASQG YSFLHEVDKE AEITFDVVET FIRGWGNKPI EYIKKERWTD SFKILAYLCQ K FLDLHKLT ...文字列: MEVHDFETDE FNDFNEDDYA TREFLNPDER MTYLNHADYN LNSPLISDDI DNLIRKFNSL PIPSMWDSKN WDGVLEMLTS CQANPISTS QMHKWMGSWL MSDNHDASQG YSFLHEVDKE AEITFDVVET FIRGWGNKPI EYIKKERWTD SFKILAYLCQ K FLDLHKLT LILNAVSEVE LLNLARTFKG KVRRSSHGTN ICRIRVPSLG PTFISEGWAY FKKLDILMDR NFLLMVKDVI IG RMQTVLS MVCRIDNLFS EQDIFSLLNI YRIGDKIVER QGNFSYDLIK MVEPICNLKL MKLARESRPL VPQFPHFENH IKT SVDEGA KIDRGIRFLH DQIMSVKTVD LTLVIYGSFR HWGHPFIDYY TGLEKLHSQV TMKKDIDVSY AKALASDLAR IVLF QQFND HKKWFVNGDL LPHDHPFKSH VKENTWPTAA QVQDFGDKWH ELPLIKCFEI PDLLDPSIIY SDKSHSMNRS EVLKH VRMN PNTPIPSKKV LQTMLDTKAT NWKEFLKEID EKGLDDDDLI IGLKGKEREL KLAGRFFSLM SWKLREYFVI TEYLIK THF VPMFKGLTMA DDLTAVIKKM LDSSSGQGLK SYEAICIANH IDYEKWNNHQ RKLSNGPVFR VMGQFLGYPS LIERTHE FF EKSLIYYNGR PDLMRVHNNT LINSTSQRVC WQGQEGGLEG LRQKGWTILN LLVIQREAKI RNTAVKVLAQ GDNQVICT Q YKTKKSRNVV ELQGALNQMV SNNEKIMTAI KIGTGKLGLL INDDETMQSA DYLNYGKIPI FRGVIRGLET KRWSRVTCV TNDQIPTCAN IMSSVSTNAL TVAHFAENPI NAMIQYNYFG TFARLLLMMH DPALRQSLYE VQDKIPGLHS STFKYAMLYL DPSIGGVSG MSLSRFLIRA FPDPVTESLS FWRFIHVHAR SEHLKEMSAV FGNPEIAKFR ITHIDKLVED PTSLNIAMGM S PANLLKTE VKKCLIESRQ TIRNQVIKDA TIYLYHEEDR LRSFLWSINP LFPRFLSEFK SGTFLGVADG LISLFQNSRT IR NSFKKKY HRELDDLIVR SEVSSLTHLG KLHLRRGSCK MWTCSATHAD TLRYKSWGRT VIGTTVPHPL EMLGPQHRKE TPC APCNTS GFNYVSVHCP DGIHDVFSSR GPLPAYLGSK TSESTSILQP WERESKVPLI KRATRLRDAI SWFVEPDSKL AMTI LSNIH SLTGEEWTKR QHGFKRTGSA LHRFSTSRMS HGGFASQSTA ALTRLMATTD TMRDLGDQNF DFLFQATLLY AQITT TVAR DGWITSCTDH YHIACKSCLR PIEEITLDSS MDYTPPDVSH VLKTWRNGEG SWGQEIKQIY PLEGNWKNLA PAEQSY QVG RCIGFLYGDL AYRKSTHAED SSLFPLSIQG RIRGRGFLKG LLDGLMRASC CQVIHRRSLA HLKRPANAVY GGLIYLI DK LSVSPPFLSL TRSGPIRDEL ETIPHKIPTS YPTSNRDMGV IVRNYFKYQC RLIEKGKYRS HYSQLWLFSD VLSIDFIG P FSISTTLLQI LYKPFLSGKD KNELRELANL SSLLRSGEGW EDIHVKFFTK DILLCPEEIR HACKFGIAKD NNKDMSYPP WGRESRGTIT TIPVYYTTTP YPKMLEMPPR IQNPLLSGIR LGQLPTGAHY KIRSILHGMG IHYRDFLSCG DGSGGMTAAL LRENVHSRG IFNSLLELSG SVMRGASPEP PSALETLGGD KSRCVNGETC WEYPSDLCDP RTWDYFLRLK AGLGLQIDLI V MDMEVRDS STSLKIETNV RNYVHRILDE QGVLIYKTYG TYICESEKNA VTILGPMFKT VDLVQTEFSS SQTSEVYMVC KG LKKLIDE PNPDWSSINE SWKNLYAFQS SEQEFARAKK VSTYFTLTGI PSQFIPDPFV NIETMLQIFG VPTGVSHAAA LKS SDRPAD LLTISLFYMA IISYYNINHI RVGPIPPNPP SDGIAQNVGI AITGISFWLS LMEKDIPLYQ QCLAVIQQSF PIRW EAVSV KGGYKQKWST RGDGLPKDTR ISDSLAPIGN WIRSLELVRN QVRLNPFNEI LFNQLCRTVD NHLKWSNLRR NTGMI EWIN RRISKEDRSI LMLKSDLHEE NSWRD UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: Phosphoprotein
| 分子 | 名称: Phosphoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vesicular stomatitis Indiana virus (strain San Juan) (ウイルス) Vesicular stomatitis Indiana virus (strain San Juan) (ウイルス)株: San Juan |
| 分子量 | 理論値: 29.941982 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDNLTKVREY LKSYSRLDQA VGEIDEIEAQ RAEKSNYELF QEDGVEEHTK PSYFQAADDS DTESEPEIED NQGLYAQDPE AEQVEGFIQ GPLDDYADEE VDVVFTSDWK PPELESDEHG KTLRLTSPEG LSGEQKSQWL STIKAVVQSA KYWNLAECTF E ASGEGVIM ...文字列: MDNLTKVREY LKSYSRLDQA VGEIDEIEAQ RAEKSNYELF QEDGVEEHTK PSYFQAADDS DTESEPEIED NQGLYAQDPE AEQVEGFIQ GPLDDYADEE VDVVFTSDWK PPELESDEHG KTLRLTSPEG LSGEQKSQWL STIKAVVQSA KYWNLAECTF E ASGEGVIM KERQITPDVY KVTPVMNTHP SQSEAVSDVW SLSKTSMTFQ PKKASLQPLT ISLDELFSSR GEFISVGGDG RM SHKEAIL LGLRYKKLYN QARVKYSL UniProtKB: Phosphoprotein |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 3 / 実像数: 3307 / 平均露光時間: 5.0 sec. / 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 35-2109 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 70.6 / 当てはまり具合の基準: CC + restraints |
| 得られたモデル |  PDB-6u1x: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)