+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1663 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Model of the Vesicular Stomatitis Virus | |||||||||
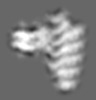
 マップデータ マップデータ | This is one octant of the volume of the top view of the VSV virion trunk. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NSRV / VSV / CryoEM / RNA / helix | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA replication / helical viral capsid / viral transcription / viral nucleocapsid / host cell cytoplasm / ribonucleoprotein complex / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Vesicular stomatitis virus (ウイルス) Vesicular stomatitis virus (ウイルス) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 10.6 Å | |||||||||
 データ登録者 データ登録者 | Ge P / Tsao J / Green TJ / Luo M / Zhou ZH | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2010 ジャーナル: Science / 年: 2010タイトル: Cryo-EM model of the bullet-shaped vesicular stomatitis virus. 著者: Peng Ge / Jun Tsao / Stan Schein / Todd J Green / Ming Luo / Z Hong Zhou /  要旨: Vesicular stomatitis virus (VSV) is a bullet-shaped rhabdovirus and a model system of negative-strand RNA viruses. Through direct visualization by means of cryo-electron microscopy, we show that each ...Vesicular stomatitis virus (VSV) is a bullet-shaped rhabdovirus and a model system of negative-strand RNA viruses. Through direct visualization by means of cryo-electron microscopy, we show that each virion contains two nested, left-handed helices: an outer helix of matrix protein M and an inner helix of nucleoprotein N and RNA. M has a hub domain with four contact sites that link to neighboring M and N subunits, providing rigidity by clamping adjacent turns of the nucleocapsid. Side-by-side interactions between neighboring N subunits are critical for the nucleocapsid to form a bullet shape, and structure-based mutagenesis results support this description. Together, our data suggest a mechanism of VSV assembly in which the nucleocapsid spirals from the tip to become the helical trunk, both subsequently framed and rigidified by the M layer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1663.map.gz emd_1663.map.gz | 72.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1663-v30.xml emd-1663-v30.xml emd-1663.xml emd-1663.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  Fig1.b.down.left-500.jpg Fig1.b.down.left-500.jpg | 131.8 KB | ||
| マスクデータ |  emd_1663_msk_1.map emd_1663_msk_1.map emd_1663_msk_2.map emd_1663_msk_2.map | 6.7 MB 5.5 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1663 http://ftp.pdbj.org/pub/emdb/structures/EMD-1663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1663 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1663_validation.pdf.gz emd_1663_validation.pdf.gz | 293.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1663_full_validation.pdf.gz emd_1663_full_validation.pdf.gz | 293 KB | 表示 | |
| XML形式データ |  emd_1663_validation.xml.gz emd_1663_validation.xml.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1663 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1663 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1663 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1663 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1663.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1663.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is one octant of the volume of the top view of the VSV virion trunk. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.532 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-セグメンテーションマップ: This is 5 consequetive N's in the nucleocapsid
| 注釈 | This is 5 consequetive N's in the nucleocapsid | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_1663_msk_1.map emd_1663_msk_1.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-セグメンテーションマップ: This is 2N with 5M in closest neighborhood of a central M-hub domain
| 注釈 | This is 2N with 5M in closest neighborhood of a central M-hub domain | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_1663_msk_2.map emd_1663_msk_2.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : VSV (Indiana)
| 全体 | 名称: VSV (Indiana) |
|---|---|
| 要素 |
|
-超分子 #1000: VSV (Indiana)
| 超分子 | 名称: VSV (Indiana) / タイプ: sample / ID: 1000 / 集合状態: Mature virion / Number unique components: 1 |
|---|
-超分子 #1: Vesicular stomatitis virus
| 超分子 | 名称: Vesicular stomatitis virus / タイプ: virus / ID: 1 / Name.synonym: VSV 詳細: Horse is the primary host species of the virus but Human is also possible. NCBI-ID: 11276 / 生物種: Vesicular stomatitis virus / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No / Syn species name: VSV |
|---|---|
| 宿主 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 100mM NaCl, 10nM Tris, 1mM EDTA |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: Cryo sample |
| グリッド | 詳細: Quantifoil 3.5/1 200 mesh |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 50 % / チャンバー内温度: 80 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: manual plunger. Vitrification carried out in open room 手法: manual blot 1 second before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 平均: 80 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 250,000 times magnification |
| 日付 | 2007年1月5日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC TVIPS / デジタル化 - サンプリング間隔: 1.532 µm / 実像数: 212 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 98000 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 10.6 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN / 詳細: Helicity determined and applied by IHRSR |
|---|
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid Body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Cross-correlation |
| 得られたモデル |  PDB-2wyy: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)