+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Pam3 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Lnt / apolipoprotein N-acyltransferase / bacterial lipoprotein / transferase / cryo-EM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報apolipoprotein N-acyltransferase / N-acyltransferase activity / lipoprotein biosynthetic process / outer membrane-bounded periplasmic space / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
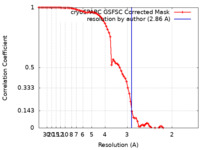
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.86 Å | |||||||||
 データ登録者 データ登録者 | Degtjarik O / Smithers L / Boland C / Caffrey M / Shalev Benami M | |||||||||
| 資金援助 |  アイルランド, 2件 アイルランド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Structure snapshots reveal the mechanism of a bacterial membrane lipoprotein -acyltransferase. 著者: Luke Smithers / Oksana Degtjarik / Dietmar Weichert / Chia-Ying Huang / Coilín Boland / Katherine Bowen / Abraham Oluwole / Corinne Lutomski / Carol V Robinson / Eoin M Scanlan / Meitian ...著者: Luke Smithers / Oksana Degtjarik / Dietmar Weichert / Chia-Ying Huang / Coilín Boland / Katherine Bowen / Abraham Oluwole / Corinne Lutomski / Carol V Robinson / Eoin M Scanlan / Meitian Wang / Vincent Olieric / Moran Shalev-Benami / Martin Caffrey /     要旨: Bacterial lipoproteins (BLPs) decorate the surface of membranes in the cell envelope. They function in membrane assembly and stability, as enzymes, and in transport. The final enzyme in the BLP ...Bacterial lipoproteins (BLPs) decorate the surface of membranes in the cell envelope. They function in membrane assembly and stability, as enzymes, and in transport. The final enzyme in the BLP synthesis pathway is the apolipoprotein -acyltransferase, Lnt, which is proposed to act by a ping-pong mechanism. Here, we use x-ray crystallography and cryo-electron microscopy to chart the structural changes undergone during the progress of the enzyme through the reaction. We identify a single active site that has evolved to bind, individually and sequentially, substrates that satisfy structural and chemical criteria to position reactive parts next to the catalytic triad for reaction. This study validates the ping-pong mechanism, explains the molecular bases for Lnt's substrate promiscuity, and should facilitate the design of antibiotics with minimal off-target effects. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
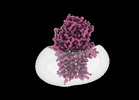
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15791.map.gz emd_15791.map.gz | 27.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15791-v30.xml emd-15791-v30.xml emd-15791.xml emd-15791.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15791_fsc.xml emd_15791_fsc.xml | 8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15791.png emd_15791.png | 48.7 KB | ||
| Filedesc metadata |  emd-15791.cif.gz emd-15791.cif.gz | 5.9 KB | ||
| その他 |  emd_15791_additional_1.map.gz emd_15791_additional_1.map.gz emd_15791_half_map_1.map.gz emd_15791_half_map_1.map.gz emd_15791_half_map_2.map.gz emd_15791_half_map_2.map.gz | 26.2 MB 48.9 MB 48.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15791 http://ftp.pdbj.org/pub/emdb/structures/EMD-15791 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15791 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15791 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15791_validation.pdf.gz emd_15791_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15791_full_validation.pdf.gz emd_15791_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_15791_validation.xml.gz emd_15791_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_15791_validation.cif.gz emd_15791_validation.cif.gz | 20 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15791 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15791 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15791 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15791 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8b0pMC  8aq2C  8aq3C  8aq4C  8b0kC  8b0lC  8b0mC  8b0nC  8b0oC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15791.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15791.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.826 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_15791_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
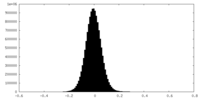
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15791_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
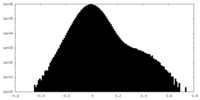
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15791_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Apolipoprotein N-acyltransferase
| 全体 | 名称: Apolipoprotein N-acyltransferase |
|---|---|
| 要素 |
|
-超分子 #1: Apolipoprotein N-acyltransferase
| 超分子 | 名称: Apolipoprotein N-acyltransferase / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Apolipoprotein N-acyltransferase
| 分子 | 名称: Apolipoprotein N-acyltransferase / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.248695 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MAFASLIERQ RIRLLLALLF GACGTLAFSP YDVWPAAIIS LMGLQALTFN RRPLQSAAIG FCWGFGLFG SGINWVYVSI ATFGGMPGPV NIFLVVLLAA YLSLYTGLFA GVLSRLWPKT TWLRVAIAAP ALWQVTEFLR G WVLTGFPW ...文字列: MGSSHHHHHH SSGLVPRGSH MAFASLIERQ RIRLLLALLF GACGTLAFSP YDVWPAAIIS LMGLQALTFN RRPLQSAAIG FCWGFGLFG SGINWVYVSI ATFGGMPGPV NIFLVVLLAA YLSLYTGLFA GVLSRLWPKT TWLRVAIAAP ALWQVTEFLR G WVLTGFPW LQFGYSQIDG PLKGLAPIMG VEAINFLLMM VSGLLALALV KRNWRPLVVA VVLFALPFPL RYIQWFTPQP EK TIQVSMV QGDIPQSLKW DEGQLLNTLK IYYNATAPLM GKSSLIIWPE SAITDLEINQ QPFLKALDGE LRDKGSSLVT GIV DARLNK QNRYDTYNTI ITLGKGAPYS YESADRYNKN HLVPFGEFVP LESILRPLAP FFDLPMSSFS RGPYIQPPLS ANGI ELTAA IAYEIILGEQ VRDNFRPDTD YLLTISNDAW FGKSIGPWQH FQMARMRALE LARPLLRSTN NGITAVIGPQ GEIQA MIPQ FTREVLTTNV TPTTGLTPYA RTGNWPLWVL TALFGFAAVL MSLRQRRK UniProtKB: Apolipoprotein N-acyltransferase |
-分子 #2: Pam3-SKKKK
| 分子 | 名称: Pam3-SKKKK / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 621.812 Da |
| 配列 | 文字列: SKKKK |
-分子 #3: [(2~{S})-3-[(2~{S})-3-azanyl-2-(hexadecanoylamino)-3-oxidanyliden...
| 分子 | 名称: [(2~{S})-3-[(2~{S})-3-azanyl-2-(hexadecanoylamino)-3-oxidanylidene-propyl]sulfanyl-2-hexadecanoyloxy-propyl] hexadecanoate タイプ: ligand / ID: 3 / コピー数: 1 / 式: IG7 |
|---|---|
| 分子量 | 理論値: 909.478 Da |
| Chemical component information | 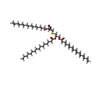 ChemComp-IG7: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 14 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6 構成要素:
| ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 33.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)