+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13882 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the ligand-free GPCR dimer Ste2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | mating-type factor pheromone receptor activity / GPCR fungal pheromone mating factor, STE2 / Pheromone alpha factor receptor, double transmembrane domain superfamily / Fungal pheromone mating factor STE2 GPCR / response to pheromone / membrane / Pheromone alpha factor receptor 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
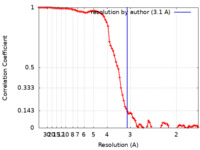
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Velazhahan V / Tate CG | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structure of the class D GPCR Ste2 dimer coupled to two G proteins. 著者: Vaithish Velazhahan / Ning Ma / Gáspár Pándy-Szekeres / Albert J Kooistra / Yang Lee / David E Gloriam / Nagarajan Vaidehi / Christopher G Tate /     要旨: G-protein-coupled receptors (GPCRs) are divided phylogenetically into six classes, denoted A to F. More than 370 structures of vertebrate GPCRs (belonging to classes A, B, C and F) have been ...G-protein-coupled receptors (GPCRs) are divided phylogenetically into six classes, denoted A to F. More than 370 structures of vertebrate GPCRs (belonging to classes A, B, C and F) have been determined, leading to a substantial understanding of their function. By contrast, there are no structures of class D GPCRs, which are found exclusively in fungi where they regulate survival and reproduction. Here we determine the structure of a class D GPCR, the Saccharomyces cerevisiae pheromone receptor Ste2, in an active state coupled to the heterotrimeric G protein Gpa1-Ste4-Ste18. Ste2 was purified as a homodimer coupled to two G proteins. The dimer interface of Ste2 is formed by the N terminus, the transmembrane helices H1, H2 and H7, and the first extracellular loop ECL1. We establish a class D1 generic residue numbering system (CD1) to enable comparisons with orthologues and with other GPCR classes. The structure of Ste2 bears similarities in overall topology to class A GPCRs, but the transmembrane helix H4 is shifted by more than 20 Å and the G-protein-binding site is a shallow groove rather than a cleft. The structure provides a template for the design of novel drugs to target fungal GPCRs, which could be used to treat numerous intractable fungal diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13882.map.gz emd_13882.map.gz | 56.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13882-v30.xml emd-13882-v30.xml emd-13882.xml emd-13882.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13882_fsc.xml emd_13882_fsc.xml | 8.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13882.png emd_13882.png | 58.3 KB | ||
| その他 |  emd_13882_half_map_1.map.gz emd_13882_half_map_1.map.gz emd_13882_half_map_2.map.gz emd_13882_half_map_2.map.gz | 55.3 MB 55.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13882 http://ftp.pdbj.org/pub/emdb/structures/EMD-13882 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13882 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13882 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13882_validation.pdf.gz emd_13882_validation.pdf.gz | 492.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13882_full_validation.pdf.gz emd_13882_full_validation.pdf.gz | 492 KB | 表示 | |
| XML形式データ |  emd_13882_validation.xml.gz emd_13882_validation.xml.gz | 15.8 KB | 表示 | |
| CIF形式データ |  emd_13882_validation.cif.gz emd_13882_validation.cif.gz | 20.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13882 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13882 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13882 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13882 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7qb9MC  7ad3C  7qa8C  7qbcC  7qbiC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10878 (タイトル: Structure of the ligand-free GPCR dimer Ste2 / Data size: 8.2 TB EMPIAR-10878 (タイトル: Structure of the ligand-free GPCR dimer Ste2 / Data size: 8.2 TBData #1: unaligned multiframe micrographs of Ste2 in the ligand-free state [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13882.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13882.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8466 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
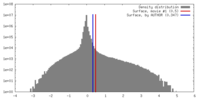
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_13882_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
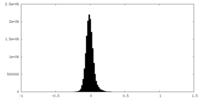
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13882_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
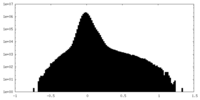
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of the ligand-free GPCR dimer Ste2
| 全体 | 名称: Structure of the ligand-free GPCR dimer Ste2 |
|---|---|
| 要素 |
|
-超分子 #1: Structure of the ligand-free GPCR dimer Ste2
| 超分子 | 名称: Structure of the ligand-free GPCR dimer Ste2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-分子 #1: Pheromone alpha factor receptor
| 分子 | 名称: Pheromone alpha factor receptor / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 47.885402 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSDAAPSLSN LFYDPTYNPG QSTINYTSIY GNGSTITFDE LQGLVNSTVT QAIMFGVRCG AAALTLIVMW MTSRSRKTPI FIINQVSLF LIILHSALYF KYLLSNYSSV TYALTGFPQF ISRGDVHVYG ATNIIQVLLV ASIETSLVFQ IKVIFTGDNF K RIGLMLTS ...文字列: MSDAAPSLSN LFYDPTYNPG QSTINYTSIY GNGSTITFDE LQGLVNSTVT QAIMFGVRCG AAALTLIVMW MTSRSRKTPI FIINQVSLF LIILHSALYF KYLLSNYSSV TYALTGFPQF ISRGDVHVYG ATNIIQVLLV ASIETSLVFQ IKVIFTGDNF K RIGLMLTS ISFTLGIATV TMYFVSAVKG MIVTYNDVSA TQDKYFNAST ILLASSINFM SFVLVVKLIL AIRSRRFLGL KQ FDSFHIL LIMSCQSLLV PSIIFILAYS LKPNQGTDVL TTVATLLAVL SLPLSSMWAT AANNASKTNT ITSDFTTSTD RFY PGTLSS FQTDSINNDA KSSLRSRLYD LYPRRKETTS DKHSERTFVS ETADDIEKNQ FYQLPTPTSS KNTRIGPFAD ASYK EGEVE PVDMYTPDTA ADEEARKFWT EDNNNL |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #3: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 3 / コピー数: 22 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)