+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13662 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the membrane soluble spike complex from the Lassa virus in a C3-symmetric map | |||||||||
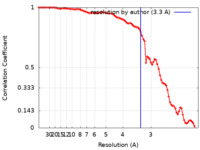
 マップデータ マップデータ | 3.3A low-pass filtered, C3-symmetric map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / receptor-mediated endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lassa virus Josiah (ウイルス) / Lassa virus Josiah (ウイルス) /  Lassa virus (strain Mouse/Sierra Leone/Josiah/1976) (ウイルス) Lassa virus (strain Mouse/Sierra Leone/Josiah/1976) (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Diskin R / Katz M | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Structure and receptor recognition by the Lassa virus spike complex. 著者: Michael Katz / Jonathan Weinstein / Maayan Eilon-Ashkenazy / Katrin Gehring / Hadas Cohen-Dvashi / Nadav Elad / Sarel J Fleishman / Ron Diskin /  要旨: Lassa virus (LASV) is a human pathogen, causing substantial morbidity and mortality. Similar to other Arenaviridae, it presents a class-I spike complex on its surface that facilitates cell entry. The ...Lassa virus (LASV) is a human pathogen, causing substantial morbidity and mortality. Similar to other Arenaviridae, it presents a class-I spike complex on its surface that facilitates cell entry. The virus's cellular receptor is matriglycan, a linear carbohydrate that is present on α-dystroglycan, but the molecular mechanism that LASV uses to recognize this glycan is unknown. In addition, LASV and other arenaviruses have a unique signal peptide that forms an integral and functionally important part of the mature spike; yet the structure, function and topology of the signal peptide in the membrane remain uncertain. Here we solve the structure of a complete native LASV spike complex, finding that the signal peptide crosses the membrane once and that its amino terminus is located in the extracellular region. Together with a double-sided domain-switching mechanism, the signal peptide helps to stabilize the spike complex in its native conformation. This structure reveals that the LASV spike complex is preloaded with matriglycan, suggesting the mechanism of binding and rationalizing receptor recognition by α-dystroglycan-tropic arenaviruses. This discovery further informs us about the mechanism of viral egress and may facilitate the rational design of novel therapeutics that exploit this binding site. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13662.map.gz emd_13662.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13662-v30.xml emd-13662-v30.xml emd-13662.xml emd-13662.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13662_fsc.xml emd_13662_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13662.png emd_13662.png | 104.6 KB | ||
| マスクデータ |  emd_13662_msk_1.map emd_13662_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13662_additional_1.map.gz emd_13662_additional_1.map.gz emd_13662_half_map_1.map.gz emd_13662_half_map_1.map.gz emd_13662_half_map_2.map.gz emd_13662_half_map_2.map.gz | 32.1 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13662 http://ftp.pdbj.org/pub/emdb/structures/EMD-13662 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13662 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13662 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13662_validation.pdf.gz emd_13662_validation.pdf.gz | 494.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13662_full_validation.pdf.gz emd_13662_full_validation.pdf.gz | 493.9 KB | 表示 | |
| XML形式データ |  emd_13662_validation.xml.gz emd_13662_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_13662_validation.cif.gz emd_13662_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13662 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13662 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13662 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13662 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13662.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13662.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3.3A low-pass filtered, C3-symmetric map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.038 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
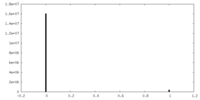
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13662_msk_1.map emd_13662_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
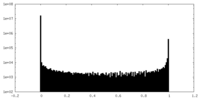
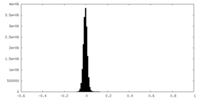
| 密度ヒストグラム |
-追加マップ: Unfiltered C3-symmetric map
| ファイル | emd_13662_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered C3-symmetric map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
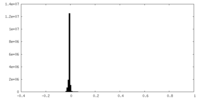
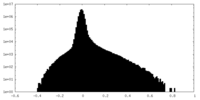
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_13662_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
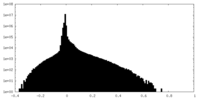
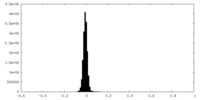
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_13662_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
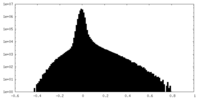
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The complete spike complex
| 全体 | 名称: The complete spike complex |
|---|---|
| 要素 |
|
-超分子 #1: The complete spike complex
| 超分子 | 名称: The complete spike complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Lassa virus Josiah (ウイルス) Lassa virus Josiah (ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Glycoprotein G2
| 分子 | 名称: Glycoprotein G2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa virus (strain Mouse/Sierra Leone/Josiah/1976) (ウイルス) Lassa virus (strain Mouse/Sierra Leone/Josiah/1976) (ウイルス)株: Mouse/Sierra Leone/Josiah/1976 |
| 分子量 | 理論値: 28.083273 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GTFTWTLSDS EGKDTPGGYC LTRWMLIEAE LKCFGNTAVA KCNEKHDEEF CDMLRLFDFN KQAIQRLKAE AQMSIQLINK AVNALINDQ LIMKNHLRDI MGIPYCNYSK YWYLNHTTTG RTSLPKCWLV SNGSYLNETH FSDDIEQQAD NMITEMLQKE Y MERQGKTP ...文字列: GTFTWTLSDS EGKDTPGGYC LTRWMLIEAE LKCFGNTAVA KCNEKHDEEF CDMLRLFDFN KQAIQRLKAE AQMSIQLINK AVNALINDQ LIMKNHLRDI MGIPYCNYSK YWYLNHTTTG RTSLPKCWLV SNGSYLNETH FSDDIEQQAD NMITEMLQKE Y MERQGKTP LGLVDLFVFS TSFYLISIFL HLVKIPTHRH IVGKSCPKPH RLNHMGICSC GLYKQPGVPV KWKRGGGSDY KD DDDK |
-分子 #2: Pre-glycoprotein polyprotein GP complex
| 分子 | 名称: Pre-glycoprotein polyprotein GP complex / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa virus (strain Mouse/Sierra Leone/Josiah/1976) (ウイルス) Lassa virus (strain Mouse/Sierra Leone/Josiah/1976) (ウイルス)株: Mouse/Sierra Leone/Josiah/1976 |
| 分子量 | 理論値: 29.064402 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGQIVTFFQE VPHVIEEVMN IVLIALSVLA VLKGLYNFAT CGLVGLVTFL LLCGRSCTTS LYKGVYELQT LELNMETLNM TMPLSCTKN NSHHYIMVGN ETGLELTLTN TSIINHKFCN LSDAHKKNLY DHALMSIIST FHLSIPNFNQ YEAMSCDFNG G KISVQYNL ...文字列: MGQIVTFFQE VPHVIEEVMN IVLIALSVLA VLKGLYNFAT CGLVGLVTFL LLCGRSCTTS LYKGVYELQT LELNMETLNM TMPLSCTKN NSHHYIMVGN ETGLELTLTN TSIINHKFCN LSDAHKKNLY DHALMSIIST FHLSIPNFNQ YEAMSCDFNG G KISVQYNL SHSYAGDAAN HCGTVANGVL QTFMRMAWGG SYIALDSGRG NWDCIMTSYQ YLIIQNTTWE DHCQFSRPSP IG YLGLLSQ RTRDIYISRR LL |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 15 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GRAPHENE OXIDE / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 73.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)