+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13581 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | human SLFN5 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | nucleotide binding protein antiviral linked to tumorigenesis transcription regulation / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.44 Å | |||||||||
 データ登録者 データ登録者 | Lammens K / Metzner FJ | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: Structural and biochemical characterization of human Schlafen 5. 著者: Felix J Metzner / Elisabeth Huber / Karl-Peter Hopfner / Katja Lammens /  要旨: The Schlafen family belongs to the interferon-stimulated genes and its members are involved in cell cycle regulation, T cell quiescence, inhibition of viral replication, DNA-repair and tRNA ...The Schlafen family belongs to the interferon-stimulated genes and its members are involved in cell cycle regulation, T cell quiescence, inhibition of viral replication, DNA-repair and tRNA processing. Here, we present the cryo-EM structure of full-length human Schlafen 5 (SLFN5) and the high-resolution crystal structure of the highly conserved N-terminal core domain. We show that the core domain does not resemble an ATPase-like fold and neither binds nor hydrolyzes ATP. SLFN5 binds tRNA as well as single- and double-stranded DNA, suggesting a potential role in transcriptional regulation. Unlike rat Slfn13 or human SLFN11, human SLFN5 did not cleave tRNA. Based on the structure, we identified two residues in proximity to the zinc finger motif that decreased DNA binding when mutated. These results indicate that Schlafen proteins have divergent enzymatic functions and provide a structural platform for future biochemical and genetic studies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13581.map.gz emd_13581.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13581-v30.xml emd-13581-v30.xml emd-13581.xml emd-13581.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13581.png emd_13581.png | 152.4 KB | ||
| Filedesc metadata |  emd-13581.cif.gz emd-13581.cif.gz | 5.6 KB | ||
| その他 |  emd_13581_half_map_1.map.gz emd_13581_half_map_1.map.gz emd_13581_half_map_2.map.gz emd_13581_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13581 http://ftp.pdbj.org/pub/emdb/structures/EMD-13581 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13581 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13581 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13581_validation.pdf.gz emd_13581_validation.pdf.gz | 747.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13581_full_validation.pdf.gz emd_13581_full_validation.pdf.gz | 747.1 KB | 表示 | |
| XML形式データ |  emd_13581_validation.xml.gz emd_13581_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_13581_validation.cif.gz emd_13581_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13581 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13581 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13581 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13581 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13581.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13581.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.046 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_13581_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
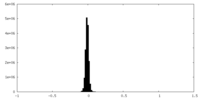
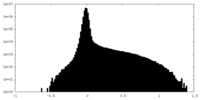
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13581_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
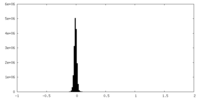
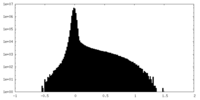
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : full length human Schlafen 5
| 全体 | 名称: full length human Schlafen 5 |
|---|---|
| 要素 |
|
-超分子 #1: full length human Schlafen 5
| 超分子 | 名称: full length human Schlafen 5 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Schlafen family member 5
| 分子 | 名称: Schlafen family member 5 / タイプ: protein_or_peptide / ID: 1 詳細: N-terminal 2 x Flag tag HRV 3C site flexible C-terminus コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 104.420016 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MADYKDDDDK GTDYKDDDDK LEVLFQGPMS LRIDVDTNFP ECVVDAGKVT LGTQQRQEMD PRLREKQNEI ILRAVCALLN SGGGIIKAE IENKGYNYER HGVGLDVPPI FRSHLDKMQK ENHFLIFVKS WNTEAGVPLA TLCSNLYHRE RTSTDVMDSQ E ALAFLKCR ...文字列: MADYKDDDDK GTDYKDDDDK LEVLFQGPMS LRIDVDTNFP ECVVDAGKVT LGTQQRQEMD PRLREKQNEI ILRAVCALLN SGGGIIKAE IENKGYNYER HGVGLDVPPI FRSHLDKMQK ENHFLIFVKS WNTEAGVPLA TLCSNLYHRE RTSTDVMDSQ E ALAFLKCR TQTPTNINVS NSLGPQAAQG SVQYEGNINV SAAALFDRKR LQYLEKLNLP ESTHVEFVMF STDVSHCVKD RL PKCVSAF ANTEGGYVFF GVHDETCQVI GCEKEKIDLT SLRASIDGCI KKLPVHHFCT QRPEIKYVLN FLEVHDKGAL RGY VCAIKV EKFCCAVFAK VPSSWQVKDN RVRQLPTREW TAWMMEADPD LSRCPEMVLQ LSLSSATPRS KPVCIHKNSE CLKE QQKRY FPVFSDRVVY TPESLYKELF SQHKGLRDLI NTEMRPFSQG ILIFSQSWAV DLGLQEKQGV ICDALLISQN NTPIL YTIF SKWDAGCKGY SMIVAYSLKQ KLVNKGGYTG RLCITPLVCV LNSDRKAQSV YSSYLQIYPE SYNFMTPQHM EALLQS LVI VLLGFKSFLS EELGSEVLNL LTNKQYELLS KNLRKTRELF VHGLPGSGKT ILALRIMEKI RNVFHCEPAN ILYICEN QP LKKLVSFSKK NICQPVTRKT FMKNNFEHIQ HIIIDDAQNF RTEDGDWYGK AKFITQTARD GPGVLWIFLD YFQTYHLS C SGLPPPSDQY PREEINRVVR NAGPIANYLQ QVMQEARQNP PPNLPPGSLV MLYEPKWAQG VPGNLEIIED LNLEEILIY VANKCRFLLR NGYSPKDIAV LFTKASEVEK YKDRLLTAMR KRKLSQLHEE SDLLLQIGDA SDVLTDHIVL DSVCRFSGLE RNIVFGINP GVAPPAGAYN LLLCLASRAK RHLYILKASV UniProtKB: Schlafen family member 5 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 9 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 41.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.44 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 140715 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)