+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12174 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
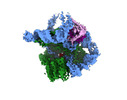
| タイトル | The catalytic core lobe of human telomerase in complex with a telomeric DNA substrate | |||||||||
 マップデータ マップデータ | Post-processed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Reverse transcriptase / ribonucleoprotein / complex / DNA / RNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of hair cycle / template-free RNA nucleotidyltransferase / positive regulation of transdifferentiation / TERT-RMRP complex / DNA strand elongation / RNA-directed RNA polymerase complex / siRNA transcription / positive regulation of protein localization to nucleolus / telomerase catalytic core complex / RNA-templated DNA biosynthetic process ...positive regulation of hair cycle / template-free RNA nucleotidyltransferase / positive regulation of transdifferentiation / TERT-RMRP complex / DNA strand elongation / RNA-directed RNA polymerase complex / siRNA transcription / positive regulation of protein localization to nucleolus / telomerase catalytic core complex / RNA-templated DNA biosynthetic process / : / establishment of protein localization to telomere / telomerase activity / nuclear telomere cap complex / siRNA processing / telomerase holoenzyme complex / positive regulation of vascular associated smooth muscle cell migration / telomerase RNA binding / DNA biosynthetic process / RNA-templated transcription / telomeric DNA binding / positive regulation of stem cell proliferation / mitochondrial nucleoid / negative regulation of cellular senescence / Telomere Extension By Telomerase / telomere maintenance via telomerase / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / replicative senescence / positive regulation of Wnt signaling pathway / positive regulation of G1/S transition of mitotic cell cycle / negative regulation of endothelial cell apoptotic process / response to cadmium ion / positive regulation of vascular associated smooth muscle cell proliferation / telomere maintenance / mitochondrion organization / positive regulation of nitric-oxide synthase activity / positive regulation of glucose import / Formation of the beta-catenin:TCF transactivating complex / regulation of protein stability / PML body / transcription coactivator binding / positive regulation of miRNA transcription / RNA-directed DNA polymerase / structural constituent of chromatin / positive regulation of angiogenesis / RNA-directed DNA polymerase activity / nucleosome / positive regulation of protein binding / protein-folding chaperone binding / cellular response to hypoxia / negative regulation of neuron apoptotic process / chromosome, telomeric region / tRNA binding / nuclear speck / protein heterodimerization activity / negative regulation of gene expression / RNA-dependent RNA polymerase activity / nucleolus / protein homodimerization activity / DNA binding / RNA binding / nucleoplasm / identical protein binding / nucleus / metal ion binding / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
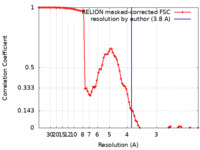
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Nguyen THD / Ghanim GE | |||||||||
| 資金援助 |  英国, 英国,  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structure of human telomerase holoenzyme with bound telomeric DNA. 著者: George E Ghanim / Adam J Fountain / Anne-Marie M van Roon / Ramya Rangan / Rhiju Das / Kathleen Collins / Thi Hoang Duong Nguyen /   要旨: Telomerase adds telomeric repeats at chromosome ends to compensate for the telomere loss that is caused by incomplete genome end replication. In humans, telomerase is upregulated during embryogenesis ...Telomerase adds telomeric repeats at chromosome ends to compensate for the telomere loss that is caused by incomplete genome end replication. In humans, telomerase is upregulated during embryogenesis and in cancers, and mutations that compromise the function of telomerase result in disease. A previous structure of human telomerase at a resolution of 8 Å revealed a vertebrate-specific composition and architecture, comprising a catalytic core that is flexibly tethered to an H and ACA (hereafter, H/ACA) box ribonucleoprotein (RNP) lobe by telomerase RNA. High-resolution structural information is necessary to develop treatments that can effectively modulate telomerase activity as a therapeutic approach against cancers and disease. Here we used cryo-electron microscopy to determine the structure of human telomerase holoenzyme bound to telomeric DNA at sub-4 Å resolution, which reveals crucial DNA- and RNA-binding interfaces in the active site of telomerase as well as the locations of mutations that alter telomerase activity. We identified a histone H2A-H2B dimer within the holoenzyme that was bound to an essential telomerase RNA motif, which suggests a role for histones in the folding and function of telomerase RNA. Furthermore, this structure of a eukaryotic H/ACA RNP reveals the molecular recognition of conserved RNA and protein motifs, as well as interactions that are crucial for understanding the molecular pathology of many mutations that cause disease. Our findings provide the structural details of the assembly and active site of human telomerase, which paves the way for the development of therapeutic agents that target this enzyme. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12174.map.gz emd_12174.map.gz | 48.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12174-v30.xml emd-12174-v30.xml emd-12174.xml emd-12174.xml | 26.6 KB 26.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12174_fsc.xml emd_12174_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12174.png emd_12174.png | 109.9 KB | ||
| Filedesc metadata |  emd-12174.cif.gz emd-12174.cif.gz | 8 KB | ||
| その他 |  emd_12174_half_map_1.map.gz emd_12174_half_map_1.map.gz emd_12174_half_map_2.map.gz emd_12174_half_map_2.map.gz | 40.7 MB 40.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12174 http://ftp.pdbj.org/pub/emdb/structures/EMD-12174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12174 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12174_validation.pdf.gz emd_12174_validation.pdf.gz | 949.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12174_full_validation.pdf.gz emd_12174_full_validation.pdf.gz | 949.4 KB | 表示 | |
| XML形式データ |  emd_12174_validation.xml.gz emd_12174_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_12174_validation.cif.gz emd_12174_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12174 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12174 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12174 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12174 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7bg9MC  7bgbC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10732 (タイトル: Structure of human telomerase holoenzyme with bound telomeric DNA EMPIAR-10732 (タイトル: Structure of human telomerase holoenzyme with bound telomeric DNAData size: 11.2 TB Data #1: Unaligned multiframe micrographs of human telomerase holoenzyme bound to a telomeric DNA [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12174.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12174.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_12174_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12174_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human telomerase catalytic core in complex with a telomeric DNA s...
| 全体 | 名称: Human telomerase catalytic core in complex with a telomeric DNA substrate |
|---|---|
| 要素 |
|
-超分子 #1: Human telomerase catalytic core in complex with a telomeric DNA s...
| 超分子 | 名称: Human telomerase catalytic core in complex with a telomeric DNA substrate タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|
-超分子 #2: Telomerase reverse transcriptase
| 超分子 | 名称: Telomerase reverse transcriptase / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1, #5 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Histone
| 超分子 | 名称: Histone / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #4: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Telomerase reverse transcriptase,Telomerase reverse transcriptase
| 分子 | 名称: Telomerase reverse transcriptase,Telomerase reverse transcriptase タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed DNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 器官: Kidney Homo sapiens (ヒト) / 器官: Kidney |
| 分子量 | 理論値: 149.158578 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKTAALAQHD EAVDNKFNKE QQNAFYEILH LPNLNEEQRN AFIQSLKDDP SQSANLLAEA KKLNDAQAPK VDNKFNKEQQ NAFYEILHL PNLNEEQRNA FIQSLKDDPS QSANLLAEAK KLNGAQAPKV DANSAGKSTD YDIPTTASEN LYFQGHKLGT F QGPWSHPQ ...文字列: MKTAALAQHD EAVDNKFNKE QQNAFYEILH LPNLNEEQRN AFIQSLKDDP SQSANLLAEA KKLNDAQAPK VDNKFNKEQQ NAFYEILHL PNLNEEQRNA FIQSLKDDPS QSANLLAEAK KLNGAQAPKV DANSAGKSTD YDIPTTASEN LYFQGHKLGT F QGPWSHPQ FEKGSAGSAA GSGAGWSHPQ FEKSRPTTAS GTMPRAPRCR AVRSLLRSHY REVLPLATFV RRLGPQGWRL VQ RGDPAAF RALVAQCLVC VPWDARPPPA APSFRQVSCL KELVARVLQR LCERGAKNVL AFGFALLDGA RGGPPEAFTT SVR SYLPNT VTDALRGSGA WGLLLRRVGD DVLVHLLARC ALFVLVAPSC AYQVCGPPLY QLGAATQARP PPHASGPRRR LGCE RAWNH SVREAGVPLG LPAPGARRRG GSASRSLPLP KRPRRGAAPE PERTPVGQGS WAHPGRTRGP SDRGFCVVSP ARPAE EATS LEGALSGTRH SHPSVGRQHH AGPPSTSRPP RPWDTPCPPV YAETKHFLYS SGDKEQLRPS FLLSSLRPSL TGARRL VET IFLGSRPWMP GTPRRLPRLP QRYWQMRPLF LELLGNHAQC PYGVLLKTHC PLRAAVTPAA GVCAREKPQG SVAAPEE ED TDPRRLVQLL RQHSSPWQVY GFVRACLRRL VPPGLWGSRH NERRFLRNTK KFISLGKHAK LSLQELTWKM SVRDCAWL R RSPGVGCVPA AEHRLREEIL AKFLHWLMSV YVVELLRSFF YVTETTFQKN RLFFYRKSVW SKLQSIGIRQ HLKRVQLRE LSEAEVRQHR EARPALLTSR LRFIPKPDGL RPIVNMDYVV GARTFRREKR AERLTSRVKA LFSVLNYERA RRPGLLGASV LGLDDIHRA WRTFVLRVRA QDPPPELYFV KVDVTGAYDT IPQDRLTEVI ASIIKPQNTY CVRRYAVVQK AAHGHVRKAF K SHVSTLTD LQPYMRQFVA HLQETSPLRD AVVIEQSSSL NEASSGLFDV FLRFMCHHAV RIRGKSYVQC QGIPQGSILS TL LCSLCYG DMENKLFAGI RRDGLLLRLV DDFLLVTPHL THAKTFLRTL VRGVPEYGCV VNLRKTVVNF PVEDEALGGT AFV QMPAHG LFPWCGLLLD TRTLEVQSDY SSYARTSIRA SLTFNRGFKA GRNMRRKLFG VLRLKCHSLF LDLQVNSLQT VCTN IYKIL LLQAYRFHAC VLQLPFHQQV WKNPTFFLRV ISDTASLCYS ILKAKNAGMS LGAKGAAGPL PSEAVQWLCH QAFLL KLTR HRVTYVPLLG SLRTAQTQLS RKLPGTTLTA LEAAANPALP SDFKTILD UniProtKB: Telomerase reverse transcriptase |
-分子 #3: Histone H2B
| 分子 | 名称: Histone H2B / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 器官: Kidney Homo sapiens (ヒト) / 器官: Kidney |
| 分子量 | 理論値: 18.074932 KDa |
| 配列 | 文字列: MPDPAKSAPA PKKGSKKAVT KVQKKDGKKR KRSRKESYSV YVYKVLKQVH PDTGISSKAM GIMNSFVNDI FERIAGEASR LAHYNKRST ITSREIQTAV RLLLPGELAK HAVSEGTKAV TKYTSSNPRN LSPTKPGGSE DRQPPPSQLS AIPPFCLVLR A GIAGQV UniProtKB: Histone H2B |
-分子 #4: Histone H2A
| 分子 | 名称: Histone H2A / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 器官: Kidney Homo sapiens (ヒト) / 器官: Kidney |
| 分子量 | 理論値: 14.140584 KDa |
| 配列 | 文字列: MSGRGKQGGK ARAKAKTRSS RAGLQFPVGR VRRLLRKGNY AERVGAGAPV YLAAVLEYLT AEILELAGNA ARDNKKTRII PRHLQLAIR NDEELNKLLG KVTIAQGGVL PNIQAVLLPK KTESHHKAKG K UniProtKB: Histone H2A |
-分子 #2: DNA (5'-D(P*TP*TP*AP*GP*GP*G)-3')
| 分子 | 名称: DNA (5'-D(P*TP*TP*AP*GP*GP*G)-3') / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 5.514567 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DA)(DG)(DG)(DG) |
-分子 #5: RNA (256-MER)
| 分子 | 名称: RNA (256-MER) / タイプ: rna / ID: 5 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 器官: Kidney Homo sapiens (ヒト) / 器官: Kidney |
| 分子量 | 理論値: 145.477797 KDa |
| 配列 | 文字列: GGGUUGCGGA GGGUGGGCCU GGGAGGGGUG GUGGCCAUUU UUUGUCUAAC CCUAACUGAG AAGGGCGUAG GCGCCGUGCU UUUGCUCCC CGCGCGCUGU UUUUCUCGCU GACUUUCAGC GGGCGGAAAA GCCUCGGCCU GCCGCCUUCC ACCGUUCAUU C UAGAGCAA ...文字列: GGGUUGCGGA GGGUGGGCCU GGGAGGGGUG GUGGCCAUUU UUUGUCUAAC CCUAACUGAG AAGGGCGUAG GCGCCGUGCU UUUGCUCCC CGCGCGCUGU UUUUCUCGCU GACUUUCAGC GGGCGGAAAA GCCUCGGCCU GCCGCCUUCC ACCGUUCAUU C UAGAGCAA ACAAAAAAUG UCAGCUGCUG GCCCGUUCGC CCCUCCCGGG GACCUGCGGC GGGUCGCCUG CCCAGCCCCC GA ACCCCGC CUGGAGGCCG CGGUCGGCCC GGGGCUUCUC CGGAGGCACC CACUGCCACC GCGAAGAGUU GGGCUCUGUC AGC CGCGGG UCUCUCGGGG GCGAGGGCGA GGUUCAGGCC UUUCAGGCCG CAGGAAGAGG AACGGAGCGA GUCCCCGCGC GCGG CGCGA UUCCCUGAGC UGUGGGACGU GCACCCAGGA CUCGGCUCAC ACAUGC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 5 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 12 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 4-5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 78.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 43639 / 平均露光時間: 1.0 sec. / 平均電子線量: 47.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 温度因子: 80 |
|---|---|
| 得られたモデル |  PDB-7bg9: |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X