+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12163 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Integrator cleavage module with focused classification on the INTS9/11 CTD | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報snRNA 3'-end processing / snRNA processing / integrator complex / regulation of transcription elongation by RNA polymerase II / 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ / RNA polymerase II transcribes snRNA genes / RNA endonuclease activity / negative regulation of transforming growth factor beta receptor signaling pathway / blood microparticle / nucleolus ...snRNA 3'-end processing / snRNA processing / integrator complex / regulation of transcription elongation by RNA polymerase II / 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ / RNA polymerase II transcribes snRNA genes / RNA endonuclease activity / negative regulation of transforming growth factor beta receptor signaling pathway / blood microparticle / nucleolus / nucleoplasm / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.5 Å | |||||||||
 データ登録者 データ登録者 | Pfleiderer MM / Galej WP | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Structure of the catalytic core of the Integrator complex. 著者: Moritz M Pfleiderer / Wojciech P Galej /  要旨: The Integrator is a specialized 3' end-processing complex involved in cleavage and transcription termination of a subset of nascent RNA polymerase II transcripts, including small nuclear RNAs (snRNAs) ...The Integrator is a specialized 3' end-processing complex involved in cleavage and transcription termination of a subset of nascent RNA polymerase II transcripts, including small nuclear RNAs (snRNAs). We provide evidence of the modular nature of the Integrator complex by biochemically characterizing its two subcomplexes, INTS5/8 and INTS10/13/14. Using cryoelectron microscopy (cryo-EM), we determined a 3.5-Å-resolution structure of the INTS4/9/11 ternary complex, which constitutes Integrator's catalytic core. Our structure reveals the spatial organization of the catalytic nuclease INTS11, bound to its catalytically impaired homolog INTS9 via several interdependent interfaces. INTS4, a helical repeat protein, plays a key role in stabilizing nuclease domains and other components. In this assembly, all three proteins form a composite electropositive groove, suggesting a putative RNA binding path within the complex. Comparison with other 3' end-processing machineries points to distinct features and a unique architecture of the Integrator's catalytic module. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12163.map.gz emd_12163.map.gz | 78 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12163-v30.xml emd-12163-v30.xml emd-12163.xml emd-12163.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_12163.png emd_12163.png | 87.6 KB | ||
| マスクデータ |  emd_12163_msk_1.map emd_12163_msk_1.map | 160.8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_12163_half_map_1.map.gz emd_12163_half_map_1.map.gz emd_12163_half_map_2.map.gz emd_12163_half_map_2.map.gz | 75.9 MB 75.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12163 http://ftp.pdbj.org/pub/emdb/structures/EMD-12163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12163 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12163_validation.pdf.gz emd_12163_validation.pdf.gz | 377.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12163_full_validation.pdf.gz emd_12163_full_validation.pdf.gz | 376.9 KB | 表示 | |
| XML形式データ |  emd_12163_validation.xml.gz emd_12163_validation.xml.gz | 12.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12163 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12163 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12163.map.gz / 形式: CCP4 / 大きさ: 160.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12163.map.gz / 形式: CCP4 / 大きさ: 160.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12163_msk_1.map emd_12163_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
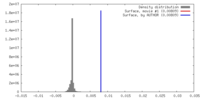
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12163_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
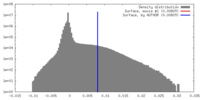
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_12163_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
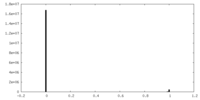
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Terneray complex of the Integrator cleavage module containing Int...
| 全体 | 名称: Terneray complex of the Integrator cleavage module containing Integrator subunit 4, 9 and 11 |
|---|---|
| 要素 |
|
-超分子 #1: Terneray complex of the Integrator cleavage module containing Int...
| 超分子 | 名称: Terneray complex of the Integrator cleavage module containing Integrator subunit 4, 9 and 11 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Integrator subunit 4
| 超分子 | 名称: Integrator subunit 4 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) / 組換株: Hi5 / 組換プラスミド: pBIG1a Trichoplusia ni (イラクサキンウワバ) / 組換株: Hi5 / 組換プラスミド: pBIG1a |
-超分子 #3: Integrator subunit 9
| 超分子 | 名称: Integrator subunit 9 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) / 組換株: Hi5 / 組換プラスミド: pBIG1a Trichoplusia ni (イラクサキンウワバ) / 組換株: Hi5 / 組換プラスミド: pBIG1a |
-超分子 #4: Integrator subunit 11
| 超分子 | 名称: Integrator subunit 11 / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) / 組換株: Hi5 / 組換プラスミド: pBIG1a Trichoplusia ni (イラクサキンウワバ) / 組換株: Hi5 / 組換プラスミド: pBIG1a |
-分子 #1: Integrator subunit 4
| 分子 | 名称: Integrator subunit 4 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MHHHHHHHHP PSGADPMAAH LKKRVYEEFT KVVQPQEEIA TKKLRLTKPS KSAALHIDLC KATSPADALQ YLLQFARKPV EAESVEGVVR ILLEHYYKEN DPSVRLKIAS LLGLLSKTAG FSPDCIMDDA INILQNEKSH QVLAQLLDTL LAIGTKLPEN QAIQMRLVDV ...文字列: MHHHHHHHHP PSGADPMAAH LKKRVYEEFT KVVQPQEEIA TKKLRLTKPS KSAALHIDLC KATSPADALQ YLLQFARKPV EAESVEGVVR ILLEHYYKEN DPSVRLKIAS LLGLLSKTAG FSPDCIMDDA INILQNEKSH QVLAQLLDTL LAIGTKLPEN QAIQMRLVDV ACKHLTDTSH GVRNKCLQL LGNLGSLEKS VTKDAEGLAA RDVQKIIGDY FSDQDPRVRT AAIKAMLQLH ERGLKLHQTI YNQACKLLSD DYEQVRSAAV QLIWVVSQLY PESIVPIPSS NEEIRLVDDA F GKICHMVS DGSWVVRVQA AKLLGSMEQV SSHFLEQTLD KKLMSDLRRK RTAHERAKEL YSSGEFSSGR KWGDDAPKEE VDTGAVNLIE SGACGAFVHG LEDEMYEVRI AAVEALCMLA QSSPSFAEKC LDFLVDMFND EIEEVRLQSI HTMRKISNNI TLREDQLDTV LAVLEDSSRD IREALHELLC CTNVSTKEGI HLALVELLKN LTKYPTDRDS IWKCLKFLGS RHPTLVLPLV PELLSTHPFF DTAEPDMDDP AYIAVLVLIF NAAKTCPTMP ALFSDHTFRH YAYLRDSLSH LVPALRLPGR KLVSSAVSPS IIPQEDPSQQ FLQQSLERVY SLQHLDPQGA QELLEFTIRD LQRLGELQSE LAGVADFSAT YLRCQLLLIK ALQEKLWNVA APLYLKQSDL ASAAAKQIME ETYKMEFMYS GVENKQVVII HHMRLQAKAL QLIVTARTTR GLDPLFGMCE KFLQEVDFFQ RYFIADLPHL QDSFVDKLLD LMPRLMTSKP AEVVKILQTM LRQSAFLHLP LPEQIHKASA TIIEPAGESD NPLRFTSGLV VALDVDATLE HVQDPQNTVK VQVLYPDGQA QMIHPKPADF RNPGPGRHRL ITQVYLSHTA WTEACQVEVR LLLAYNSSAR IPKCPWMEGG EMSPQVETSI EGTIPFSKPV KVYIMPKPAR R |
-分子 #2: Integrator subunit 9
| 分子 | 名称: Integrator subunit 9 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKLYCLSGHP TLPCNVLKFK STTIMLDCGL DMTSTLNFLP LPLVQSPRLS NLPGWSLKDG NAFLDKELKE CSGHVFVDSV PEFCLPETEL IDLSTVDVIL ISNYHCMMAL PYITEHTGFT GTVYATEPTV QIGRLLMEEL VNFIERVPKA QSASLWKNKD IQRLLPSPLK ...文字列: MKLYCLSGHP TLPCNVLKFK STTIMLDCGL DMTSTLNFLP LPLVQSPRLS NLPGWSLKDG NAFLDKELKE CSGHVFVDSV PEFCLPETEL IDLSTVDVIL ISNYHCMMAL PYITEHTGFT GTVYATEPTV QIGRLLMEEL VNFIERVPKA QSASLWKNKD IQRLLPSPLK DAVEVSTWRR CYTMQEVNSA LSKIQLVGYS QKIELFGAVQ VTPLSSGYAL GSSNWIIQSH YEKVSYVSGS SLLTTHPQPM DQASLKNSDV LVLTGLTQIP TANPDGMVGE FCSNLALTVR NGGNVLVPCY PSGVIYDLLE CLYQYIDSAG LSSVPLYFIS PVANSSLEFS QIFAEWLCHN KQSKVYLPEP PFPHAELIQT NKLKHYPSIH GDFSNDFRQP CVVFTGHPSL RFGDVVHFME LWGKSSLNTV IFTEPDFSYL EALAPYQPLA MKCIYCPIDT RLNFIQVSKL LKEVQPLHVV CPEQYTQPPP AQSHRMDLMI DCQPPAMSYR RAEVLALPFK RRYEKIEIMP ELADSLVPME IKPGISLATV SAVLHTKDNK HLLQPPPRPA QPTSGKKRKR VSDDVPDCKV LKPLLSGSIP VEQFVQTLEK HGFSDIKVED TAKGHIVLLQ EAETLIQIEE DSTHIICDND EMLRVRLRDL VLKFLQKF |
-分子 #3: Integrator subunit 11
| 分子 | 名称: Integrator subunit 11 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDEKTTGWRG GHVVEGLAGE LEQLRARLEH HPQGQREPPP SGADPMPEIR VTPLGAGQDV GRSCILVSIA GKNVMLDCGM HMGFNDDRRF PDFSYITQNG RLTDFLDCVI ISHFHLDHCG ALPYFSEMVG YDGPIYMTHP TQAICPILLE DYRKIAVDKK GEANFFTSQM ...文字列: MDEKTTGWRG GHVVEGLAGE LEQLRARLEH HPQGQREPPP SGADPMPEIR VTPLGAGQDV GRSCILVSIA GKNVMLDCGM HMGFNDDRRF PDFSYITQNG RLTDFLDCVI ISHFHLDHCG ALPYFSEMVG YDGPIYMTHP TQAICPILLE DYRKIAVDKK GEANFFTSQM IKDCMKKVVA VHLHQTVQVD DELEIKAYYA GHVLGAAMFQ IKVGSESVVY TGDYNMTPDR HLGAAWIDKC RPNLLITEST YATTIRDSKR CRERDFLKKV HETVERGGKV LIPVFALGRA QELCILLETF WERMNLKVPI YFSTGLTEKA NHYYKLFIPW TNQKIRKTFV QRNMFEFKHI KAFDRAFADN PGPMVVFATP GMLHAGQSLQ IFRKWAGNEK NMVIMPGYCV QGTVGHKILS GQRKLEMEGR QVLEVKMQVE YMSFSAHADA KGIMQLVGQA EPESVLLVHG EAKKMEFLKQ KIEQELRVNC YMPANGETVT LLTSPSIPVG ISLGLLKREM AQGLLPEAKK PRLLHGTLIM KDSNFRLVSS EQALKELGLA EHQLRFTCRV HLHDTRKEQE TALRVYSHLK SVLKDHCVQH LPDGSVTVES VLLQAAAPSE DPGTKVLLVS WTYQDEELGS FLTSLLKKGL PQAPS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 / 詳細: 150 mM KCl, 20 mM HEPES-KOH pH 7.8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 撮影したグリッド数: 2 / 実像数: 19268 / 平均露光時間: 5.0 sec. / 平均電子線量: 44.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.5 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 165000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: OTHER |
|---|
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)