+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11997 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a nanoparticle for a COVID-19 vaccine candidate | |||||||||
 マップデータ マップデータ | RBD Spy mi3 cage vlp | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SARS-CoV-2 / SpyTag / VLP / Receptor-binding domain / vaccine / COVID-19 / SpyCatcher / nanocage / icosahedral / nanoparticle / coronavirus / mi3 / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報D-galactonate catabolic process / 2-dehydro-3-deoxy-6-phosphogalactonate aldolase activity / cell adhesion / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Thermotoga maritima (strain ATCC 43589 / MSB8 / DSM 3109 / JCM 10099) (バクテリア) Thermotoga maritima (strain ATCC 43589 / MSB8 / DSM 3109 / JCM 10099) (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Duyvesteyn HME / Stuart DI | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: A COVID-19 vaccine candidate using SpyCatcher multimerization of the SARS-CoV-2 spike protein receptor-binding domain induces potent neutralising antibody responses. 著者: Tiong Kit Tan / Pramila Rijal / Rolle Rahikainen / Anthony H Keeble / Lisa Schimanski / Saira Hussain / Ruth Harvey / Jack W P Hayes / Jane C Edwards / Rebecca K McLean / Veronica Martini / ...著者: Tiong Kit Tan / Pramila Rijal / Rolle Rahikainen / Anthony H Keeble / Lisa Schimanski / Saira Hussain / Ruth Harvey / Jack W P Hayes / Jane C Edwards / Rebecca K McLean / Veronica Martini / Miriam Pedrera / Nazia Thakur / Carina Conceicao / Isabelle Dietrich / Holly Shelton / Anna Ludi / Ginette Wilsden / Clare Browning / Adrian K Zagrajek / Dagmara Bialy / Sushant Bhat / Phoebe Stevenson-Leggett / Philippa Hollinghurst / Matthew Tully / Katy Moffat / Chris Chiu / Ryan Waters / Ashley Gray / Mehreen Azhar / Valerie Mioulet / Joseph Newman / Amin S Asfor / Alison Burman / Sylvia Crossley / John A Hammond / Elma Tchilian / Bryan Charleston / Dalan Bailey / Tobias J Tuthill / Simon P Graham / Helen M E Duyvesteyn / Tomas Malinauskas / Jiandong Huo / Julia A Tree / Karen R Buttigieg / Raymond J Owens / Miles W Carroll / Rodney S Daniels / John W McCauley / David I Stuart / Kuan-Ying A Huang / Mark Howarth / Alain R Townsend /   要旨: There is need for effective and affordable vaccines against SARS-CoV-2 to tackle the ongoing pandemic. In this study, we describe a protein nanoparticle vaccine against SARS-CoV-2. The vaccine is ...There is need for effective and affordable vaccines against SARS-CoV-2 to tackle the ongoing pandemic. In this study, we describe a protein nanoparticle vaccine against SARS-CoV-2. The vaccine is based on the display of coronavirus spike glycoprotein receptor-binding domain (RBD) on a synthetic virus-like particle (VLP) platform, SpyCatcher003-mi3, using SpyTag/SpyCatcher technology. Low doses of RBD-SpyVLP in a prime-boost regimen induce a strong neutralising antibody response in mice and pigs that is superior to convalescent human sera. We evaluate antibody quality using ACE2 blocking and neutralisation of cell infection by pseudovirus or wild-type SARS-CoV-2. Using competition assays with a monoclonal antibody panel, we show that RBD-SpyVLP induces a polyclonal antibody response that recognises key epitopes on the RBD, reducing the likelihood of selecting neutralisation-escape mutants. Moreover, RBD-SpyVLP is thermostable and can be lyophilised without losing immunogenicity, to facilitate global distribution and reduce cold-chain dependence. The data suggests that RBD-SpyVLP provides strong potential to address clinical and logistic challenges of the COVID-19 pandemic. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11997.map.gz emd_11997.map.gz | 66.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11997-v30.xml emd-11997-v30.xml emd-11997.xml emd-11997.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11997.png emd_11997.png | 178.9 KB | ||
| Filedesc metadata |  emd-11997.cif.gz emd-11997.cif.gz | 7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11997 http://ftp.pdbj.org/pub/emdb/structures/EMD-11997 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11997 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11997 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11997.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11997.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RBD Spy mi3 cage vlp | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.63 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
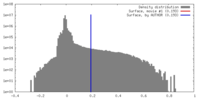
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : RBD-Spycatcher-mi3
| 全体 | 名称: RBD-Spycatcher-mi3 |
|---|---|
| 要素 |
|
-超分子 #1: RBD-Spycatcher-mi3
| 超分子 | 名称: RBD-Spycatcher-mi3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: Fibronectin binding protein,2-dehydro-3-deoxyphosphogluconate ald...
| 分子 | 名称: Fibronectin binding protein,2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase タイプ: protein_or_peptide / ID: 1 詳細: Model just contains mi3 cage component of VLP. Note that the Spy and RBD (spy tag and RBD are omitted from the sequence) are are not included in the model. The spycatcher003-mi3 can be found ...詳細: Model just contains mi3 cage component of VLP. Note that the Spy and RBD (spy tag and RBD are omitted from the sequence) are are not included in the model. The spycatcher003-mi3 can be found on genbank with code MT945417 and that of the SpyTag-RBD at MT945427.,Model just contains mi3 cage component of VLP. Note that the Spy and RBD (spy tag and RBD are omitted from the sequence) are are not included in the model. The spycatcher003-mi3 can be found on genbank with code MT945417 and that of the SpyTag-RBD at MT945427. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima (strain ATCC 43589 / MSB8 / DSM 3109 / JCM 10099) (バクテリア) Thermotoga maritima (strain ATCC 43589 / MSB8 / DSM 3109 / JCM 10099) (バクテリア)株: ATCC 43589 / MSB8 / DSM 3109 / JCM 10099 |
| 分子量 | 理論値: 36.000715 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSVTTLSG LSGEQGPSGD MTTEEDSATH IKFSKRDEDG RELAGATMEL RDSSGKTIST WISDGHVKDF YLYPGKYTFV ETAAPDGYE VATPIEFTVN EDGQVTVDGE ATEGDAHTGG SGGSGGSGGS MKMEELFKKH KIVAVLRANS VEEAKKKALA V FLGGVHLI ...文字列: MGSSVTTLSG LSGEQGPSGD MTTEEDSATH IKFSKRDEDG RELAGATMEL RDSSGKTIST WISDGHVKDF YLYPGKYTFV ETAAPDGYE VATPIEFTVN EDGQVTVDGE ATEGDAHTGG SGGSGGSGGS MKMEELFKKH KIVAVLRANS VEEAKKKALA V FLGGVHLI EITFTVPDAD TVIKELSFLK EMGAIIGAGT VTSVEQARKA VESGAEFIVS PHLDEEISQF AKEKGVFYMP GV MTPTELV KAMKLGHTIL KLFPGEVVGP QFVKAMKGPF PNVKFVPTGG VNLDNVCEWF KAGVLAVGVG SALVKGTPVE VAE KAKAFV EKIRGCTEGS GEPEA UniProtKB: Fibronectin binding protein, 2-dehydro-3-deoxyphosphogluconate aldolase/4-hydroxy-2-oxoglutarate aldolase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: PBS |
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 30 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 7393 / 平均電子線量: 48.1 e/Å2 詳細: SerialEM collection employing beam tilt for multishots per hole using custom script. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 165000 / 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)