+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11852 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bovine Papillomavirus E1 DNA helicase-replication fork complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA / virus / helicase / replisome / DNA replication. / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hydrolase activity, acting on acid anhydrides / DNA helicase activity / DNA replication / DNA helicase / host cell nucleus / ATP hydrolysis activity / DNA binding / ATP binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Bovine papillomavirus (パピローマウイルス) Bovine papillomavirus (パピローマウイルス) | |||||||||
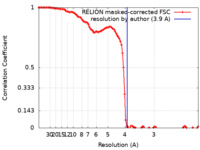
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Javed A / Major B | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Unwinding of a DNA replication fork by a hexameric viral helicase. 著者: Abid Javed / Balazs Major / Jonathan A Stead / Cyril M Sanders / Elena V Orlova /  要旨: Hexameric helicases are motor proteins that unwind double-stranded DNA (dsDNA) during DNA replication but how they are optimised for strand separation is unclear. Here we present the cryo-EM ...Hexameric helicases are motor proteins that unwind double-stranded DNA (dsDNA) during DNA replication but how they are optimised for strand separation is unclear. Here we present the cryo-EM structure of the full-length E1 helicase from papillomavirus, revealing all arms of a bound DNA replication fork and their interactions with the helicase. The replication fork junction is located at the entrance to the helicase collar ring, that sits above the AAA + motor assembly. dsDNA is escorted to and the 5´ single-stranded DNA (ssDNA) away from the unwinding point by the E1 dsDNA origin binding domains. The 3´ ssDNA interacts with six spirally-arranged β-hairpins and their cyclical top-to-bottom movement pulls the ssDNA through the helicase. Pulling of the RF against the collar ring separates the base-pairs, while modelling of the conformational cycle suggest an accompanying movement of the collar ring has an auxiliary role, helping to make efficient use of ATP in duplex unwinding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11852.map.gz emd_11852.map.gz | 64.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11852-v30.xml emd-11852-v30.xml emd-11852.xml emd-11852.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11852_fsc.xml emd_11852_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11852.png emd_11852.png | 134.4 KB | ||
| Filedesc metadata |  emd-11852.cif.gz emd-11852.cif.gz | 7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11852 http://ftp.pdbj.org/pub/emdb/structures/EMD-11852 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11852 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11852 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11852_validation.pdf.gz emd_11852_validation.pdf.gz | 385.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11852_full_validation.pdf.gz emd_11852_full_validation.pdf.gz | 385.2 KB | 表示 | |
| XML形式データ |  emd_11852_validation.xml.gz emd_11852_validation.xml.gz | 11.9 KB | 表示 | |
| CIF形式データ |  emd_11852_validation.cif.gz emd_11852_validation.cif.gz | 15.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11852 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11852 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11852 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11852 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11852.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11852.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.085 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : BPV E1 DNA helicase-replication fork complex
| 全体 | 名称: BPV E1 DNA helicase-replication fork complex |
|---|---|
| 要素 |
|
-超分子 #1: BPV E1 DNA helicase-replication fork complex
| 超分子 | 名称: BPV E1 DNA helicase-replication fork complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 413.4 KDa |
-超分子 #2: DNA replication fork
| 超分子 | 名称: DNA replication fork / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #3-#4 詳細: Consists of two strands and three regions: dsDNA, 5'ssDNA lagging strand and 3' ssDNA leading strand. |
|---|---|
| 由来(天然) | 生物種:  Bovine papillomavirus (パピローマウイルス) Bovine papillomavirus (パピローマウイルス) |
-超分子 #3: Full-length E1 helicase
| 超分子 | 名称: Full-length E1 helicase / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1-#2 詳細: Composed of six subunits; Two subunits contain the Origin Binding domains (Chains G, H), all six subunits contain the helicase domain and the C-terminal tail (Chains A-F). |
|---|---|
| 由来(天然) | 生物種:  Bovine papillomavirus (パピローマウイルス) Bovine papillomavirus (パピローマウイルス) |
-分子 #1: Replication protein E1
| 分子 | 名称: Replication protein E1 / タイプ: protein_or_peptide / ID: 1 / 詳細: OBD domains from subunits B and E. / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Bovine papillomavirus (パピローマウイルス) Bovine papillomavirus (パピローマウイルス) |
| 分子量 | 理論値: 17.162084 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSRATVFKLG LFKSLFLCSF HDITRLFKND KTTNQQWVLA VFGLAEVFFE ASFELLKKQC SFLQMQKRSH EGGTCAVYLI CFNTAKSRE TVRNLMANML NVREECLMLQ PPKIRGLSAA LFWFKSSLSP ATLKHGALPE WIRAQTTLNA AAA UniProtKB: Replication protein E1 |
-分子 #2: Replication protein E1
| 分子 | 名称: Replication protein E1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Bovine papillomavirus (パピローマウイルス) Bovine papillomavirus (パピローマウイルス) |
| 分子量 | 理論値: 33.859172 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TEKFDFGTMV QWAYDHKYAE ESKIAYEYAL AAGSDSNARA FLATNSQAKH VKDCATMVRH YLRAETQALS MPAYIKARCK LATGEGSWK SILTFFNYQN IELITFINAL KLWLKGIPKK NCLAFIGPPN TGKSMLCNSL IHFLGGSVLS FANHKSHFWL A SLADTRAA ...文字列: TEKFDFGTMV QWAYDHKYAE ESKIAYEYAL AAGSDSNARA FLATNSQAKH VKDCATMVRH YLRAETQALS MPAYIKARCK LATGEGSWK SILTFFNYQN IELITFINAL KLWLKGIPKK NCLAFIGPPN TGKSMLCNSL IHFLGGSVLS FANHKSHFWL A SLADTRAA LVDDATHACW RYFDTYLRNA LDGYPVSIDR KHKAAVQIKA PPLLVTSNID VQAEDRYLYL HSRVQTFRFE QP CTDESGE QPFNITDADW KSFFVRLWGR LDLIDEEEDS EEDGDSMRTF TCSARNTNAV D UniProtKB: Replication protein E1 |
-分子 #3: DNA (40-MER)
| 分子 | 名称: DNA (40-MER) / タイプ: dna / ID: 3 / 詳細: 5'-3' ssDNA strand of the DNA replication fork. / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Bovine papillomavirus (パピローマウイルス) Bovine papillomavirus (パピローマウイルス) |
| 分子量 | 理論値: 12.142779 KDa |
| 配列 | 文字列: (DT)(DG)(DT)(DA)(DT)(DT)(DT)(DC)(DA)(DC) (DA)(DC)(DC)(DG)(DC)(DA)(DC)(DC)(DT)(DC) (DA)(DG)(DC)(DG)(DC)(DG)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) |
-分子 #4: DNA (36-MER)
| 分子 | 名称: DNA (36-MER) / タイプ: dna / ID: 4 / 詳細: 3'-5' ssDNA strand of the DNA replication fork. / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Bovine papillomavirus (パピローマウイルス) Bovine papillomavirus (パピローマウイルス) |
| 分子量 | 理論値: 11.08009 KDa |
| 配列 | 文字列: (DC)(DC)(DC)(DC)(DC)(DC)(DC)(DG)(DT)(DG) (DC)(DG)(DC)(DG)(DC)(DT)(DG)(DA)(DG)(DG) (DT)(DG)(DC)(DG)(DG)(DT)(DG)(DT)(DG) (DA)(DA)(DA)(DT)(DA)(DC)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
| ||||||||||||
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV 詳細: 3 ul of sample was applied Lacey ultra-thin carbon film grids.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 95.0 K / 最高: 98.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 2 / 実像数: 12136 / 平均露光時間: 3.0 sec. / 平均電子線量: 50.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 47170 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー