+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11715 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
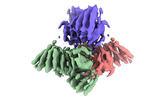
| タイトル | Cryo-EM reconstruction of the di-hexameric endocytic adaptor complex AENTH (ENTH F5A/L12A/V13A mutant) | |||||||||
 マップデータ マップデータ | final, sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Cargo recognition for clathrin-mediated endocytosis / actin cortical patch assembly / clathrin vesicle coat / clathrin light chain binding / negative regulation of Arp2/3 complex-mediated actin nucleation / actin cortical patch / incipient cellular bud site / cellular bud tip / clathrin coat assembly / clathrin adaptor activity ...Cargo recognition for clathrin-mediated endocytosis / actin cortical patch assembly / clathrin vesicle coat / clathrin light chain binding / negative regulation of Arp2/3 complex-mediated actin nucleation / actin cortical patch / incipient cellular bud site / cellular bud tip / clathrin coat assembly / clathrin adaptor activity / cellular bud neck / mating projection tip / phosphatidylinositol-3,4-bisphosphate binding / phosphatidylinositol-3,5-bisphosphate binding /  クラスリン / クラスリン /  clathrin binding / clathrin binding /  ubiquitin binding / actin filament organization / ubiquitin binding / actin filament organization /  phospholipid binding / phospholipid binding /  エンドサイトーシス / エンドサイトーシス /  actin filament binding / actin filament binding /  エンドソーム / エンドソーム /  エンドソーム / エンドソーム /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
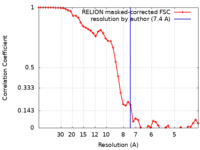
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.4 Å クライオ電子顕微鏡法 / 解像度: 7.4 Å | |||||||||
 データ登録者 データ登録者 | Klebl DP / Lizarrondo J / Sobott F / Garcia-Alai MM / Muench SP | |||||||||
| 資金援助 |  ドイツ, ドイツ,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure of the endocytic adaptor complex reveals the basis for efficient membrane anchoring during clathrin-mediated endocytosis. 著者: Javier Lizarrondo / David P Klebl / Stephan Niebling / Marc Abella / Martin A Schroer / Haydyn D T Mertens / Katharina Veith / Roland Thuenauer / Dmitri I Svergun / Michal Skruzny / Frank ...著者: Javier Lizarrondo / David P Klebl / Stephan Niebling / Marc Abella / Martin A Schroer / Haydyn D T Mertens / Katharina Veith / Roland Thuenauer / Dmitri I Svergun / Michal Skruzny / Frank Sobott / Stephen P Muench / Maria M Garcia-Alai /    要旨: During clathrin-mediated endocytosis, a complex and dynamic network of protein-membrane interactions cooperate to achieve membrane invagination. Throughout this process in yeast, endocytic coat ...During clathrin-mediated endocytosis, a complex and dynamic network of protein-membrane interactions cooperate to achieve membrane invagination. Throughout this process in yeast, endocytic coat adaptors, Sla2 and Ent1, must remain attached to the plasma membrane to transmit force from the actin cytoskeleton required for successful membrane invagination. Here, we present a cryo-EM structure of a 16-mer complex of the ANTH and ENTH membrane-binding domains from Sla2 and Ent1 bound to PIP that constitutes the anchor to the plasma membrane. Detailed in vitro and in vivo mutagenesis of the complex interfaces delineate the key interactions for complex formation and deficient cell growth phenotypes demonstrate its biological relevance. A hetero-tetrameric unit binds PIP molecules at the ANTH-ENTH interfaces and can form larger assemblies to contribute to membrane remodeling. Finally, a time-resolved small-angle X-ray scattering study of the interaction of these adaptor domains in vitro suggests that ANTH and ENTH domains have evolved to achieve a fast subsecond timescale assembly in the presence of PIP and do not require further proteins to form a stable complex. Together, these findings provide a molecular understanding of an essential piece in the molecular puzzle of clathrin-coated endocytic sites. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11715.map.gz emd_11715.map.gz | 1.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11715-v30.xml emd-11715-v30.xml emd-11715.xml emd-11715.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11715_fsc.xml emd_11715_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11715.png emd_11715.png | 141.7 KB | ||
| マスクデータ |  emd_11715_msk_1.map emd_11715_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11715_half_map_1.map.gz emd_11715_half_map_1.map.gz emd_11715_half_map_2.map.gz emd_11715_half_map_2.map.gz | 6 MB 6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11715 http://ftp.pdbj.org/pub/emdb/structures/EMD-11715 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11715 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11715 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11715.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11715.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final, sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.13 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11715_msk_1.map emd_11715_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11715_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11715_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 12 mer of 6 Sla2 ANTH and 6 Epsin-1 ENTH domains in complex with PIP2
| 全体 | 名称: 12 mer of 6 Sla2 ANTH and 6 Epsin-1 ENTH domains in complex with PIP2 |
|---|---|
| 要素 |
|
-超分子 #1: 12 mer of 6 Sla2 ANTH and 6 Epsin-1 ENTH domains in complex with PIP2
| 超分子 | 名称: 12 mer of 6 Sla2 ANTH and 6 Epsin-1 ENTH domains in complex with PIP2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
-分子 #1: ANTH domain of Sla-2
| 分子 | 名称: ANTH domain of Sla-2 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GAMGSMSRID SDLQKALKKA CSVEETAPKR KHVRACIVYT WDHQSSKAVF TTLKTLPLAN DEVQLFKMLI VLHKIIQEGH PSALAEAIRD RDWIRSLGRV HSGGSSYSKL IREYVRYLVL KLDFHAHHRG FNNGTFEYEE YVSLVSVSDP DEGYETILDL MSLQDSLDEF ...文字列: GAMGSMSRID SDLQKALKKA CSVEETAPKR KHVRACIVYT WDHQSSKAVF TTLKTLPLAN DEVQLFKMLI VLHKIIQEGH PSALAEAIRD RDWIRSLGRV HSGGSSYSKL IREYVRYLVL KLDFHAHHRG FNNGTFEYEE YVSLVSVSDP DEGYETILDL MSLQDSLDEF SQIIFASIQS ERRNTECKIS ALIPLIAESY GIYKFITSML RAMHRQLNDA EGDAALQPLK ERYELQHARL FEFYADCSSV KYLTTLVTIP KLPVDAPDVF LINDVDESKE IKFKKREPSV T |
-分子 #2: ENTH domain of Epsin-1
| 分子 | 名称: ENTH domain of Epsin-1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GAMGSMSKQA VRSAKNAAKG YSSTQVLVRN ATSNDNHQVS KDSLIELAEK SYDSADFFEI MDMLDKRLND KGKYWRHIAK ALTVIDYLIR FGSENCVLWC RENLYIIKTL KEFRHEDDEG IDQGQIVRVK AKELTALLSD DERLNEERNM NIKGRNRKGR RR |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | 20 mM Tris, 250 mM NaCl and 1 mM DTT |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 72.6 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X