+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11414 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of right-handed protein cage consisting of 24 eleven-membered ring proteins held together by DTME cross linkers | |||||||||
 マップデータ マップデータ | ChiralB of the DTME cross-linked TRAP cage composed with 24 TRAP rings interconected with 240 DTME molecules | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) | |||||||||
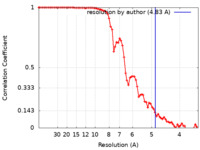
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.83 Å | |||||||||
 データ登録者 データ登録者 | Biela AP / Maskell D | |||||||||
| 資金援助 |  ポーランド, 1件 ポーランド, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Chemically induced protein cage assembly with programmable opening and cargo release. 著者: Izabela Stupka / Yusuke Azuma / Artur P Biela / Motonori Imamura / Simon Scheuring / Elżbieta Pyza / Olga Woźnicka / Daniel P Maskell / Jonathan G Heddle /    要旨: Engineered protein cages are promising tools that can be customized for applications in medicine and nanotechnology. A major challenge is developing a straightforward strategy for endowing cages with ...Engineered protein cages are promising tools that can be customized for applications in medicine and nanotechnology. A major challenge is developing a straightforward strategy for endowing cages with bespoke, inducible disassembly. Such cages would allow release of encapsulated cargoes at desired timing and location. Here, we achieve such programmable disassembly using protein cages, in which the subunits are held together by different molecular cross-linkers. This modular system enables cage disassembly to be controlled in a condition-dependent manner. Structural details of the resulting cages were determined using cryo–electron microscopy, which allowed observation of bridging cross-linkers at intended positions. Triggered disassembly was demonstrated by high-speed atomic force microscopy and subsequent cargo release using an encapsulated Förster resonance energy transfer pair whose signal depends on the quaternary structure of the cage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11414.map.gz emd_11414.map.gz | 38 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11414-v30.xml emd-11414-v30.xml emd-11414.xml emd-11414.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11414_fsc.xml emd_11414_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11414.png emd_11414.png | 100.7 KB | ||
| マスクデータ |  emd_11414_msk_1.map emd_11414_msk_1.map | 40.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_11414_half_map_1.map.gz emd_11414_half_map_1.map.gz emd_11414_half_map_2.map.gz emd_11414_half_map_2.map.gz | 37.2 MB 37.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11414 http://ftp.pdbj.org/pub/emdb/structures/EMD-11414 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11414 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11414 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11414_validation.pdf.gz emd_11414_validation.pdf.gz | 516.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11414_full_validation.pdf.gz emd_11414_full_validation.pdf.gz | 516.2 KB | 表示 | |
| XML形式データ |  emd_11414_validation.xml.gz emd_11414_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_11414_validation.cif.gz emd_11414_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11414 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11414 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11414 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11414 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11414.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11414.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ChiralB of the DTME cross-linked TRAP cage composed with 24 TRAP rings interconected with 240 DTME molecules | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.72336 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
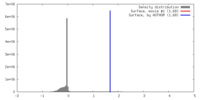
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11414_msk_1.map emd_11414_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_11414_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_11414_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : chemically induced artificial protein cage
| 全体 | 名称: chemically induced artificial protein cage |
|---|---|
| 要素 |
|
-超分子 #1: chemically induced artificial protein cage
| 超分子 | 名称: chemically induced artificial protein cage / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: DTME cross linked protein cage |
|---|---|
| 由来(天然) | 生物種:   Geobacillus stearothermophilus (バクテリア) Geobacillus stearothermophilus (バクテリア) |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 2.2 MDa |
-分子 #1: Transcription attenuation protein MtrB
| 分子 | 名称: Transcription attenuation protein MtrB / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 配列 | 文字列: MYTNSDFVVI KALEDGVNVI GLTRGADTRF HHSECLDKGE VLIAQFTEHT SAIKVRGKAY IQTSHGVIES EGKK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8.9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 4942 / 平均露光時間: 1.0 sec. / 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)