+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10836 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rix1-Rea1 pre-60S particle - Rix1-subcomplex, body 3 (rigid body refinement) | |||||||||
 マップデータ マップデータ | Rix1-subcomplex of the Rix1-Rea1 pre-60S particle | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pre-60S / Biogenesis / LSU / Large subunit / ribosome assembly / RIBOSOME | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / rixosome complex / pre-replicative complex assembly involved in nuclear cell cycle DNA replication / nuclear pre-replicative complex / regulation of DNA-templated DNA replication initiation / Major pathway of rRNA processing in the nucleolus and cytosol / MLL1 complex / maturation of LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / ribosomal large subunit assembly / DNA-templated DNA replication ...: / rixosome complex / pre-replicative complex assembly involved in nuclear cell cycle DNA replication / nuclear pre-replicative complex / regulation of DNA-templated DNA replication initiation / Major pathway of rRNA processing in the nucleolus and cytosol / MLL1 complex / maturation of LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / ribosomal large subunit assembly / DNA-templated DNA replication / rRNA processing / chromatin binding / nucleoplasm / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
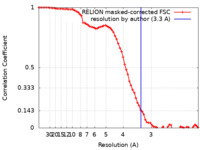
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Kater L / Beckmann R | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2020 ジャーナル: Mol Cell / 年: 2020タイトル: Construction of the Central Protuberance and L1 Stalk during 60S Subunit Biogenesis. 著者: Lukas Kater / Valentin Mitterer / Matthias Thoms / Jingdong Cheng / Otto Berninghausen / Roland Beckmann / Ed Hurt /  要旨: Ribosome assembly is driven by numerous assembly factors, including the Rix1 complex and the AAA ATPase Rea1. These two assembly factors catalyze 60S maturation at two distinct states, triggering ...Ribosome assembly is driven by numerous assembly factors, including the Rix1 complex and the AAA ATPase Rea1. These two assembly factors catalyze 60S maturation at two distinct states, triggering poorly understood large-scale structural transitions that we analyzed by cryo-electron microscopy. Two nuclear pre-60S intermediates were discovered that represent previously unknown states after Rea1-mediated removal of the Ytm1-Erb1 complex and reveal how the L1 stalk develops from a pre-mature nucleolar to a mature-like nucleoplasmic state. A later pre-60S intermediate shows how the central protuberance arises, assisted by the nearby Rix1-Rea1 machinery, which was solved in its pre-ribosomal context to molecular resolution. This revealed a Rix1-Ipi3 tetramer anchored to the pre-60S via Ipi1, strategically positioned to monitor this decisive remodeling. These results are consistent with a general underlying principle that temporarily stabilized immature RNA domains are successively remodeled by assembly factors, thereby ensuring failsafe assembly progression. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10836.map.gz emd_10836.map.gz | 4.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10836-v30.xml emd-10836-v30.xml emd-10836.xml emd-10836.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10836_fsc.xml emd_10836_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10836.png emd_10836.png | 59.8 KB | ||
| Filedesc metadata |  emd-10836.cif.gz emd-10836.cif.gz | 6.4 KB | ||
| その他 |  emd_10836_half_map_1.map.gz emd_10836_half_map_1.map.gz emd_10836_half_map_2.map.gz emd_10836_half_map_2.map.gz | 28.2 MB 28.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10836 http://ftp.pdbj.org/pub/emdb/structures/EMD-10836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10836 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10836_validation.pdf.gz emd_10836_validation.pdf.gz | 437.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10836_full_validation.pdf.gz emd_10836_full_validation.pdf.gz | 437 KB | 表示 | |
| XML形式データ |  emd_10836_validation.xml.gz emd_10836_validation.xml.gz | 13.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10836 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10836 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10836 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10836 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10836.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10836.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Rix1-subcomplex of the Rix1-Rea1 pre-60S particle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Relion half map (unfiltered, unmasked)
| ファイル | emd_10836_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion half map (unfiltered, unmasked) | ||||||||||||
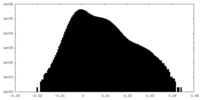
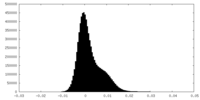
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Relion half map (unfiltered, unmasked)
| ファイル | emd_10836_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion half map (unfiltered, unmasked) | ||||||||||||
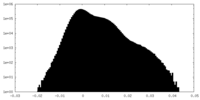
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rix1-Rea1 pre-60S assembly particle - Rix1-subcomplex
| 全体 | 名称: Rix1-Rea1 pre-60S assembly particle - Rix1-subcomplex |
|---|---|
| 要素 |
|
-超分子 #1: Rix1-Rea1 pre-60S assembly particle - Rix1-subcomplex
| 超分子 | 名称: Rix1-Rea1 pre-60S assembly particle - Rix1-subcomplex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Rix1-TAP Flag-Rea1 derived pre-60S assembly complex |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Pre-rRNA-processing protein IPI3
| 分子 | 名称: Pre-rRNA-processing protein IPI3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 61.836695 KDa |
| 配列 | 文字列: MDEQVIFTTN TSGTIASVHS FEQINLRQCS TQSRNSCVQV GNKYLFIAQA QKALINVYNL SGSFKRESVE QRLPLPEILK CLEVVENDG VQYDRIQGVN HNLPDFNLPY LLLGSTESGK LYIWELNSGI LLNVKPMAHY QSITKIKSIL NGKYIITSGN D SRVIIWQT ...文字列: MDEQVIFTTN TSGTIASVHS FEQINLRQCS TQSRNSCVQV GNKYLFIAQA QKALINVYNL SGSFKRESVE QRLPLPEILK CLEVVENDG VQYDRIQGVN HNLPDFNLPY LLLGSTESGK LYIWELNSGI LLNVKPMAHY QSITKIKSIL NGKYIITSGN D SRVIIWQT VDLVSASNDD PKPLCILHDH TLPVTDFQVS SSQGKFLSCT DTKLFTVSQD ATIRCYDLSL IGSKKKQKAN EN DVSIGKT PVLLATFTTP YSIKSIVLDP ADRACYIGTA EGCFSLNLFY KLKGNAIVNL LQSAGVNTVQ KGRVFSLVQR NSL TGGENE DLDALYAMGQ LVCENVLNSN VSCLEISMDG TLLLIGDTEG KVSIAEIYSK QIIRTIQTLT TSQDSVGEVT NLLT NPYRL ERGNLLFEGE SKGKQPSNNN GHNFMKIPNL QRVIFDGKNK GHLHDIWYQI GEPEAETDPN LALPLNDFNA YLEQV KTQE SIFSHIGKVS SNVKVIDNKI DATSSLDSNA AKDEEITELK TNIEALTHAY KELRDMHEKL YEEHQQMLDK Q UniProtKB: Pre-rRNA-processing protein IPI3 |
-分子 #2: Pre-rRNA-processing protein RIX1
| 分子 | 名称: Pre-rRNA-processing protein RIX1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 86.839844 KDa |
| 配列 | 文字列: MSEEFIAVST LARNLEIAKG NEFHTILATL RSPVYINEQL LKSELSFLVT KILKLIRSGN DFDLWKGCHT SVVTCAYNPL VLSTHGGQL LAAIYSRLEQ KTGFYSSVIS SSHGKQLFNT LISSVAIIID LMKNKPTLSR EALVPKLKAI IPTLITLSQY E PELVLPVL ...文字列: MSEEFIAVST LARNLEIAKG NEFHTILATL RSPVYINEQL LKSELSFLVT KILKLIRSGN DFDLWKGCHT SVVTCAYNPL VLSTHGGQL LAAIYSRLEQ KTGFYSSVIS SSHGKQLFNT LISSVAIIID LMKNKPTLSR EALVPKLKAI IPTLITLSQY E PELVLPVL QRILKRNTTT FKPFTNKFRT VLINLIISDY ASLGTKTQRL VCENFAYLHL LKIQVSDTSD DETQAHHKIY AD SNWRTGL MSILSQFKPI IQLCGEILDF EQDNELYKLI KSLPVIDESN NKEEFLPSLK LDFNAPLTLW EIPQRLSLLA DML VAFISL PTPFPIRVPL GGINSLCEVL LGVSNKYLPL KKELRHDNEL NGVINTILPQ IQFQGIRLWE IMVSKYGKCG LSFF EGILS SIELFIPLKK KSNNEIDFNV VGSLKFEFAT VFRLVNMILS HLGHQLNIIS VISQLIEVAL FLSHDKTLID SLFKN RKSI MKQQTKTKQS KRSKSAEGAF SDIYTHPELF VCKNSMNWFN EINDFFITAL NNWILPSTPH IQILKYSITQ SLRLKE RFG YIPESFVNLL RCEVLHPGSE RVSILPIAIS LLKNINDDMF ELLCHPKVPV GMVYQLHKPL DLGEDGEVRD DINKKEV ET NESSSNANTG LETLKALENL ENVTIPEPKH EVPKVVDDTA IFKKRSVEEV IERESTSSHK KVKFVEETTV DNGEELIV K KAVSQTKEEE KPMEDSEDEE QEEFEIPAIE LSDDEEEEEE EE UniProtKB: Pre-rRNA-processing protein RIX1 |
-分子 #3: Pre-rRNA-processing protein IPI1
| 分子 | 名称: Pre-rRNA-processing protein IPI1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 37.928895 KDa |
| 配列 | 文字列: MTKSRKQKQK KQDFLRKKLK VGKPKEKARN ATDTSFVSKT ISIRNQHLDQ NPHDLTKRLT LLKHHNINVR KETLTTFQKS IPSIIKSRL MTPLLTQSIP LICDESQQVR QGLIDLVDEI GSHDAEILKL HCNIFVLYIN MAMTHIVTQI QADSTKFLSH L LKYCGDEV ...文字列: MTKSRKQKQK KQDFLRKKLK VGKPKEKARN ATDTSFVSKT ISIRNQHLDQ NPHDLTKRLT LLKHHNINVR KETLTTFQKS IPSIIKSRL MTPLLTQSIP LICDESQQVR QGLIDLVDEI GSHDAEILKL HCNIFVLYIN MAMTHIVTQI QADSTKFLSH L LKYCGDEV VRKSWVKLLN GVFGVLGWGQ VGKNDSASIV QTKKRNAKYV TIHLNALYTL VEYGCQDERA RSDGDTAETT ED SGTLRNP YLIPDYPQPF EHLKLFTREL KVQDATSSGV NATLLSLATQ DIDTRKAVFI EQFLPIVRKK IEVIIKEGGE CGK SANKLK TLLAKIFD UniProtKB: Pre-rRNA-processing protein IPI1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R3/3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-48 / 撮影したグリッド数: 1 / 平均露光時間: 10.0 sec. / 平均電子線量: 75.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6yle: |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X