+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0814 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
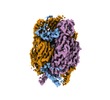
| タイトル | African swine fever virus major capsid protein p72 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | African Swine Fever virus capsomer / Trisymmetron / NCLDV / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Major capsid protein, C-terminal / Major capsid protein, C-terminal domain superfamily / Large eukaryotic DNA virus major capsid protein / Group II dsDNA virus coat/capsid protein / viral capsid / structural molecule activity / B646L 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   African swine fever virus (アフリカ豚コレラウイルス) African swine fever virus (アフリカ豚コレラウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Wang N / Rao Z | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2019 ジャーナル: Science / 年: 2019タイトル: Architecture of African swine fever virus and implications for viral assembly. 著者: Nan Wang / Dongming Zhao / Jialing Wang / Yangling Zhang / Ming Wang / Yan Gao / Fang Li / Jingfei Wang / Zhigao Bu / Zihe Rao / Xiangxi Wang /  要旨: African swine fever virus (ASFV) is a giant and complex DNA virus that causes a highly contagious and often lethal swine disease for which no vaccine is available. Using an optimized image ...African swine fever virus (ASFV) is a giant and complex DNA virus that causes a highly contagious and often lethal swine disease for which no vaccine is available. Using an optimized image reconstruction strategy, we solved the ASFV capsid structure up to 4.1 angstroms, which is built from 17,280 proteins, including one major (p72) and four minor (M1249L, p17, p49, and H240R) capsid proteins organized into pentasymmetrons and trisymmetrons. The atomic structure of the p72 protein informs putative conformational epitopes, distinguishing ASFV from other nucleocytoplasmic large DNA viruses. The minor capsid proteins form a complicated network below the outer capsid shell, stabilizing the capsid by holding adjacent capsomers together. Acting as core organizers, 100-nanometer-long M1249L proteins run along each edge of the trisymmetrons that bridge two neighboring pentasymmetrons and form extensive intermolecular networks with other capsid proteins, driving the formation of the capsid framework. These structural details unveil the basis of capsid stability and assembly, opening up new avenues for African swine fever vaccine development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0814.map.gz emd_0814.map.gz | 2.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0814-v30.xml emd-0814-v30.xml emd-0814.xml emd-0814.xml | 11.5 KB 11.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0814.png emd_0814.png | 102.7 KB | ||
| Filedesc metadata |  emd-0814.cif.gz emd-0814.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0814 http://ftp.pdbj.org/pub/emdb/structures/EMD-0814 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0814 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0814 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0814_validation.pdf.gz emd_0814_validation.pdf.gz | 409.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0814_full_validation.pdf.gz emd_0814_full_validation.pdf.gz | 408.8 KB | 表示 | |
| XML形式データ |  emd_0814_validation.xml.gz emd_0814_validation.xml.gz | 5.8 KB | 表示 | |
| CIF形式データ |  emd_0814_validation.cif.gz emd_0814_validation.cif.gz | 6.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0814 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0814 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0814 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0814 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0814.map.gz / 形式: CCP4 / 大きさ: 18.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0814.map.gz / 形式: CCP4 / 大きさ: 18.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : African swine fever virus
| 全体 | 名称:   African swine fever virus (アフリカ豚コレラウイルス) African swine fever virus (アフリカ豚コレラウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: African swine fever virus
| 超分子 | 名称: African swine fever virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10497 / 生物種: African swine fever virus / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|
-分子 #1: B646L,Major capsid protein
| 分子 | 名称: B646L,Major capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   African swine fever virus (アフリカ豚コレラウイルス) African swine fever virus (アフリカ豚コレラウイルス) |
| 分子量 | 理論値: 73.219562 KDa |
| 配列 | 文字列: MASGGAFCLI ANDGKADKII LAQDLLNSRI SNIKNVNKSY GKPDPEPTLS QIEETHLVHF NAHFKPYVPV GFEYNKVRPH TGTPTLGNK LTFGIPQYGD FFHDMVGHHI LGACHSSWQD APIQGTSQMG AHGQLQTFPR NGYDWDNQTP LEGAVYTLVD P FGRPIVPG ...文字列: MASGGAFCLI ANDGKADKII LAQDLLNSRI SNIKNVNKSY GKPDPEPTLS QIEETHLVHF NAHFKPYVPV GFEYNKVRPH TGTPTLGNK LTFGIPQYGD FFHDMVGHHI LGACHSSWQD APIQGTSQMG AHGQLQTFPR NGYDWDNQTP LEGAVYTLVD P FGRPIVPG TKNAYRNLVY YCEYPGLRLY ENVRFEVNGN SLDEYSSDVT TLVRKFCIPG DKMTGYKHLV GQEVSVEGTS GP LLCNIHD LHKPHQSKPI LTDENDTQRT CSHTNPKFLS QHFPHFPENI QTAGKQDITP ITDATYLDIR RNVHYSCNGP QTP KYYQPP LALWIKLRFW FNDNVNLAIP SVSIPFGERF ITIKLASQKD LVNEFPGLFV RQSTVIAGRP SRRNIRFKPW FIPG VINEI SLTNNELYIN NLFVTPEIHN LYVKRVRFSL IRVHKTQVTH TNNNHHDEKL MSALKWPIEY MFIGLKPTWN ISDQN PHQH RDWHKFGHVV NAIMQPTHHA EISFQDRDTA LPDACSSISD ISPVTYPITL PIIKNISVTA HGINLIDKFP SQFCSS YIP FHYGGNAIKT PDDPGAMMIT FALYPREEYQ PSGHINVSRA REFYISWDSD YVGSITTADL VVSSSAINFL LLQNGSA VL RYST |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















