+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0568 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM 3D map of human ATP-citrate lyase N-terminal segment in complex with inhibitor NDI-091143 | |||||||||
 マップデータ マップデータ | em-volume_P1 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.11 Å | |||||||||
 データ登録者 データ登録者 | Wei J / Tong L | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: An allosteric mechanism for potent inhibition of human ATP-citrate lyase. 著者: Jia Wei / Silvana Leit / Jun Kuai / Eric Therrien / Salma Rafi / H James Harwood / Byron DeLaBarre / Liang Tong /  要旨: ATP-citrate lyase (ACLY) is a central metabolic enzyme and catalyses the ATP-dependent conversion of citrate and coenzyme A (CoA) to oxaloacetate and acetyl-CoA. The acetyl-CoA product is crucial for ...ATP-citrate lyase (ACLY) is a central metabolic enzyme and catalyses the ATP-dependent conversion of citrate and coenzyme A (CoA) to oxaloacetate and acetyl-CoA. The acetyl-CoA product is crucial for the metabolism of fatty acids, the biosynthesis of cholesterol, and the acetylation and prenylation of proteins. There has been considerable interest in ACLY as a target for anti-cancer drugs, because many cancer cells depend on its activity for proliferation. ACLY is also a target against dyslipidaemia and hepatic steatosis, with a compound currently in phase 3 clinical trials. Many inhibitors of ACLY have been reported, but most of them have weak activity. Here we report the development of a series of low nanomolar, small-molecule inhibitors of human ACLY. We have also determined the structure of the full-length human ACLY homo-tetramer in complex with one of these inhibitors (NDI-091143) by cryo-electron microscopy, which reveals an unexpected mechanism of inhibition. The compound is located in an allosteric, mostly hydrophobic cavity next to the citrate-binding site, and requires extensive conformational changes in the enzyme that indirectly disrupt citrate binding. The observed binding mode is supported by and explains the structure-activity relationships of these compounds. This allosteric site greatly enhances the 'druggability' of ACLY and represents an attractive target for the development of new ACLY inhibitors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0568.map.gz emd_0568.map.gz | 4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0568-v30.xml emd-0568-v30.xml emd-0568.xml emd-0568.xml | 15.8 KB 15.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0568_fsc.xml emd_0568_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0568.png emd_0568.png | 35.9 KB | ||
| マスクデータ |  emd_0568_msk_1.map emd_0568_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0568_half_map_1.map.gz emd_0568_half_map_1.map.gz emd_0568_half_map_2.map.gz emd_0568_half_map_2.map.gz | 48.4 MB 48.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0568 http://ftp.pdbj.org/pub/emdb/structures/EMD-0568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0568 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0568 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_0568_validation.pdf.gz emd_0568_validation.pdf.gz | 78.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_0568_full_validation.pdf.gz emd_0568_full_validation.pdf.gz | 77.4 KB | 表示 | |
| XML形式データ |  emd_0568_validation.xml.gz emd_0568_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0568 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0568 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-0568 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0568.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0568.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-volume_P1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0605 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
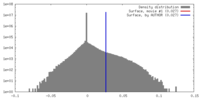
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0568_msk_1.map emd_0568_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
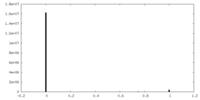
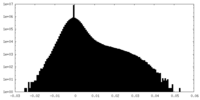
| 密度ヒストグラム |
-ハーフマップ: em-half-volume P1
| ファイル | emd_0568_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-half-volume_P1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
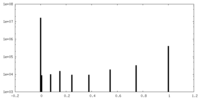
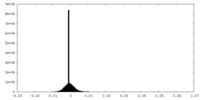
| 密度ヒストグラム |
-ハーフマップ: em-half-volume P2
| ファイル | emd_0568_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-half-volume_P2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
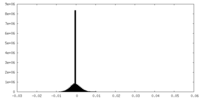
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : protein-inhibitor complex
| 全体 | 名称: protein-inhibitor complex |
|---|---|
| 要素 |
|
-超分子 #1: protein-inhibitor complex
| 超分子 | 名称: protein-inhibitor complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.7 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 2803 / 平均露光時間: 10.0 sec. / 平均電子線量: 71.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -0.002 µm / 最小 デフォーカス(公称値): -0.0008 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)