+検索条件
-Structure paper
| タイトル | Discovery and Clinical Proof-of-Concept of RLY-2608, a First-in-Class Mutant-Selective Allosteric PI3Kα Inhibitor That Decouples Antitumor Activity from Hyperinsulinemia. |
|---|---|
| ジャーナル・号・ページ | Cancer Discov, Vol. 14, Issue 2, Page 240-257, Year 2024 |
| 掲載日 | 2024年2月8日 |
 著者 著者 | Andreas Varkaris / Ermira Pazolli / Hakan Gunaydin / Qi Wang / Levi Pierce / Alessandro A Boezio / Artemisa Bulku / Lucian DiPietro / Cary Fridrich / Adam Frost / Fabrizio Giordanetto / Erika P Hamilton / Katherine Harris / Michael Holliday / Tamieka L Hunter / Amanda Iskandar / Yongli Ji / Alexandre Larivée / Jonathan R LaRochelle / André Lescarbeau / Fabien Llambi / Brenda Lormil / Mary M Mader / Brenton G Mar / Iain Martin / Thomas H McLean / Klaus Michelsen / Yakov Pechersky / Erika Puente-Poushnejad / Kevin Raynor / Dipali Rogala / Ramin Samadani / Alison M Schram / Kelley Shortsleeves / Sweta Swaminathan / Shahein Tajmir / Gege Tan / Yong Tang / Roberto Valverde / Bryan Wehrenberg / Jeremy Wilbur / Bret R Williams / Hongtao Zeng / Hanmo Zhang / W Patrick Walters / Beni B Wolf / David E Shaw / Donald A Bergstrom / James Watters / James S Fraser / Pascal D Fortin / D Randal Kipp /   |
| PubMed 要旨 | PIK3CA (PI3Kα) is a lipid kinase commonly mutated in cancer, including ∼40% of hormone receptor-positive breast cancer. The most frequently observed mutants occur in the kinase and helical domains. ...PIK3CA (PI3Kα) is a lipid kinase commonly mutated in cancer, including ∼40% of hormone receptor-positive breast cancer. The most frequently observed mutants occur in the kinase and helical domains. Orthosteric PI3Kα inhibitors suffer from poor selectivity leading to undesirable side effects, most prominently hyperglycemia due to inhibition of wild-type (WT) PI3Kα. Here, we used molecular dynamics simulations and cryo-electron microscopy to identify an allosteric network that provides an explanation for how mutations favor PI3Kα activation. A DNA-encoded library screen leveraging electron microscopy-optimized constructs, differential enrichment, and an orthosteric-blocking compound led to the identification of RLY-2608, a first-in-class allosteric mutant-selective inhibitor of PI3Kα. RLY-2608 inhibited tumor growth in PIK3CA-mutant xenograft models with minimal impact on insulin, a marker of dysregulated glucose homeostasis. RLY-2608 elicited objective tumor responses in two patients diagnosed with advanced hormone receptor-positive breast cancer with kinase or helical domain PIK3CA mutations, with no observed WT PI3Kα-related toxicities. SIGNIFICANCE: Treatments for PIK3CA-mutant cancers are limited by toxicities associated with the inhibition of WT PI3Kα. Molecular dynamics, cryo-electron microscopy, and DNA-encoded libraries were used to develop RLY-2608, a first-in-class inhibitor that demonstrates mutant selectivity in patients. This marks the advance of clinical mutant-selective inhibition that overcomes limitations of orthosteric PI3Kα inhibitors. See related commentary by Gong and Vanhaesebroeck, p. 204 . See related article by Varkaris et al., p. 227 . This article is featured in Selected Articles from This Issue, p. 201. |
 リンク リンク |  Cancer Discov / Cancer Discov /  PubMed:37916956 / PubMed:37916956 /  PubMed Central PubMed Central |
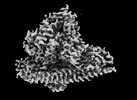
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.51 - 3.62 Å |
| 構造データ | EMDB-41617, PDB-8tu6:  PDB-8ts7:  PDB-8ts8:  PDB-8ts9:  PDB-8tsa:  PDB-8tsb:  PDB-8tsc:  PDB-8tsd: |
| 化合物 |  ChemComp-UE9:  ChemComp-UIW: 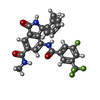 ChemComp-UJC: 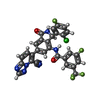 ChemComp-XUZ: |
| 由来 |
|
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  Lipid kinase (キナーゼ) / Lipid kinase (キナーゼ) /  SIGNALING PROTEIN/INHIBITOR / SIGNALING PROTEIN/INHIBITOR /  SIGNALING PROTEIN-INHIBITOR complex / TRANSFERASE/INHIBITOR / TRANSFERASE-INHIBITOR complex / SIGNALING PROTEIN-INHIBITOR complex / TRANSFERASE/INHIBITOR / TRANSFERASE-INHIBITOR complex /  CYTOSOLIC PROTEIN (細胞質基質) / PI3Kalapha / isoform selective / CYTOSOLIC PROTEIN (細胞質基質) / PI3Kalapha / isoform selective /  structure-based drug design. (医薬品設計) structure-based drug design. (医薬品設計) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について