+検索条件
-Structure paper
| タイトル | Analysis of the conformational heterogeneity of the Rieske iron-sulfur protein in complex III by cryo-EM. |
|---|---|
| ジャーナル・号・ページ | IUCrJ, Vol. 10, Issue Pt 1, Page 27-37, Year 2023 |
| 掲載日 | 2023年1月1日 |
 著者 著者 | Jan Philip Wieferig / Werner Kühlbrandt /  |
| PubMed 要旨 | Movement of the Rieske domain of the iron-sulfur protein is essential for intramolecular electron transfer within complex III (CIII) of the respiratory chain as it bridges a gap in the cofactor chain ...Movement of the Rieske domain of the iron-sulfur protein is essential for intramolecular electron transfer within complex III (CIII) of the respiratory chain as it bridges a gap in the cofactor chain towards the electron acceptor cytochrome c. We present cryo-EM structures of CIII from Yarrowia lipolytica at resolutions up to 2.0 Å under different conditions, with different redox states of the cofactors of the high-potential chain. All possible permutations of three primary positions were observed, indicating that the two halves of the dimeric complex act independently. Addition of the substrate analogue decylubiquinone to CIII with a reduced high-potential chain increased the occupancy of the Q site. The extent of Rieske domain interactions through hydrogen bonds to the cytochrome b and cytochrome c subunits varied depending on the redox state and substrate. In the absence of quinols, the reduced Rieske domain interacted more closely with cytochrome b and cytochrome c than in the oxidized state. Upon addition of the inhibitor antimycin A, the heterogeneity of the cd-helix and ef-loop increased, which may be indicative of a long-range effect on the Rieske domain. |
 リンク リンク |  IUCrJ / IUCrJ /  PubMed:36598500 / PubMed:36598500 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.0 - 3.7 Å |
| 構造データ | EMDB-15312, PDB-8ab6: EMDB-15313, PDB-8ab7: EMDB-15314, PDB-8ab8: EMDB-15315, PDB-8ab9: EMDB-15316, PDB-8aba: EMDB-15317, PDB-8abb: EMDB-15318, PDB-8abe: EMDB-15319, PDB-8abf: EMDB-15320, PDB-8abg: EMDB-15321, PDB-8abh: EMDB-15322, PDB-8abi: EMDB-15323, PDB-8abj: EMDB-15324, PDB-8abk: EMDB-15325, PDB-8abl: EMDB-15326, PDB-8abm: EMDB-15332, PDB-8ac3: EMDB-15333, PDB-8ac4: EMDB-15334, PDB-8ac5: |
| 化合物 |  ChemComp-HEM:  ChemComp-PC1:  ChemComp-PTY:  ChemComp-CDL:  ChemComp-LMT:  ChemComp-XP4:  ChemComp-HEC:  ChemComp-HOH: 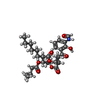 ChemComp-AWB: 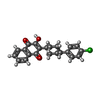 ChemComp-AOQ:  ChemComp-FES: 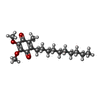 ChemComp-DCQ: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / oxidoreductase / electron transport chain / inhibitor |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について







































 yarrowia lipolytica (酵母)
yarrowia lipolytica (酵母)