+検索条件
-Structure paper
| タイトル | Structures of the AMPA receptor in complex with its auxiliary subunit cornichon. |
|---|---|
| ジャーナル・号・ページ | Science, Vol. 366, Issue 6470, Page 1259-1263, Year 2019 |
| 掲載日 | 2019年12月6日 |
 著者 著者 | Terunaga Nakagawa /  |
| PubMed 要旨 | In the brain, AMPA-type glutamate receptors (AMPARs) form complexes with their auxiliary subunits and mediate the majority of fast excitatory neurotransmission. Signals transduced by these complexes ...In the brain, AMPA-type glutamate receptors (AMPARs) form complexes with their auxiliary subunits and mediate the majority of fast excitatory neurotransmission. Signals transduced by these complexes are critical for synaptic plasticity, learning, and memory. The two major categories of AMPAR auxiliary subunits are transmembrane AMPAR regulatory proteins (TARPs) and cornichon homologs (CNIHs); these subunits share little homology and play distinct roles in controlling ion channel gating and trafficking of AMPAR. Here, I report high-resolution cryo-electron microscopy structures of AMPAR in complex with CNIH3. Contrary to its predicted membrane topology, CNIH3 lacks an extracellular domain and instead contains four membrane-spanning helices. The protein-protein interaction interface that dictates channel modulation and the lipids surrounding the complex are revealed. These structures provide insights into the molecular mechanism for ion channel modulation and assembly of AMPAR/CNIH3 complexes. |
 リンク リンク |  Science / Science /  PubMed:31806817 PubMed:31806817 |
| 手法 | EM (単粒子) |
| 解像度 | 2.97 - 4.4 Å |
| 構造データ | EMDB-20330, PDB-6peq:  EMDB-20332: EMDB-20654, PDB-6u5s: EMDB-20666, PDB-6u6i:  EMDB-20717: EMDB-20727, PDB-6ucb:  EMDB-20732: EMDB-20733, PDB-6ud4: EMDB-20734, PDB-6ud8: |
| 化合物 | 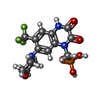 ChemComp-ZK1:  ChemComp-OLC: 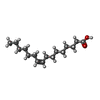 ChemComp-PAM:  ChemComp-CLR:  ChemComp-NAG: |
| 由来 |
|
 キーワード キーワード |  TRANSPORT PROTEIN (運搬体タンパク質) / TRANSPORT PROTEIN (運搬体タンパク質) /  ionotropic glutamate receptor / ionotropic glutamate receptor /  AMPA receptor (AMPA型グルタミン酸受容体) / AMPA receptor (AMPA型グルタミン酸受容体) /  cornichon (きゅうりピクルス) / auxiliary subunit / cornichon (きゅうりピクルス) / auxiliary subunit /  ion channel (イオンチャネル) / ion channel (イオンチャネル) /  ligand gated ion channel (リガンド依存性イオンチャネル) / ligand gated ion channel (リガンド依存性イオンチャネル) /  synaptic transmission / excitatory synaptic transmission / synaptic transmission / excitatory synaptic transmission /  neurotransmitter receptor (神経伝達物質受容体) / neurotransmitter receptor (神経伝達物質受容体) /  stargazin / TARP / ZK200775 / stargazin / TARP / ZK200775 /  lipid (脂質) / lipid (脂質) /  MPQX MPQX |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について
















