+検索条件
-Structure paper
| タイトル | Structural Basis for High Specificity of Amadori Compound and Mannopine Opine Binding in Bacterial Pathogens. |
|---|---|
| ジャーナル・号・ページ | J. Biol. Chem., Vol. 291, Page 22638-22649, Year 2016 |
| 掲載日 | 2016年6月10日 (構造データの登録日) |
 著者 著者 | Marty, L. / Vigouroux, A. / Aumont-Nicaise, M. / Dessaux, Y. / Faure, D. / Morera, S. |
 リンク リンク |  J. Biol. Chem. / J. Biol. Chem. /  PubMed:27609514 PubMed:27609514 |
| 手法 | X線回折 |
| 解像度 | 1.5 - 2.54 Å |
| 構造データ |  PDB-5l9g:  PDB-5l9i:  PDB-5l9l:  PDB-5l9o:  PDB-5l9p:  PDB-5l9s:  PDB-5lom:  PDB-6hqh: |
| 化合物 | 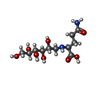 ChemComp-MO0:  ChemComp-EDO:  ChemComp-CA:  ChemComp-HOH: 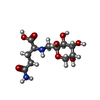 ChemComp-SNW:  ChemComp-PEG: 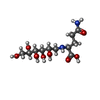 ChemComp-GOP:  ChemComp-SO4:  ChemComp-ACT: |
| 由来 |
|
 キーワード キーワード | TRANSPORT PROTEIN / periplasmic binding protein ABC transporter / Periplasmic binding protein / ABC transporter / synthetic opine / Amadori compound / opine / DFG / protein transport |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 agrobacterium tumefaciens str. b6 (植物への病原性)
agrobacterium tumefaciens str. b6 (植物への病原性)