+検索条件
-Structure paper
| タイトル | Structural Basis for Interactions Between Contactin Family Members and Protein-tyrosine Phosphatase Receptor Type G in Neural Tissues. |
|---|---|
| ジャーナル・号・ページ | J. Biol. Chem., Vol. 291, Page 21335-21349, Year 2016 |
| 掲載日 | 2015年10月6日 (構造データの登録日) |
 著者 著者 | Nikolaienko, R.M. / Hammel, M. / Dubreuil, V. / Zalmai, R. / Hall, D.R. / Mehzabeen, N. / Karuppan, S.J. / Harroch, S. / Stella, S.L. / Bouyain, S. |
 リンク リンク |  J. Biol. Chem. / J. Biol. Chem. /  PubMed:27539848 PubMed:27539848 |
| 手法 | X線回折 |
| 解像度 | 2 - 2.822 Å |
| 構造データ |  PDB-5e4i:  PDB-5e4q:  PDB-5e4s:  PDB-5e52:  PDB-5e53:  PDB-5e55:  PDB-5e5r:  PDB-5e5u:  PDB-5e7l:  PDB-5i99: |
| 化合物 |  ChemComp-NAG:  ChemComp-HOH:  ChemComp-PO4:  ChemComp-IMD:  ChemComp-MLI:  ChemComp-FMT: 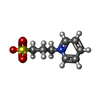 ChemComp-1PS:  ChemComp-ACY: 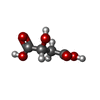 ChemComp-MLT:  ChemComp-GOL: |
| 由来 |
|
 キーワード キーワード | CELL ADHESION / Neural cell adhesion molecule / Immunoglobulin-like domains / Horseshoe-like conformation / Fibronectin type III domains / Hydrolase/Cell Adhesion / Receptor-type protein tyrosine phosphatase / Immunoglobulin domains / Carbonic anhydrase-like domain / Hydrolase-Cell Adhesion complex / Cell Adhesion/Hydrolase / Cell Adhesion-Hydrolase complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 homo sapiens (ヒト)
homo sapiens (ヒト)
