+検索条件
-Structure paper
| タイトル | Experimental and computational active site mapping as a starting point to fragment-based lead discovery. |
|---|---|
| ジャーナル・号・ページ | Chemmedchem, Vol. 7, Page 248-261, Year 2012 |
| 掲載日 | 2010年4月29日 (構造データの登録日) |
 著者 著者 | Behnen, J. / Koster, H. / Neudert, G. / Craan, T. / Heine, A. / Klebe, G. |
 リンク リンク |  Chemmedchem / Chemmedchem /  PubMed:22213702 PubMed:22213702 |
| 手法 | X線回折 |
| 解像度 | 1.22 - 2.09 Å |
| 構造データ |  PDB-3ms3:  PDB-3msa:  PDB-3msf:  PDB-3msn:  PDB-3n21:  PDB-3n4a:  PDB-3n9w:  PDB-3nn7:  PDB-3nx8:  PDB-3pcz:  PDB-3prs:  PDB-3pvk:  PDB-3pww: |
| 化合物 |  ChemComp-ZN:  ChemComp-CA:  ChemComp-ANL:  ChemComp-IPA:  ChemComp-GOL:  ChemComp-DMS:  ChemComp-HOH:  ChemComp-B3R:  ChemComp-URE:  ChemComp-NMU:  ChemComp-PGO:  ChemComp-MN:  ChemComp-CL:  ChemComp-PGR:  ChemComp-BXA:  ChemComp-IPH:  ChemComp-BEN:  ChemComp-RIT:  ChemComp-1PE:  ChemComp-PGE:  ChemComp-MPD:  ChemComp-IMD: 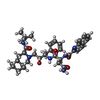 ChemComp-ROC: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / PROTEASE / Aniline / Fragement soaking / Fragment based lead discovery / METALLOPROTEASE / Metal-binding / Secreted / Zymogen / Fragment soaking / 3-Bromophenol / Urea / N-methylurea / S-1 / 2-Propandiol / ISOMERASE / INTRAMOLECULAR OXIDOREDUCTSE / TIM BARREL-BETA-ALPHA-BARRELS / TWO METAL BINDING ITES / Manganese / TRANSFERASE / YGBP / CYTIDYLYLTRANSFERASE / DEOXYXYLULOSE-5-PHOSPHATE PATHWAY (DXP) / ISOPRENOID BIOSYNTHESIS / 1 / 2-Propanediol / FBLD / 2-Bromoacetate / Fragment-based lead discovery / ATP Binding / Phosphorylation / HYDROLASE/HYDROLASE INHIBITOR / HYDROLASE-HYDROLASE INHIBITOR complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 homo sapiens (ヒト)
homo sapiens (ヒト) cryphonectria parasitica (菌類)
cryphonectria parasitica (菌類) candida albicans (酵母)
candida albicans (酵母)