+検索条件
-Structure paper
| タイトル | Oligomeric interactions maintain active-site structure in a noncooperative enzyme family. |
|---|---|
| ジャーナル・号・ページ | EMBO J, Vol. 41, Issue 17, Page e108368, Year 2022 |
| 掲載日 | 2022年9月1日 |
 著者 著者 | Yaohui Li / Rongzhen Zhang / Chi Wang / Farhad Forouhar / Oliver B Clarke / Sergey Vorobiev / Shikha Singh / Gaetano T Montelione / Thomas Szyperski / Yan Xu / John F Hunt /   |
| PubMed 要旨 | The evolutionary benefit accounting for widespread conservation of oligomeric structures in proteins lacking evidence of intersubunit cooperativity remains unclear. Here, crystal and cryo-EM ...The evolutionary benefit accounting for widespread conservation of oligomeric structures in proteins lacking evidence of intersubunit cooperativity remains unclear. Here, crystal and cryo-EM structures, and enzymological data, demonstrate that a conserved tetramer interface maintains the active-site structure in one such class of proteins, the short-chain dehydrogenase/reductase (SDR) superfamily. Phylogenetic comparisons support a significantly longer polypeptide being required to maintain an equivalent active-site structure in the context of a single subunit. Oligomerization therefore enhances evolutionary fitness by reducing the metabolic cost of enzyme biosynthesis. The large surface area of the structure-stabilizing oligomeric interface yields a synergistic gain in fitness by increasing tolerance to activity-enhancing yet destabilizing mutations. We demonstrate that two paralogous SDR superfamily enzymes with different specificities can form mixed heterotetramers that combine their individual enzymological properties. This suggests that oligomerization can also diversify the functions generated by a given metabolic investment, enhancing the fitness advantage provided by this architectural strategy. |
 リンク リンク |  EMBO J / EMBO J /  PubMed:35801308 / PubMed:35801308 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.59 - 4.28 Å |
| 構造データ | EMDB-32211, PDB-7vyq:  EMDB-32212: Oligomeric interactions maintain active-site structure in a non-cooperative enzyme family 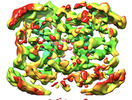 EMDB-32213: Oligomeric interactions maintain active-site structure in a non-cooperative enzyme family  PDB-7dld:  PDB-7dll:  PDB-7dlm:  PDB-7dmg:  PDB-7dn1: |
| 化合物 |  ChemComp-MG:  ChemComp-HOH:  ChemComp-NDP:  ChemComp-NAP: 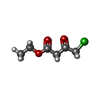 ChemComp-83I: |
| 由来 |
|
 キーワード キーワード | OXIDOREDUCTASE / Rossman fold / tetramer / tag-free / wild type / wild type with NADPH |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





 candida parapsilosis (酵母)
candida parapsilosis (酵母)