+検索条件
-Structure paper
| タイトル | Fusion protein strategies for cryo-EM study of G protein-coupled receptors. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 13, Issue 1, Page 4366, Year 2022 |
| 掲載日 | 2022年7月28日 |
 著者 著者 | Kaihua Zhang / Hao Wu / Nicholas Hoppe / Aashish Manglik / Yifan Cheng /  |
| PubMed 要旨 | Single particle cryogenic-electron microscopy (cryo-EM) is used extensively to determine structures of activated G protein-coupled receptors (GPCRs) in complex with G proteins or arrestins. However, ...Single particle cryogenic-electron microscopy (cryo-EM) is used extensively to determine structures of activated G protein-coupled receptors (GPCRs) in complex with G proteins or arrestins. However, applying it to GPCRs without signaling proteins remains challenging because most receptors lack structural features in their soluble domains to facilitate image alignment. In GPCR crystallography, inserting a fusion protein between transmembrane helices 5 and 6 is a highly successful strategy for crystallization. Although a similar strategy has the potential to broadly facilitate cryo-EM structure determination of GPCRs alone without signaling protein, the critical determinants that make this approach successful are not yet clear. Here, we address this shortcoming by exploring different fusion protein designs, which lead to structures of antagonist bound A adenosine receptor at 3.4 Å resolution and unliganded Smoothened at 3.7 Å resolution. The fusion strategies explored here are likely applicable to cryo-EM interrogation of other GPCRs and small integral membrane proteins. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:35902590 / PubMed:35902590 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.4 - 6.7 Å |
| 構造データ | EMDB-25648, PDB-7t32: EMDB-27062, PDB-8cxo:  EMDB-27063: Cryo-EM structure of the unliganded mSMO-PGS1 in a lipidic environment |
| 化合物 | 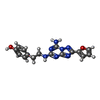 ChemComp-ZMA:  ChemComp-CLR: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / A2AAR / GPCR / ADENOSINE RECEPTOR / G-protein coupled receptor / Smoothened / unliganded state / lipid system |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について







 homo sapiens (ヒト)
homo sapiens (ヒト)

 pyrococcus abyssi (strain ge5 / orsay) (古細菌)
pyrococcus abyssi (strain ge5 / orsay) (古細菌)
