| タイトル | Snapshots of PLP-substrate and PLP-product external aldimines as intermediates in two types of cysteine desulfurase enzymes. |
|---|
| ジャーナル・号・ページ | Febs J., Year 2019 |
|---|
| 掲載日 | 2016年12月9日 (構造データの登録日) |
|---|
 著者 著者 | Nakamura, R. / Hikita, M. / Ogawa, S. / Takahashi, Y. / Fujishiro, T. |
|---|
 リンク リンク |  Febs J. / Febs J. /  PubMed:31587510 PubMed:31587510 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.96 - 3.24 Å |
|---|
| 構造データ | PDB-5wt2:
NifS from Helicobacter pylori
手法: X-RAY DIFFRACTION / 解像度: 2.301 Å PDB-5wt4:
L-Cysteine-PLP intermediate of NifS from Helicobacter pylori
手法: X-RAY DIFFRACTION / 解像度: 2.92 Å PDB-5zs9:
SufS from Bacillus subtilis in the resting state
手法: X-RAY DIFFRACTION / 解像度: 2.8 Å PDB-5zsk:
SufS from Bacillus subtilis soaked with L-cysteine for 63 sec
手法: X-RAY DIFFRACTION / 解像度: 3.24 Å PDB-5zso:
SufS from Bacillus subtilis, soaked with L-cysteine for 90 sec
手法: X-RAY DIFFRACTION / 解像度: 2.7 Å PDB-5zsp:
NifS from Hydrogenimonas thermophila
手法: X-RAY DIFFRACTION / 解像度: 2.57 Å PDB-5zst:
NifS from Hydrogenimonas thermopile in a persulfurated form
手法: X-RAY DIFFRACTION / 解像度: 3.1 Å PDB-6kfz:
SufS from Bacillus subtilis, soaked with L-cysteine for 90 sec at 1.96 angstrom resolution
手法: X-RAY DIFFRACTION / 解像度: 1.96 Å PDB-6kg0:
NifS from Helicobacter pylori, soaked with L-cysteine for 118 sec
手法: X-RAY DIFFRACTION / 解像度: 2.78 Å PDB-6kg1:
NifS from Helicobacter pylori, soaked with L-cysteine for 180 sec
手法: X-RAY DIFFRACTION / 解像度: 2.7 Å |
|---|
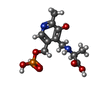
| 化合物 | ChemComp-C6P:
N-({3-HYDROXY-2-METHYL-5-[(PHOSPHONOOXY)METHYL]PYRIDIN-4-YL}METHYL)-L-CYSTEINE
|
|---|
| 由来 |   helicobacter pylori (strain atcc 700392 / 26695) (ピロリ菌) helicobacter pylori (strain atcc 700392 / 26695) (ピロリ菌)   bacillus subtilis (strain 168) (枯草菌) bacillus subtilis (strain 168) (枯草菌) hydrogenimonas thermophila (バクテリア) hydrogenimonas thermophila (バクテリア)
|
|---|
 キーワード キーワード |  TRANSFERASE (転移酵素) / Iron-sulfur cluster biogenesis / TRANSFERASE (転移酵素) / Iron-sulfur cluster biogenesis /  cysteine desulfurase / cysteine desulfurase /  BIOSYNTHETIC PROTEIN (生合成) / BIOSYNTHETIC PROTEIN (生合成) /  iron (鉄) / iron (鉄) /  sulfur (硫黄) / sulfur (硫黄) /  reaction intermediate (反応中間体) reaction intermediate (反応中間体) |
|---|
 著者
著者 リンク
リンク Febs J. /
Febs J. /  PubMed:31587510
PubMed:31587510















 キーワード
キーワード TRANSFERASE (転移酵素) / Iron-sulfur cluster biogenesis /
TRANSFERASE (転移酵素) / Iron-sulfur cluster biogenesis /  cysteine desulfurase /
cysteine desulfurase /  BIOSYNTHETIC PROTEIN (生合成) /
BIOSYNTHETIC PROTEIN (生合成) /  iron (鉄) /
iron (鉄) /  sulfur (硫黄) /
sulfur (硫黄) /  reaction intermediate (反応中間体)
reaction intermediate (反応中間体) ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




