+検索条件
-Structure paper
| タイトル | Structure of deformed wing virus, a major honey bee pathogen. |
|---|---|
| ジャーナル・号・ページ | Proc Natl Acad Sci U S A, Vol. 114, Issue 12, Page 3210-3215, Year 2017 |
| 掲載日 | 2017年3月21日 |
 著者 著者 | Karel Škubník / Jiří Nováček / Tibor Füzik / Antonín Přidal / Robert J Paxton / Pavel Plevka /   |
| PubMed 要旨 | The worldwide population of western honey bees () is under pressure from habitat loss, environmental stress, and pathogens, particularly viruses that cause lethal epidemics. Deformed wing virus (DWV) ...The worldwide population of western honey bees () is under pressure from habitat loss, environmental stress, and pathogens, particularly viruses that cause lethal epidemics. Deformed wing virus (DWV) from the family , together with its vector, the mite , is likely the major threat to the world's honey bees. However, lack of knowledge of the atomic structures of iflaviruses has hindered the development of effective treatments against them. Here, we present the virion structures of DWV determined to a resolution of 3.1 Å using cryo-electron microscopy and 3.8 Å by X-ray crystallography. The C-terminal extension of capsid protein VP3 folds into a globular protruding (P) domain, exposed on the virion surface. The P domain contains an Asp-His-Ser catalytic triad that is, together with five residues that are spatially close, conserved among iflaviruses. These residues may participate in receptor binding or provide the protease, lipase, or esterase activity required for entry of the virus into a host cell. Furthermore, nucleotides of the DWV RNA genome interact with VP3 subunits. The capsid protein residues involved in the RNA binding are conserved among honey bee iflaviruses, suggesting a putative role of the genome in stabilizing the virion or facilitating capsid assembly. Identifying the RNA-binding and putative catalytic sites within the DWV virion structure enables future analyses of how DWV and other iflaviruses infect insect cells and also opens up possibilities for the development of antiviral treatments. |
 リンク リンク |  Proc Natl Acad Sci U S A / Proc Natl Acad Sci U S A /  PubMed:28270616 / PubMed:28270616 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.45 - 3.802 Å |
| 構造データ |  PDB-5g51:  PDB-5g52: |
| 化合物 |  ChemComp-HOH: 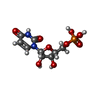 ChemComp-U5P: 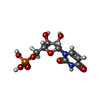 ChemComp-U: |
| 由来 |
|
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  PICORNAVIRALES (ピコルナウイルス目) / PICORNAVIRALES IFLAVIRIDAE IFLAVIRUS DWV CAPSID P-DOMAIN JELLYROLL INHIBITOR ANTIVIRAL CATALYTIC SITE PROTEASE LIPASE ESTERASE RECEPTOR / PICORNAVIRALES (ピコルナウイルス目) / PICORNAVIRALES IFLAVIRIDAE IFLAVIRUS DWV CAPSID P-DOMAIN JELLYROLL INHIBITOR ANTIVIRAL CATALYTIC SITE PROTEASE LIPASE ESTERASE RECEPTOR /  VIRUS (ウイルス) / VIRUS (ウイルス) /  IFLAVIRIDAE / IFLAVIRIDAE /  IFLAVIRUS / DWV CAPSID / P JELLYROLL / CATALYTIC SITE PROTEASE / LIPASE ESTERASE RECEPTO / IFLAVIRUS / DWV CAPSID / P JELLYROLL / CATALYTIC SITE PROTEASE / LIPASE ESTERASE RECEPTO /  Deformed wing virus / bee pathogen Deformed wing virus / bee pathogen |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について















