+検索条件
-Structure paper
| タイトル | The ribosome termination complex remodels release factor RF3 and ejects GDP. |
|---|---|
| ジャーナル・号・ページ | Nat Struct Mol Biol, Year 2024 |
| 掲載日 | 2024年7月19日 |
 著者 著者 | Li Li / Mariia Yu Rybak / Jinzhong Lin / Matthieu G Gagnon /   |
| PubMed 要旨 | Translation termination involves release factors RF1, RF2 and the GTPase RF3 that recycles RF1 and RF2 from the ribosome. RF3 dissociates from the ribosome in the GDP-bound form and must then ...Translation termination involves release factors RF1, RF2 and the GTPase RF3 that recycles RF1 and RF2 from the ribosome. RF3 dissociates from the ribosome in the GDP-bound form and must then exchange GDP for GTP. The 70S ribosome termination complex (70S-TC) accelerates GDP exchange in RF3, suggesting that the 70S-TC can function as the guanine nucleotide exchange factor for RF3. Here, we use cryogenic-electron microscopy to elucidate the mechanism of GDP dissociation from RF3 catalyzed by the Escherichia coli 70S-TC. The non-rotated ribosome bound to RF1 remodels RF3 and induces a peptide flip in the phosphate-binding loop, efficiently ejecting GDP. Binding of GTP allows RF3 to dock at the GTPase center, promoting the dissociation of RF1 from the ribosome. The structures recapitulate the functional cycle of RF3 on the ribosome and uncover the mechanism by which the 70S-TC allosterically dismantles the phosphate-binding groove in RF3, a previously overlooked function of the ribosome. |
 リンク リンク |  Nat Struct Mol Biol / Nat Struct Mol Biol /  PubMed:39030416 PubMed:39030416 |
| 手法 | EM (単粒子) |
| 解像度 | 2.9 - 3.6 Å |
| 構造データ | EMDB-29620, PDB-8fzd: EMDB-29621, PDB-8fze:  EMDB-29622: Cryo-EM structure of an E. coli rotated ribosome complex bound with RF3-ppGpp and p/E-tRNAPhe (State I-C)  EMDB-29623: RF3-ppGpp bound to an E. coli rotated ribosome, from focused classification and refinement (State I-C) EMDB-29624, PDB-8fzf: EMDB-29627, PDB-8fzg: EMDB-29628, PDB-8fzh: EMDB-29631, PDB-8fzi: EMDB-29634, PDB-8fzj: |
| 化合物 |  ChemComp-MG:  ChemComp-ZN:  ChemComp-HOH: 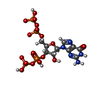 ChemComp-G4P:  ChemComp-GCP: |
| 由来 |
|
 キーワード キーワード | RIBOSOME / release factor 1 / release factor 3 / termination complex / cryo-EM / tRNA / RIBOSOME/RNA / RIBOSOME-RNA complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN文献について
EMN文献について


















 escherichia phage t4 (ファージ)
escherichia phage t4 (ファージ)
